Xenonukleinsäure
Xenonukleinsäuren (englisch Xeno nucleic acid, kurz XNA) sind künstliche Varianten der natürlichen Nukleinsäuren RNA und DNA und damit Teil der Synthetischen Biologie. XNA ist ein synthetisches Polymer, das die gleichen Informationen wie DNA tragen kann, jedoch mit unterschiedlichen molekularen Bestandteilen. Wesentliches Merkmal ist, dass das Zuckermolekül (Ribose bzw. Desoxyribose) durch einen anderen Zucker (beispielsweise Threose oder eine Hexose), ein Zuckeranalogon (wie Ethylenglycol) oder eine andere Gruppe ersetzt ist.[1] Die Entwicklung von sechs verschiedenen dieser künstlichen XNA-Nukleotide wurde im Dezember 2011 von Vitor B. Pinheiro und Kollegen zur Publikation eingereicht. Diese können wie RNA und DNA Nukleinsäurebrückenketten bilden, wodurch sich genetische Informationen speichern und abrufen lassen.[2] Die Vorsilbe „Xeno“ (und damit das „X“ in XNA) leitet sich ab vom griechischen ξένος und bedeutet „fremd“ oder „Fremdkörper“, was sich auf den Unterschied in der Molekülstruktur im Vergleich zu DNA oder RNA bezieht.[3]
Selbst wenn keine Nicht-Standard-Basen vorkommen, d. h. die genetische Information in den vier kanonischen DNA-Basen gespeichert ist, können natürliche DNA-Polymerasen diese Informationen nicht lesen und duplizieren. Somit ist die in XNA gespeicherte genetische Information „unsichtbar“ und daher für natürliche Organismen auf DNA-Basis unbrauchbar.[4]
Forschungsgeschichte
Die Struktur der DNA wurde 1953 entdeckt. Die exotischen DNA-ähnlichen Strukturen von XNA wurden erstmals in den frühen 2000er Jahren geschaffen. In Schwung kam die Forschung an XNA aber erst, als es gelang, ein spezielles Polymerase-Enzym zu entwickeln, das XNA aus einer DNA-Vorlage kopieren und auch wieder XNA in die DNA zurückkopieren kann.[3] Beispielsweise haben Pinheiro et al. 2012 eine solche XNA-fähige Polymerase gefunden und patentiert[5], die mit Sequenzen von ca. 100 bp Länge arbeiten kann.[2] Das Studium der Herstellung und Anwendung von XNA hat so das Feld der Xenobiologie (als Teildisziplin der Synthetischen Biologie) geschaffen. Derzeit wird weiter an der Entwicklung synthetischer Polymerasen zur Transformation von XNA geforscht.
In jüngerer Zeit gelang es Philipp Holliger und Alexander Taylor (beide University of Cambridge), sogenannte XNAzyme herzustellen. Das sind XNA-Äquivalente zu den natürlichen Ribozymen und künstlichen Desoxyribozymen, die wie Enzyme als Bio-Katalysatoren wirken. Dies zeigt, dass XNAs nicht nur erbliche Informationen speichern, sondern auch katalytisch wirken können, was die Möglichkeit erhöht, dass das Leben einst mit anderen Nukleinsäuren als RNA oder DNA begonnen haben könnte.[6]
Aufbau
DNA- und RNA-Stränge werden gebildet, indem sogenannte Nukleotide als Bausteine zu lange Molekülketten aneinandergereiht werden. Ein Nukleotid besteht aus drei chemischen Bestandteilen:
- einem Phosphat
- einer Zuckergruppe mit fünf Kohlenstoff-Atomen (Pentose): Ribose bei RNA bzw. ihr Abkömmling Desoxyribose bei DNA
- und eine von fünf Standardbasen: Adenin (A), Guanin (G), Cytosin (C), sowie Uracil (U) bei RNA bzw. Thymin (T) bei DNA
Die Moleküle, die sich zu den sechs Xeno-Nukleinsäuren von Pinheiro et al. (Dezember 2011) zusammenfügen, sind nahezu identisch mit denen von DNA und RNA, mit einer Ausnahme: In XNA-Nukleotiden wurden die Desoxyribose- bzw. Ribosezuckergruppen von DNA und RNA durch andere chemische Strukturen ersetzt. Gleich sind meist das Phosphatrückgrat und im Normalfall auch die Basen. Diese Substitutionen machen XNAs funktional und strukturell analog zu DNA und RNA, obwohl sie unnatürlich und künstlich sind.[2] Allerdings können die Basen ebenfalls – wie auch bei RNA oder DNA – modifiziert sein, etwa zu Pyrimidin.
Zu den bisher geschaffenen Arten von synthetischer XNA gehören:[4]
- 1,5-Anhydrohexitol-Nukleinsäure (englisch hexose nucleic acid, HNA)
- Cyclohexen-Nukleinsäure (englisch cyclohexenyl nucleic acid, CeNA)
- Threonukleinsäure, genauer: α-l-Threose-Nukleinsäure (englisch threonucleic acid, threose nucleic acid, TNA)
- Glycolnukleinsäure (englisch glycol nucleic acid, GNA) mit Ethylenglycol
- Peptid-Nukleinsäure (englisch peptide nucleic acid, PNA) mit einem ‚Pseudopeptid‘, kein Phosphat
- 2′-Desoxy-2′-fluoro-arabino-Nukleinsäure (englisch 2′-Deoxy-2′-fluoro-arabinonucleic acid, FANA) und 2′-Desoxy-arabino-Nukleinsäure (englisch 2′-Deoxy-arabinonucleic acid, ANA),[7][8] siehe Arabinosylnukleoside.
- L-aTNA, D-aTNA und SNA (englisch acyclic L/D-threoninol nucleic acid, serinol nucleic acid)[9][10] mit den stickstoffhaltigen Alkoholen Threoninol bzw. Serinol.[11][10]
Ein Spezialfall mit zusätzlichen Bindungen an der Zuckergruppe sind
- verbrückte Nukleinsäuren (englisch locked nucleic acid, inaccessible RNA, LNA) im Gegensatz zu unverbrückten Nukleinsäuren (englisch unlocked nucleic acid, UNA)[12][13]
| Nukleinsäure-Bausteine von Xenonukleinsäuren vs. RNA/DNA | |
|---|---|
.png.webp) |
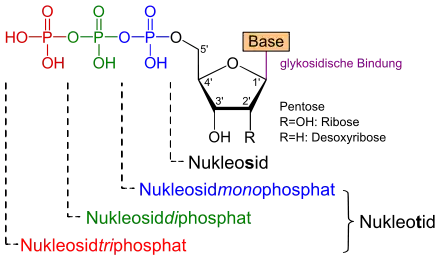 |
| Grundaufbau eines künstlichen XNA-Nukleotids – hier mit einer Hexose als Zucker |
Grundaufbau eines natürlichen Nukleotids – mit Ribose (RNA) bzw. Desoxyribose (DNA) als Zucker |
 |
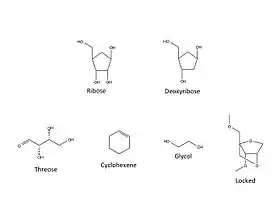 |
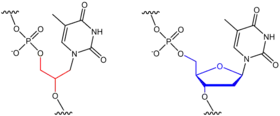
| Die Glycolnukleinsäure (GNA) (links) ist ein Beispiel für eine Xenonukleinsäure, da sie ein anderes Rückgrat (englisch backbone) als die DNA (rechts) hat | Unterschiedliche in XNAs verwendete Zuckersubstituenten im Vergleich zu herkömmlicher, biologischer DNA und RNA |
 |
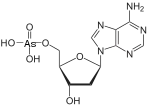 |
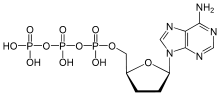
| 2',3'-Ddidesoxyadenosinetriphosphate (ddATP) als Beispiel eines Didesoxynukleotids, die für die DNA-Sequenzierung nach Sanger verwendet werden | Desoxyadenosinmonoarsenat (dAMAs) als Beispiel für eine substituierte Phosphatgruppe (ab 2010 diskutiert als natürlich vorkommend in GFAJ-1-Bakterien) |
Eigenschaften und potentielle Anwendungen
XNA kann mit natürlicher Nukleinsäure wechselwirken, ist aber wesentlich stabiler gegen Nukleinsäureabbaumechanismen, da es keine natürlichen Enzyme gibt, die für den Abbau von hexosebasierten Nukleotiden geeignet wären. Dadurch ließe sich diese Form von Erbinformationsträger verwenden, um virale oder bakterielle Genome bzw. Genomabschnitte zu markieren.
HNA könnte möglicherweise als Arzneimittel Verwendung finden, da es bestimmte Sequenzen erkennen und binden kann. Wissenschaftler konnten HNAs für die Bindung von Sequenzen isolieren, die auf HIV abzielen.[14]
Cyclohexen-Nukleinsäuren (CeNAs) mit einer Stereochemie, die der D-Form ähnelt, können mit sich selbst und mit RNA stabile Duplexe bilden, wohingegen die von CeNAs mit DNA gebildeten Duplexe weniger stabil sind.[15]
XNA kann ebenfalls als Katalysator eingesetzt werden, ähnlich wie RNA als Enzym (Ribozym) wirken kann. Es hat sich gezeigt, dass XNA sowohl DNA-, RNA- als auch andere XNA-Sequenzen spalten und ligieren kann, wobei die meisten Aktivitäten XNA-katalysierte Reaktionen an XNA-Molekülen selbst sind. Mit Hilfe dieser Forschung könnte entschieden werden, ob die Rollen von DNA und RNA im Leben durch natürliche Selektionsprozesse entstanden sind oder ob es sich um ein eher zufälliges Ereignis handelt.[16]
Siehe auch
- Xenobiologie
- DNA: Nicht-Standard-Basen
- Didesoxyribonukleosidtriphosphate (ddNTPs): Artifizielle Zwischenstufen bei der DNA-Sequenzierung nach Sanger.
- Desoxyadenosinmonoarsenat (dAMAs) siehe GFAJ-1 §Diskussion um den Einbau von Arsen in Biomoleküle (fraglicher Einbau in DNA bei Halomonas-Spezies GFAJ-1)
Weblinks
- Nina Weber: Forscher erschaffen künstliche DNA-Alternativen. Spiegel online, 19. April 2012.
- Synthetische Biologie – Was ist das?
Einzelnachweise
- Markus Schmidt: Synthetic Biology. John Wiley & Sons, 9. Mai 2012, ISBN 978-3-527-65926-5, S. 151– (Abgerufen am 6. März 2019).
- Vitor B. Pinheiro et al.: Synthetic Genetic Polymers Capable of Heredity and Evolution. In: Science. 336, Nr. 6079, 2012, S. 341–344. bibcode:2012Sci...336..341P. doi:10.1126/science.1217622. PMID 22517858. PMC 3362463 (freier Volltext).
- Robbie Gonzales: XNA Is Synthetic DNA That's Stronger than the Real Thing. In: Io9. 19. April 2012. Abgerufen am 7. März 2019.
- Markus Schmidt: Xenobiology: A new form of life as the ultimate biosafety tool. In: BioEssays. 32, Nr. 4, April 2010, S. 322–331. doi:10.1002/bies.200900147. PMID 20217844. PMC 2909387 (freier Volltext).
- Patent WO2013156786A1: Polymerase capable of producing non-dna nucleotide polymers. Veröffentlicht am 24. Oktober 2013, Erfinder: Chris Cozens, Philipp Holliger, Vitor Pinheiro.
- World's first artificial enzymes created using synthetic biology. In: Medical Research Council. 1. Dezember 2014.
- Feng Li Sanjay Sarkhel, Christopher J. Wilds et al.: 2′-Fluoroarabino- and Arabinonucleic Acid Show Different Conformations, Resulting in Deviating RNA Affinities and Processing of Their Heteroduplexes with RNA by RNase H, in: Biochemistry, 4. April 2006 Apr, 45(13), S. 4141–4152, doi:10.1021/bi052322r. PMC 2553321 (freier Volltext)
- Mike McCrae: Life's First Genes May Have Contained a Nucleic Acid You've Probably Never Heard Of , auf: ScienceAlert/Nature vom 20. Januar 2020
- Keiji Murayama, Hiromu Kashida, Hiroyuki Asanuma: Acyclic L-threoninol nucleic acid (L-aTNA) with suitable structural rigidity cross-pairs with DNA and RNA, in: Chemical Communications Issue 30, 2015, doi:10.1039/C4CC09244A
- Adele Alagia, Montserrat Terrazas, Ramon Eritja: Modulation of the RNA Interference Activity Using Central Mismatched siRNAs and Acyclic Threoninol Nucleic Acids (aTNA) Units, in: Molecules 2015, 20(5), S. 7602–7619 doi:10.3390/molecules20057602, PDF, Fig. 1
- Externe Identifikatoren von bzw. Datenbank-Links zu L-Threoninol: CAS-Nummer: 3228-51-1, EG-Nummer: 803-664-7, ECHA-InfoCard: 100.230.446, PubChem: 2033049, ChemSpider: 1534111, DrugBank: DB01724, Wikidata: Q27273483.
- Niels Langkjær, Anna Pasternak, Jesper Wengel: UNA (unlocked nucleic acid): A flexible RNA mimic that allows engineering of nucleic acid duplex stability. In: Bioorganic & Medicinal Chemistry. Band 17, Nr. 15, 2009, S. 5420–5425, doi:10.1016/j.bmc.2009.06.045.
- M. A. Campbell, J. Wengel: Locked vs. unlocked nucleic acids (LNA vs. UNA): contrasting structures work towards common therapeutic goals. In: Chemical Society reviews. Band 40, Nr. 12, Dezember 2011, S. 5680–5689, doi:10.1039/c1cs15048k, PMID 21556437 (Review).
- Andy Extance: Polymers perform non-DNA evolution. In: Royal Society of Chemistry. 19. April 2012. Abgerufen am 6. März 2019.
- Ping Gu, Guy Schepers, Jef Rozenski, Arthur Van Aerschot, Piet Herdewijn: Base Pairing Properties of D- and L-Cyclohexene Nucleic Acids (CeNA). In: Oligonucleotides. 13, Nr. 6, 2003, S. 479–489. doi:10.1089/154545703322860799. PMID 15025914.
- Alexander I. Taylor, Vitor B. Pinheiro, Matthew J. Smola, Alexey S. Morgunov, Sew Peak-Chew, Christopher Cozens, Kevin M. Weeks, Piet Herdewijn, Philipp Holliger: Catalysts from synthetic genetic polymers. In: Nature. 518, Nr. 7539, 2015, S. 427–430. bibcode:2015Natur.518..427T. doi:10.1038/nature13982. PMID 25470036. PMC 4336857 (freier Volltext).