Okazaki-Fragment
Okazaki-Fragment heißt in der Molekularbiologie einer der während der DNA-Replikation entstehenden kurzen Abschnitte des Folgestrangs aus DNA.[1] Bei Prokaryoten ist ein solches Fragment 1000 bis 2000 Nukleotide lang, bei Eukaryoten 100 bis 200.[2] Benannt ist es nach der japanischen Wissenschaftlerin Tsuneko Okazaki und ihrem Mann Reiji Okazaki, die 1968[3] das Modell eines Replikationsmechanismus mit diskontinuierlicher DNA-Synthese vorschlugen.[4][5]
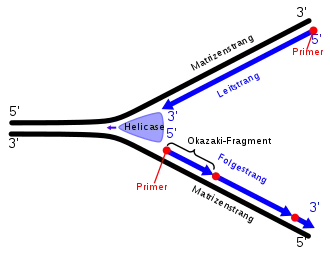
Im Prozess der Replikation wird ein doppelsträngiges DNA-Molekül verdoppelt, sodass zwei gleiche doppelsträngige DNA-Moleküle entstehen. Für den Beginn der Replikation muss die vorliegende DNA-Doppelhelix zuerst an bestimmten Stellen durch eine Helikase-Aktivität entwunden und die Bindung der Wasserstoffbrücken zwischen den Basenpaaren der beiden antiparallel verlaufenden Strängen gelöst werden.
An der geöffneten Stelle, dem Replikationsursprung oder Origin, sind die DNA-Stränge somit voneinander getrennt als zwei einzelsträngige Abschnitte. Diese werden nun jeweils als Matrize für den Aufbau eines durch Basenpaarung bestimmten komplementären Stranges benutzt. Jeder der beiden alten Stränge wird also durch einen neu gebildeten Strang zu einem Doppelstrang ergänzt. Die so semikonservativ gebildeten zwei DNA-Doppelstränge haben – abgesehen von selten auftretenden Fehlern – die gleiche Basensequenz, da auch die originalen Stränge komplementär zueinander waren. Damit liegt die genetische Information dann in zwei Kopien vor.
Vom Replikationsursprung ausgehend schreitet die Auftrennung des Doppelstrangs in zwei einzelsträngige Bereiche als Y-förmige Aufgabelung während der Replikation längs der DNA fort in eine Richtung und, bidirektional, auch in die andere entgegengesetzte. Betrachtet man den in einer Replikationsgabel ablaufenden Prozess der Neusynthese komplementärer DNA-Stränge genauer, wird ein Unterschied deutlich: Vom Origin aus kann das Enzym DNA-Polymerase an dem einen Matrizenstrang kontinuierlich fortlaufend den komplementären Strang aufbauen, an dem anderen Matrizenstrang aber nicht – hier werden diskontinuierlich nacheinander komplementäre DNA-Teilstränge synthetisiert, in sogenannten Okazaki-Fragmenten, und später miteinander verbunden.
Denn die von DNA-Polymerasen katalysierte Polymerisation erfolgt schrittweise jeweils durch Anfügen eines aktivierten Nukleotids (dNTP) an das 3′-Ende des bereits synthetisierten Nukleinsäurestranges, und ist so ausschließlich in 5′→3′ Richtung möglich. Die dabei für den komplementären Aufbau notwendige Matrize ist antiparallel orientiert, also in Richtung 3′→5′. Da auch die beiden aufgetrennten Matrizenstränge antiparallel zueinander orientiert sind, kann eine DNA-Polymerase nur an dem einen Matrizenstrang in Bewegungsrichtung der wandernden Replikationsgabel fortschreiten und kontinuierlich den sogenannten Leitstrang (englisch leading strand) aufbauen. An dem anderen Matrizenstrang hingegen muss eine DNA-Polymerase in umgekehrter Richtung prozessieren und damit entgegen der Bewegungsrichtung der Replikationsgabel. Daher werden hier am freigelegten Matrizenstrang regelmäßig zunächst fragmentarisch Teilabschnitte aufgebaut und diese Okazaki-Fragmente anschließend zum sogenannten Folgestrang (englisch lagging strand) verknüpft. Bei den üblicherweise zwei bidirektional in Gegenrichtung wandernden Replikationsgabeln schließt sich der Folgestrang der einen an den Leitstrang der anderen Replikationsgabel an.
In den kleinen ringförmigen Genomen von Prokaryoten oder Mitochondrien gibt es meist nur einen Replikationsursprung, in großen eukaryotischen Genomen dagegen oft mehrere Tausend, um durch viele gleichzeitig fortschreitende Replikationsgabeln die Geschwindigkeit der Replikation zu erhöhen. Das für die Polymerisation der DNA zuständige Enzym – in Bakterien der Proteinkomplex DNA-Polymerase III (Pol III), in Säugerzellen DNA-Polymerase δ und DNA-Polymerase ε bzw. in Mitochondrien DNA-Polymerase γ – braucht als Starthilfe ein kurzes, Primer genanntes Oligonukleotid. Dieses wird als ein an die DNA-Matrize gebundenes kleines RNA-Molekül von einer Primase bereitgestellt. Die DNA-Polymerase fügt daran dann weitere Nukleotide an, nun aber Bausteine der DNA (dNTP) – bei der fragmentarischen Synthese des Folgestranges solange, bis sie auf das 5′-Ende des zuvor gebildeten kurzen Fragments stößt. Diese diskontinuierlich wiederholt gebildeten kurzen Abschnitte aus DNA heißen Okazaki-Fragmente.
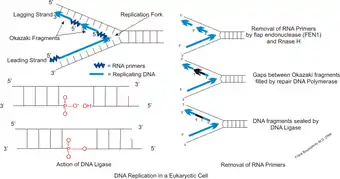
Ein weiteres Enzym mit Nuklease-Aktivität entfernt dann den RNA-Primer, eine Reparaturpolymerase – die prokaryotische DNA-Polymerase I (Pol I) bzw. die eukaryotische DNA-Polymerase α – ersetzt ihn durch DNA und füllt die Lücke zwischen Fragmenten mit komplementären Nukleotiden. Eine DNA-Ligase verknüpft die einzelnen komplettierten Fragmente schließlich durch eine Esterbindung zwischen der Hydroxygruppe des Zuckers Desoxyribose am 3′-Ende des einen und dem Phosphatrest am 5′-Ende des nächsten Teilstrangs zu einem Folgestrang mit bruchlosem Rückgrat. Diese für die Integration der Okazaki-Fragmente nötigen Verfahrensschritte sind denen einer DNA-Reparatur ähnlich.
Literatur
- Alberts, Bray, Johnson, Lewis: Lehrbuch der molekularen Zellbiologie. 2., korrigierte Auflage. Wiley-VCH, Weinheim 2001, ISBN 3-527-30493-2.
- (englische Ausgabe: B. Alberts, A. Johnson, J. Lewis et al.: Molecular Biology of the Cell. 4. Ausgabe. Garland Science, New York 2002, Kapitel: DNA Replication Mechanisms. Online auf dem NCBI-Bookshelf)
Einzelnachweise
- Graw, Jochen.: Genetik. 6., überarbeitete und aktualisierte Auflage. Springer Berlin Heidelberg, Berlin, Heidelberg 2015, ISBN 978-3-662-44816-8.
- D. Voet, J. G. Voet, C. W. Pratt: Lehrbuch der Biochemie. 2. Auflage. Wiley-VCH, Weinheim 2010, S. 963.
- R. Okazaki, T. Okazaki et al.: Mechanism of DNA chain growth. I. Possible discontinuity and unusual secondary structure of newly synthesized chains. In: PNAS. Band 59, Nr. 2, Februar 1968, doi:10.1073/pnas.59.2.598, PMID 4967086, PMC 224714 (freier Volltext), S. 598–605.
- T. Okazaki: Days weaving the lagging strand synthesis of DNA - A personal recollection of the discovery of Okazaki fragments and studies on discontinuous replication mechanism. In: Proceedings of the Japan Academy. Series B, Physical and Biological Sciences. Band 93, Nr. 5, Mai 2017, doi: 10.2183/pjab.93.020, PMID 28496054, PMC 5489436 (freier Volltext), S. 322–338.
- A. Yudelevich, B. Ginsberg, J. Hurwitz: Discontinuous synthesis of DNA during replication. In. PNAS. Band 61, Nr. 3, November 1968, doi:10.1073/pnas.61.3.1129, PMID 4879822, PMC 305445 (freier Volltext), S. 1129–1136.