xDNA
xDNA ist eine künstliche DNA, bei der acht verschiedene Nukleinbasen verwendet werden.
Eigenschaften
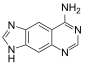
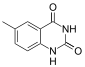
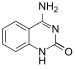
Die DNA-Sequenz von xDNA kann zusätzlich zu den vier natürlichen Nukleinbasen Adenin, Thymin, Cytosin und Guanin auch die vier synthetischen Nukleinbasen xA, xT, xC und xG enthalten.[1] Die synthetischen Basen wurden jeweils um einen Benzolring erweitert, wodurch der Durchmesser der Doppelhelix etwas größer (2,4 Nanometer im Vergleich zu 2 nm bei B-DNA),[2] die beiden Furchen erweitert[3] und die Fluoreszenz verstärkt wird.[4][5] Es wurden auch synthetische Nukleinbasen erzeugt, die um einen Naphthylring erweitert wurden (xxDNA).[6]
 |
 |
 |
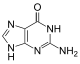 |
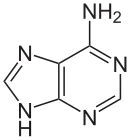
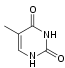
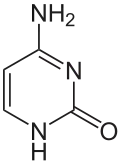
| Adenin | Thymin | Cytosin | Guanin |
 |
 |
 |
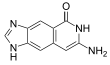 |
| xA | xT | xC | xG |
Die Basenpaarung der synthetischen Nukleinbasen in einer Doppelhelix ist analog zu ihren natürlichen Gegenstücken. Bei der bakteriellen Genexpression werden die synthetischen Nukleinbasen als ihre natürlichen Gegenstücke erkannt, wenn auch mit geringerer Effizienz.[2][7]
Einzelnachweise
- S. R. Lynch, H. Liu, J. Gao, E. T. Kool: Toward a designed, functioning genetic system with expanded-size base pairs: solution structure of the eight-base xDNA double helix. In: Journal of the American Chemical Society. Band 128, Nummer 45, November 2006, S. 14704–14711, doi:10.1021/ja065606n. PMID 17090058. PMC 2519095 (freier Volltext).
- A. T. Krueger, L. W. Peterson, J. Chelliserry, D. J. Kleinbaum, E. T. Kool: Encoding phenotype in bacteria with an alternative genetic set. In: Journal of the American Chemical Society. Band 133, Nummer 45, November 2011, S. 18447–18451, doi:10.1021/ja208025e. PMID 21981660. PMC 3255458 (freier Volltext).
- J. R. Blas, O. Huertas, C. Tabares, B. G. Sumpter, M. Fuentes-Cabrera, M. Orozco, P. Ordejón, F. J. Luque: Structural, dynamical, and electronic transport properties of modified DNA duplexes containing size-expanded nucleobases. In: The journal of physical chemistry. A. Band 115, Nummer 41, Oktober 2011, S. 11344–11354, doi:10.1021/jp205122c. PMID 21888322.
- S. K. Jarchow-Choy, A. T. Krueger, H. Liu, J. Gao, E. T. Kool: Fluorescent xDNA nucleotides as efficient substrates for a template-independent polymerase. In: Nucleic acids research. Band 39, Nummer 4, März 2011, S. 1586–1594, doi:10.1093/nar/gkq853. PMID 20947563. PMC 3045586 (freier Volltext).
- D. Varsano, A. Garbesi, R. Di Felice: Ab initio optical absorption spectra of size-expanded xDNA base assemblies. In: The journal of physical chemistry. B. Band 111, Nummer 50, Dezember 2007, S. 14012–14021, doi:10.1021/jp075711z. PMID 18034470.
- P. Sharma, L. A. Lait, S. D. Wetmore: Exploring the limits of nucleobase expansion: computational design of naphthohomologated (xx-) purines and comparison to the natural and xDNA purines. In: Physical chemistry chemical physics: PCCP. Band 15, Nummer 37, Oktober 2013, S. 15538–15549, doi:10.1039/c3cp52656a. PMID 23942832.
- J. C. Delaney, J. Gao, H. Liu, N. Shrivastav, J. M. Essigmann, E. T. Kool: Efficient replication bypass of size-expanded DNA base pairs in bacterial cells. In: Angewandte Chemie (International ed. in English). Band 48, Nummer 25, 2009, S. 4524–4527, doi:10.1002/anie.200805683. PMID 19444841. PMC 3434874 (freier Volltext).