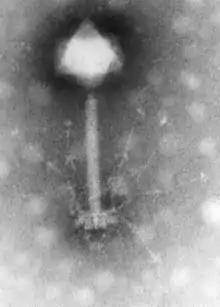
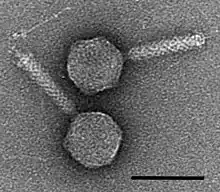
Myoviridae
Die Familie Myoviridae umfasst sechs Gattungen von Viren mit einem Molekül doppelsträngiger DNA(dsDNA) als Genom. Die Familie gehört zur Ordnung der Caudovirales („Schwanzviren“ – Viren mit Kopf-Schwanz-Struktur): Ihre Mitglieder besitzen ein ikosaedrisches Kapsid und ein Schwanzteil; wegen dessen kontraktiler Eigenschaft leitet sich der Familienname von griechisch μυός myos, deutsch ‚Muskel‘ ab. Als Wirte dienen Bakterien (Bakteriophagen), aber teilweise auch Archaeen. Innerhalb der Ordnung Caudovirales zeichnen sich die Myoviridae außerdem durch besonders große, teilweise langgestreckte Kapside (z. B. T4-Phage: 111 × 78 nm) und ein sehr großes Genom (34-169 kBp) aus (siehe Riesenphagen). Der wichtigste Vertreter ist der schon früh entdeckte T4-Phage (Spezies Enterobacteria-Virus T4, Gattung Tequatrovirus), dessen besondere Morphologie und genetische Eigenschaften in der molekularbiologischen Forschung eine herausragende Rolle spielte. Die von diesem Phagen abgeleitete T4-DNA-Ligase findet heute noch Verwendung in der Molekularbiologie. Weitere Mitglieder von Bedeutung sind der Vibrio-Phage KVP40 (Spezies Vibrio-Virus KVP40, Gattung Schizotequatrovirus), der Pseudomonas-Phage phiKZ (Spezies Pseudomonas-Virus phiKZ, Gattung Phikzvirus), der Bakteriophage P1 (Spezies Escherichia-Virus P1, Gattung Punavirus, siehe P1 Artificial Chromosome) u. a.
| Myoviridae | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
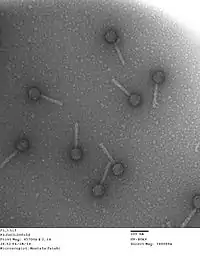
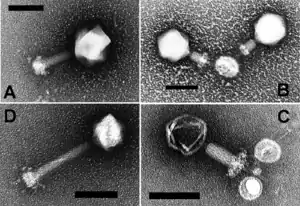
P2-Phagen im TEM | ||||||||||||||
| Systematik | ||||||||||||||
| ||||||||||||||
| Taxonomische Merkmale | ||||||||||||||
| ||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||
| Myoviridae | ||||||||||||||
| Links | ||||||||||||||
|
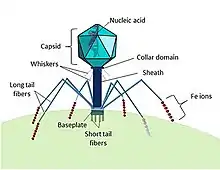
Im März 2021 wurde vorgeschlagen, die Familie mitsamt der Ordnung Caudovirales wegen fehlender Monophylie aufzulösen und durch neu zu schaffende Familien zu ersetzen, damit neue Ergebnisse aus der Metagenomik in die Taxonomie aufgenommen werden können.[3]
Systematik
Die folgende Systematik nach ICTV (Stand 11. Juni 2021)[1] umfasst nur einen Teil der zugehörigen Spezies:
Familie Myoviridae
- Genus Ceceduovirus
- Genus Ishigurovirus
- Genus Firehammervirus (veraltet Cp220virus, Cp220likevirus, Cp220-ähnliche Viren)
- Spezies Campylobacter-Virus CP220
- Genus Fletchervirus (veraltet Cp8virus, Cp8unalikevirus)
- Spezies Campylobacter-Virus CP81
- Genus Firehammervirus (veraltet Cp220virus, Cp220likevirus, Cp220-ähnliche Viren)
- Genus Aphroditevirus
- Genus Tidunavirus
- Genus Felixounavirus (veraltet Felixo1virus, Felixounalikevirus)
- Spezies Salmonella-Virus FelixO1
- Genus Kolesnikvirus (veraltet Ea214virus)
- Genus Mooglevirus
- Spezies Citrobacter-Virus Moogle
- Genus Suspvirus
- Genus Felixounavirus (veraltet Felixo1virus, Felixounalikevirus)
- Genus Aresaunavirus
- Genus Baylorvirus
- Genus Bielevirus
- Genus Canoevirus
- Genus Catalunyavirus
- Genus Citexvirus
- Spezies Pseudomonas-Virus phiCTX (en. Pseudomonas virus phiCTX, mit Bacillus-Phage ΦCTX alias Phage φCTX, wohl zu unterscheiden vom Vibrio-Phage CTXφ, Gattung Affertcholeramvirus, Familie Inoviridae, Ordnung Tubulavirales)[4][5][6]
- Genus Eganvirus
- Genus Entnonagintavirus
- Genus Felsduovirus
- Genus Hpunavirus (veraltet Hp1virus, Hpunalikevirus, Hp1likevirus)
- Spezies Haemophilus-Virus HP1
- Genus Irrigatiovirus
- Genus Irtavirus
- Genus Kisquattuordecimvirus
- Genus Kisquinquevirus
- Genus Longwoodvirus
- Genus Nampongvirus
- Genus Novemvirus
- Genus Peduovirus (veraltet P2virus, P2likevirus, P2-ähnliche Viren, 13 Spezies)
- Spezies Escherichia-Virus P2 (Escherichia virus P2, mit Enterobacteria-Phage P2 alias P2-Phage)[Anmerkung 1][9]
- Genus Phitrevirus
- Genus Playavirus
- Genus Reginaelenavirus
- Genus Reipivirus
- Genus Senquatrovirus
- Genus Seongnamvirus
- Genus Simpcentumvirus
- Genus Stockinghallvirus
- Genus Tigrvirus
- Genus Vimunumvirus
- Genus Vulnificusvirus
- Genus Xuanwuvirus
- Genus Yongunavirus
- Genus Dhakavirus (veraltet Js98virus)
- Genus Gaprivervirus (veraltet Sp18virus)
- Genus Gelderlandvirus (veraltet S16virus)
- Genus Jiaodavirus (veraltet Jd18virus)
- Genus Karamvirus (veraltet Cc31virus)
- Genus Krischvirus (veraltet Rb49virus)
- Genus Moonvirus
- Genus Mosigvirus (veraltet Rb69virus)
- Genus Pseudotevenvirus
- Genus Schizotequatrovirus (veraltet Schizot4virus)
- Genus Slopekvirus (veraltet Kp15virus, nicht zu verwechseln mit der Unterfamilie Slopekvirinae in Familie Autographiviridae)
- Genus Tequatrovirus (veraltet T4virus, T4likevirus, T4-ähnliche Viren)
- Genus Acajnonavirus
- Genus Hadassahvirus
- Genus Lasallevirus
- Genus Lazarusvirus
- Genus Zedzedvirus
- Genus Avunavirus
- Genus Certrevirus (veraltet Cr3virus)
- Genus Henunavirus
- Genus Mydovirus
- Genus Seunavirus (veraltet Se1virus)
- Spezies Salmonella-Virus PVPSE1 (mit Salmonella-Phage PVP-SE1)
- Genus Vequintavirus (veraltet V5virus, Rv5likevirus, rV5-like viruses)[10]
- Spezies Escherichia virus APECc02
- Spezies Escherichia virus FFH2
- Spezies Escherichia virus FV3 (mit Escherichia-Phage vB_EcoM_FV3)[11]
- Spezies Escherichia virus JES2013
- Spezies Escherichia virus Murica
- Spezies Escherichia virus slur16
- Spezies Escherichia virus V5 (*, mit Escherichia-Phage rV5)[12]
- Spezies Escherichia virus V18
- Genus Abouovirus
- Genus Acionnavirus
- Genus Agricanvirus (veraltet Agrican357virus)
- Genus Ahtivirus
- Genus Alcyoneusvirus
- Genus Alexandravirus
- Genus Anamdongvirus
- Genus Anaposvirus
- Genus Aokuangvirus
- Genus Aphroditevirus
- Genus Asteriusvirus
- Genus Atlauavirus
- Genus Aurunvirus
- Genus Ayohtrevirus
- Genus Baikalvirus
- Genus Bakolyvirus
- Genus Barbavirus
- Genus Bcepfunavirus
- Genus Bcepmuvirus (alias Bcepmulikevirus, Bcepmu-ähnliche Viren)
- Spezies Burkholderia-Virus BcepMu (en. Burkholderia virus BcepMu, Typus)
- Genus Becedseptimavirus
- Genus Bellamyvirus
- Genus Bendigovirus
- Genus Biquartavirus
- Genus Bixzunavirus (veraltet Bxzunalikevirus, Bxz1virus, I3likevirus, I3-like viruses, I3-ähnliche Viren)
- Spezies Mycobacterium-Virus Bxz1 (en. Mycobacterium virus Bxz1, mit Mycobacterium-Phage Bxz1) (*)
- Mycobacterium phage Cali
- Mycobacterium phage Catera
- Mycobacterium phage Rizal
- Mycobacterium phage ScottMcG
- Mycobacterium phage Spud
- Spezies Mycobacterium-Virus I3 (en. Mycobacterium virus I3, mit Mycobacterium-Phage I3)
- Spezies „Mycobacterium-Phage Myrna“ (vorgeschlagen)
- Spezies Mycobacterium-Virus Bxz1 (en. Mycobacterium virus Bxz1, mit Mycobacterium-Phage Bxz1) (*)
- Genus Borockvirus
- Genus Brigitvirus
- Genus Brizovirus
- Genus Brunovirus
- Genus Busanvirus
- Genus Carpasinavirus
- Genus Chakrabartyvirus
- Genus Charybdisvirus
- Genus Chiangmaivirus (veraltet Rsl2virus)
- Genus Colneyvirus
- Genus Cymopoleiavirus
- Genus Derbicusvirus
- Genus Dibbivirus
- Genus Donellivirus
- Spezies Bacillus virus G
- Genus Elmenteitavirus
- Genus Elvirus (veraltet Ellikevirus)
- Genus Emdodecavirus (veraltet M12virus)
- Genus Eneladusvirus
- Genus Eponavirus
- Genus Erskinevirus (veraltet Eah2virus)
- Genus Eurybiavirus
- Genus Ficleduovirus
- Genus Flaumdravirus
- Genus Fukuivirus
- Spezies Microcystis virus LMM01 (veraltet Microcystis virus Ma-LMM01)
- Genus Gofduovirus
- Genus Goslarvirus
- Genus Haloferacalesvirus (veraltet Hfunalikevirus)
- Spezies Haloferax virus HF1 (veraltet Halovirus HF1, *)
- Spezies Halorubrum Tailed Virus 5
- Spezies Halorubrum Tailed Virus 7
- Spezies Halorubrum Tailed Virus 8
- Spezies Halorubrum virus HF2 (veraltet Halovirus HF2)
- Genus Hapunavirus (veraltet Hapunalikevirus, Hap1likevirus)
- Spezies Halomonas-Virus HAP1
- Genus Heilongjiangvirus
- Genus Iapetusvirus
- Genus Iodovirus
- Spezies Iodobacter-Virus PLPE (mit Iodobacter-Phage phiPLPE) (*)
- Genus Ionavirus
- Genus Jedunavirus
- Genus Jilinvirus (veraltet Cvm10virus)
- Genus Jimmervirus
- Genus Kanagawavirus
- Genus Kanaloavirus
- Genus Klausavirus
- Genus Kleczkowskavirus (veraltet Rheph4virus)
- Genus Kungbxnavirus
- Genus Kylevirus
- Genus Lagaffevirus
- Genus Leucotheavirus
- Genus Libanvirus
- Genus Llyrvirus
- Genus Loughboroughvirus
- Genus Lubbockvirus (veraltet Cd119virus, Phicd119likevirus)
- Spezies Clostridium-Virus phiCD119
- Spezies Clostridium virus phiCD27 (ΦCD27)[15]
- Genus Machinavirus
- Spezies Erwinia-Virus Machina
- Spezies „Salmonella phage pSal-SNUABM-04“[17][16][Anmerkung 2]
- Genus Marthavirus
- Genus Mazuvirus
- Genus Menderavirus
- Genus Metrivirus
- Genus Mieseafarmvirus (veraltet Rslunavirus)
- Genus Mimasvirus
- Genus Moabitevirus
- Genus Moturavirus
- Genus Muldoonvirus
- Genus Mushuvirus
- Genus Muvirus (veraltet Mulikevirus, Mu-like viruses, Mu-like phages, Mu-ähnliche Viren, 2 Spezies)
- Spezies Escherichia-Virus Mu (mit Enterobacteria-Phage Mu, alias Bacteriophage Mu oder Myovirus Mu)[18]
- Genus Myohalovirus (veraltet Phihlikevirus, phiH-like viruses, PhiH-ähnliche Viren)
- Spezies Halobacterium-Virus phiH (mit Halobacterium-Phage PhiH, Phage ϕH – Akronym ΦH, Fehlschreibung: Phage øH)
- Spezies „Halobacterium-Phage Hs1“ (vorläufig)
- Genus Myosmarvirus
- Genus Naesvirus (veraltet Vidavervirus, Bcep78virus)
- Spezies Burkholderia virus Bcep1
- Spezies Burkholderia virus Bcep43
- Spezies Burkholderia virus Bcep781 (*, ehemals Genus Vidavervirus; veraltet Bcep78virus, Bcep78likevirus, Bcep78-ähnliche Viren)
- Spezies Xanthomonas virus OP2
- Genus Namakavirus
- Genus Machinavirus
- Genus Nankokuvirus (veraltet Kpp10virus)
- Species Pseudomonas-Virus PAKP3 (mit Pseudomonas-Phage PAK_P3)
- Genus Neptunevirus
- Spezies Synechococcus-Virus SRIM8 (en. Synechococcus virus SRIM8)
- Spezies Synechococcus-Virus SRIM50 (en. Synechococcus virus SRIM50)
- Genus Nereusvirus
- Spezies Synechococcus-Virus ACG2014bSyn7803C61 (en. Synechococcus virus ACG2014bSyn7803C61)
- Spezies Synechococcus-Virus ACG2014bSyn9311C4 (en. Synechococcus virus ACG2014bSyn9311C4)
- Genus Nerrivikvirus
- Spezies Synechococcus-Virus SRIM2 (en. Synechococcus virus SRIM2)
- Genus Nankokuvirus (veraltet Kpp10virus)
- Genus Pakpunavirus
- Spezies Pseudomonas-Virus PAKP1 (en. Pseudomonas virus PAKP1, Typus)
- Spezies Pseudomonas-Virus PAKP2 (en. Pseudomonas virus PAKP2)
- Spezies Pseudomonas-Virus PAKP4 (en. Pseudomonas virus PAKP4)
- Spezies Pseudomonas-Virus phiMK (en. Pseudomonas virus phiMK)
- Spezies Pseudomonas-Virus Zigelbrucke (en. Pseudomonas virus Zigelbrucke)
- Spezies „Pseudomonas-Phage K5“ (en. „Pseudomonas phage K5“)
- Spezies „Pseudomonas-Phage K8“ (en. „Pseudomonas phage K8“)[22][21]
- Genus Palaemonvirus
- Genus Pbunavirus (veraltet Pbunalikevirus, PB1likevirus)
- Spezies Pseudomonas-Virus PB1 (mit Pseudomonas-Phage PB1, Typus)
- Genus Peatvirus
- Genus Pemunavirus
- Genus Petsuvirus
- Genus Phabquatrovirus
- Genus Phapecoctavirus
- Genus Phikzvirus (veraltet Phikzlikevirus, PhiKZ-like viruses, PhiKZ-ähnliche Viren)
- Genus Pippivirus
- Genus Plaisancevirus
- Genus Plateaulakevirus
- Genus Polybotosvirus
- Genus Pontusvirus
- Genus Popoffvirus (veraltet Plpelikevirus, Dwarf myoviruses)
- Spezies Aeromonas-Virus 56 (mit Aeromonas-Phage 56)
- Genus Punavirus (veraltet P1virus, Punalikevirus, P1likevirus, P1-ähnliche Viren, 2 Spezies)
- Spezies Escherichia-Virus P1 (Typus)
- Enterobacteria-Phage P1 alias P1-Phage (Typusstamm) — siehe P1 Artificial Chromosome (PAC)[26]
- Spezies Aeromonas-Virus 43 (mit Aeromonas-Phage 43)
- Spezies Escherichia-Virus P1 (Typus)
- Genus Qingdaovirus
- Genus Radnorvirus (veraltet Arv1virus)
- Genus Ripduovirus
- Genus Risingsunvirus
- Genus Ronodorvirus
- Genus Pakpunavirus
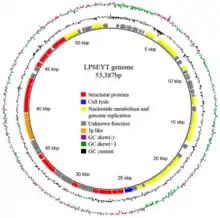
- Genus Rosemountvirus (veralteter Vorschlag LPSEYTvirus)[27][28]
- Spezies Salmonella-Virus birk (en. Salmonella virus birk)
- Spezies Salmonella-Virus BP63 (en. Salmonella virus BP63)
- Spezies Salmonella-Virus SE13 (en. Salmonella virus SE13)
- Spezies Salmonella-Virus UPFBP2 (en. Salmonella virus UPFBP2)
- Spezies Salmonella-Virus yarpen (en. Salmonella virus yarpen)
- Spezies „Salmonella-Phage LPSEYT“ (en. „Salmonella phage LPSEYT“)[27]
- Genus Saclayvirus
- Genus Saintgironsvirus
- Genus Salacisavirus
- Genus Rosemountvirus (veralteter Vorschlag LPSEYTvirus)[27][28]
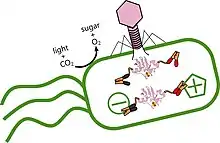
- Spezies Prochlorococcus-Virus PSSM2[32][33][34]
- Prochlorococcus-Phage P-SSM2 alias Cyanophage P-SSM2[20]
- Phage P-SSM2 Fd (Fd wie Ferredoxin, infiziert Prochlorococcus marinus)[35][36]
- Spezies Prochlorococcus-Virus PSSM2[32][33][34]
- Genus Salmondvirus
- Genus Sarumanvirus
- Genus Sasquatchvirus
- Genus Schmittlotzvirus
- Genus Seoulvirus (veraltet Spn3virus)
.png.webp)
-
- Genus Shandongvirus
- Genus Sherbrookevirus
- Genus Shirahamavirus
- Genus Svunavirus
- Genus Tabernariusvirus
- Genus Takahashivirus
- Genus Tamkungvirus
- Genus Taranisvirus
- Genus Tefnutvirus
- Genus Tegunavirus (veraltet Tg1virus)
- Genus Thaumasvirus
- Genus Thetisvirus
- Genus Thornevirus
- Genus Tijeunavirus
- Genus Toutatisvirus
- Genus Tulanevirus (veraltet Secunda5virus)
- Genus Vellamovirus
- Genus Vhmlvirus
- Genus Vibakivirus
- Genus Wellingtonvirus
- Genus Wifcevirus
- Genus Winklervirus
- Genus Yokohamavirus (veraltet Msw3virus)
- Genus Yoloswagvirus
- Genus Yongloolinvirus
- Genus „Cbasmlikevirus“
- Spezies „Cellulophaga-Phage phiSM“ (*)
- Spezies „Cellulophaga-Phage phi3:1“
- Spezies „Cellulophaga-Phage phi3ST:2“
- Spezies „Cellulophaga-Phage phi38:2“
- Spezies „Cellulophaga-Phage phi47:1“
- Genus „Cyanomyovirus“ (informell, nicht zugeordnete Cyanophagen mit Myovirus-Morphologie, zu Autographiviridae?)
- Spezies „Synechococcus-Phage Syn9“ (alias „Bacteriophage Syn9“, „Cyanophage Syn9“)[46][47]
- Spezies „Synechococcus-Phage S-RSM2“ (alias „Bacteriophage S-RSM2“)[48][20]
- Spezies „Synechococcus-Phage S-BM4“ (alias „Cyanophage S-BM4“, „Bacteriophage S-BM4“)[49][20]
- Spezies „Synechococcus-Phage S-WHM1“ (alias „Cyanophage S-WHM1“)[50][20]
- Spezies „Synechococcus-Phage S-RSM88“ (alias „Cyanophage S-RSM88“, „Bacteriophage S-RSM88“)[51][20]
- Spezies „Prochlorococcus-Phage P-SSM4“ (alias „Cyanophage P-SSM4“)[52][20]
- Genus „Sfv and relatives“ (informell, zu unterscheiden von Spezies Rabbit fibroma virus alias Shope fibroma virus,[53] Gattung Leporipoxvirus, Poxviridae)
- Spezies „Enterobacteria-Phage SfV“ (alias „Shigella-Phage SfV“, „Shigella flexneri bacteriophage V“)[54] (*)
- ohne vorgeschlagene Gattung:
- Genus „Cbasmlikevirus“
- Spezies „Alteromonas phage AltPT11-V22“ (alias „Alteromonas-Myovirus V22“, vorgeschlagen)[58]
- Spezies „Bdellovibrio-Phage phi1402“ (englisch „Bdellovibrio phage phi1402“)
- Spezies „Bdellovibrio-Phage phi1422“ (englisch „Bdellovibrio phage phi1422“)
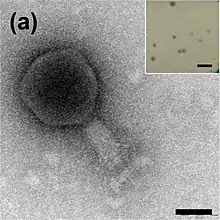
- Spezies „Ochrobactrum-Phage vB_OspM_OC“ (englisch „Ochrobactrum phage vB_OspM_OC“)[59][60]
- Spezies „Pectobacterium-Phage ZF40“ (englisch „Pectobacterium phage ZF40“)
- Spezies „Pseudomonas-Phage OBP“ (alias Pseudomonas fluorescens bacteriophage OBP, vorgeschlagen)[14]
- Spezies „Shigella-Phage A1-1“ (en. „Shigella phage A1-1“)[61]
- Spezies „Sphingomonas-Phage PAU“[62][63]
- Spezies „Vibrio-Phage 138“
- Spezies „Vibrio-Phage CP-T1“
- Spezies „Yersinia-Phage PY100“
- Spezies „Enterobacteria-Phage phi92“ (englisch „Enterobacteria phage phi92“)[64][10]
- „Kabirphage“
- „Mahaphage“ mit der Untergruppe
- „Lak-Phagen“ mit den Vertretern Lak-A1, Lak-A2, Lak-B1 bis Lak-B9 und Lak-C1[67]
- „Biggiephage“
- „Dakhmphage“
- „Kyodaiphage“
- „Kaempephage“
- „Jabbarphage“
- „Enormephage“
- „Judaphage“
- „Whopperphage“
Verschiebungen:
- Genus Viunavirus (veraltet Vi1virus, Viunalikevirus) wurde komplett in das Genus Kuttervirus (veraltet Cba120virus) der Ackermannviridae eingefügt.
- Genus Siminovitchvirus (veraltet Cp51virus) wurde zur neuen Caudovirales-Familie Herelleviridae, Unterfamilie Spounavirinae verschoben.
- Die Genera Agatevirus, Bequatrovirus (veraltet B4virus), Bastillevirus, Caeruleovirus (veraltet Bc431virus), Nitunavirus (veraltet Nit1virus) mit Spezies Bacillus virus NIT1 und Wphvirus wurden ebenfalls zur neuen Familie Herelleviridae, aber Unterfamilie Bastillevirinae verschoben.
- Die neue Unterfamilie Twortvirinae der Herelleviridae enthält nun die Genera Baoshanvirus, Harbinvirus, Kayvirus, Sciuriunavirus, Sepunavirus (veraltet Sep1virus), Silviavirus, Twortvirus (alias Twortlikevirus, Twort-ähnliche Viren) mit Typusspezies Staphylococcus virus Twort
Anmerkungen
- P2 ist ein temperenter Phage. Der „Escherichia-Phage P4“ (ein Coliphage ebenfalls in der Ordnung Caudovirales, aber bisher nicht näher klassifiziert) ist ein Satellitenvirus (d. h. Satellitenphage), das den Phagen P2 als Helfervirus benötigt. Siehe Cruz (2013) und NCBI: Phage P4 satellite (no rank)
- Das Material wurde von dieser Quelle kopiert, die unter einer Creative Commons Attribution 4.0 International License verfügbar ist.
Literatur
- A. M. Q. King, M. J. Adams, E. B. Carstens, E. J. Lefkowitz (Hrsg.): Virus Taxonomy. Ninth Report of the International Committee on Taxonomy of Viruses. Amsterdam 2012, ISBN 978-0-12-384684-6, S. 46–62.
- C. M. Fauquet, M. A. Mayo et al.: Eighth Report of the International Committee on Taxonomy of Viruses. London / San Diego 2004.
Weblinks
- Myoviridae: Genera und Spezies. NCBI
- Bildergalerie der Familie Myoviridae. Big Picture Book of Viruses
Einzelnachweise
- ICTV: ICTV Master Species List 2020.v1, New MSL including all taxa updates since the 2019 release, March 2021 (MSL #36)
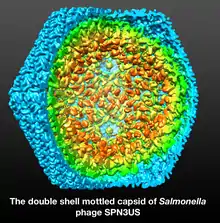
- J. Bernard Heymann et al.: The Mottled Capsid of the Salmonella Giant Phage SPN3US, a Likely Maturation Intermediate with a Novel Internal Shell. In: Viruses, Band 12, Nr. 9, 910, 19. August 2020, doi:10.3390/v12090910.
- Dann Turner, Andrew M. Kropinski, Evelien M. Adriaenssens: A Roadmap for Genome-Based Phage Taxonomy. In: MDPI Viruses, Band 13, Nr. 3, Section Bacterial Viruses, 18. März 2021, 506, doi:10.3390/v13030506
- Pseudomonas virus phiCTX (species). NCBI
- Viral exotoxin. SIB
- Harald Brüssow, Carlos Canchaya, Wolf-Dietrich Hard: Phages and the Evolution of Bacterial Pathogens: from Genomic Rearrangements to Lysogenic Conversion. In: Microbiol Mol Biol Rev., 68(3), September 2004, S. 560–602, doi:10.1128/MMBR.68.3.560-602.2004, PMC 515249 (freier Volltext), PMID 15353570, siehe insbes. Table 1
- Pasteurella virus F108 (species). NCBI
- J. D. Boyce, T. Seemann, B. Adler and M. Harper: Pathogenomics of Pasteurella multocida (PDF) in: Current Topics in Microbiology and Immunology 361, S. 23–38, 9. März 2012, doi:10.1007/82_2012_203
- Renata Filipa Cruz de Matos: Enterococcus faecalis V583 prophages: Dynamic interactions and contribution to bacterial pathogenic traits (PDF; 10 MB) Dissertation, Universidade Nova de Lisboa (UNL), Juli 2013
- L. Truncaite, E. Šimoliūnas, A. Zajančkauskaite, L. Kaliniene, R. Mankevičiūte, J. Staniulis, V. Klausa, R. Meškys: Bacteriophage vB_EcoM_FV3: A new member of "rV5-like viruses". In: Archives of Virology. 157, Nr. 12, 2012, S. 2431–2435. doi:10.1007/s00705-012-1449-x. PMID 22907825.
- Escherichia phage FV3 (species). NCBI
- Escherichia virus V5 (species). NCBI
- Victor Krylov, Maria Bourkaltseva, Elena Pleteneva, Olga Shaburova, Sergey Krylov, Alexander Karaulov, Sergey Zhavoronok, Oxana Svitich, Vitaly Zverev: Phage phiKZ—The First of Giants. In: Viruses. Band 13, Nr. 2. MDPI, 20. Januar 2021, 149, doi:10.3390/v13020149 (englisch).
- Victor Krylov, Olga Shaburova, Sergey Krylov, Elena Pleteneva: A Genetic Approach to the Development of New Therapeutic Phages to Fight Pseudomonas Aeruginosa in Wound Infections. In: MDPI Viruses, Band 5, Nr. 1, 21. Dezember 2012, Special Issue Recent Progress in Bacteriophage Research, S. 15–53, doi:10.3390/v5010015
- Eunsu Ha, Bokyung Son, Sangryeol Ryu: Clostridium perfringens Virulent Bacteriophage CPS2 and Its Thermostable Endolysin LysCPS2. In: MDPI Viruses, Band 10, Nr. 5, Special Issue Biotechnological Applications of Phage and Phage-Derived Proteins, 251, 11. Mai 2018, doi:10.3390/v10050251
- Jun Kwon et al.: Isolation and Characterization of Salmonella Jumbo-Phage pSal-SNUABM-04. In: Viruses, Band 13, Nr. 1, Giant or Jumbo Phages, 27, 25. Dezember 2020, doi:10.3390/v13010027.
- Salmonella phage pSal-SNUABM-04 (species). NCBI
- Mart Krupovic, Anja Spang, Simonetta Gribaldo, Patrick Forterre, Christa Schleper: A thaumarchaeal provirus testifies for an ancient association of tailed viruses with archaea. In: Biochem Soc Trans, Band 39, Nr. 1, Januar 2011, s. 82-88, doi:10.1042/BST0390082, PMID 21265751, ResearchGate
- Synechococcus phage S-PM2 (species). NCBI
- John H. Paul, Matthew B. Sullivan: Marine phage genomics: what have we learned? In: Curr. Op. in Biotechnology, Band 16, Nr. 3, Juni 2005, S. 299–307, doi:10.1016/j.copbio.2005.03.007, PMID 15961031, PMC 15961031 (freier Volltext); siehe Fig. 3 (Genomkarte)
- Xuewei Pan, Xiaoli Cui, Fenjiao Zhang, Yang He, Lingyan Li, Hongjiang Yang: Genetic Evidence for O-Specific Antigen as Receptor of Pseudomonas aeruginosa Phage K8 and Its Genomic Analysis. In: Front. Microbiol., 2. März 2016, doi:10.3389/fmicb.2016.00252
- Pseudomonas phage K8 (species). NCBI
- Pseudomonas phage 201phi2-1, auf: Virus-Host DB, TAX:198110
- Proteomes - Pseudomonas phage 201phi2-1, auf: UniProt
- Pseudomonas phage 201phi2-1 (species). NCBI
- Katherine S. Wetzel, Haley G. Aull, Kira M. Zack, Rebecca A. Garlena, Graham F. Hatfull: Protein-Mediated and RNA-Based Origins of Replication of Extrachromosomal Mycobacterial Prophages. In: mBio, 11(2), März/April 2020, e00385-20, Epub24. März 2020, doi:10.1128/mBio.00385-20, PMC 7157519 (freier Volltext), PMID 32209683
- Ting Yan, Lu Liang, Ping Yin, Yang Zhou, Ashraf Mahdy Sharoba, Qun Lu, Xingxing Dong, Kun Liu, Ian F. Connerton, Jinquan Li: Application of a Novel Phage LPSEYT for Biological Control of Salmonella in Foods. In: MDPI Microorganisms, Band 8, Nr. 3, Section Microbial Biotechnology, 400; doi:10.3390/microorganisms8030400
- Rosemountvirus (genus). NCBI
- Ocean virus hijacks carbon-storing bacteria, auf: EurekAlert vom 26. Mai 2020, Quelle: Rice University
- Ocean virus hijacks carbon-storing bacteria, auf: ScienceDaily vom 26. Mai 2020, Quelle: Rice University
- Ocean virus hijacks carbon-storing bacteria, auf: National Science Foundation (NSF) vom 29. Mai 2020
- Prochlorococcus virus PSSM2 (species). NCBI
- Denise Brehm: Viruses Use Bacteria for Reproduction. SciTechDaily, 26. Januar 2012
- Qinglu Zeng, Sallie W. Chisholm: Marine Viruses Exploit Their Host’s Two-Component Regulatory System in Response to Resource Limitation. In: Curr Biol, Band 22, Nr. 2, CellPress vom 24. Januar 2012, S. 124–128, doi:10.1016/j.cub.2011.11.055, cell.com (PDF) Supplement 1 (PDF; 329 kB)
- Ian J. Campbell, Jose Luis Olmos Jr., Weijun Xu, Dimithree Kahanda, Joshua T. Atkinson, Othneil Noble Sparks, Mitchell D. Miller, George N. Phillips Jr., George N. Bennett, Jonathan J. Silberg: Prochlorococcus phage ferredoxin: Structural characterization and electron transfer to cyanobacterial sulfite reductases – Phage Fd characterization and host SIR interactions. In: J. Biol. Chem., ASBMB Publications, 19. Mai 2020, doi:10.1074/jbc.RA120.013501, jbc.org (PDF)
- Beneath the Ocean’s Surface, a Virus Is Hijacking the Most Abundant Organism on Earth, auf: SciTechDaily vom 6. Juni 2020, Quelle: Rice University
- Matthew Dunne, Mario Hupfeld, Jochen Klumpp, Martin J. Loessner: Molecular Basis of Bacterial Host Interactions by Gram-Positive Targeting Bacteriophages, in: MDPI Viruses, Band 10, Nr. 8, Special Issue Phage-Host Interactions, 397, 28. Juli 2018, doi:10.3390/v10080397. Anm.: B. subtilis phage PBS1 gehört nach ICTV (und NCBI) nicht zu den Siphoviridae sondern hierher. Myoviridae Listeria phage A511 wurde vom ICTV zu den Herelleviridae verschoben.
- ICTV: ICTV Taxonomy history: Bacillus virus PBS1
- NCBI: Bacillus virus PBS1 (species)
- Synechococcus virus S-SM1 (species). NCBI
- S. B. Santos, A. M. Kropinski, P.-J. Ceyssens, H.-W. Ackermann, A. Villegas, R. Lavigne, V. N. Krylov, C. M. Carvalho, E. C. Ferreira, J. Azeredo: Genomic and Proteomic Characterization of the Broad-Host-Range Salmonella Phage PVP-SE1: Creation of a New Phage Genus. In: Journal of Virology. 85, Nr. 21, 2011, S. 11265–11273. doi:10.1128/JVI.01769-10. PMID 21865376. PMC 3194984 (freier Volltext).
- A. Cornelissen, S. C. Hardies, O. V. Shaburova, V. N. Krylov, W. Mattheus, A. M. Kropinski, R. Lavigne: Complete Genome Sequence of the Giant Virus OBP and Comparative Genome Analysis of the Diverse KZ-Related Phages. In: Journal of Virology. 86, Nr. 3, 2011, S. 1844–1852. doi:10.1128/JVI.06330-11. PMID 22130535. PMC 3264338 (freier Volltext).
- C. M. Mizuno, F. Rodriguez-Valera, N. E. Kimes, R. Ghai: Expanding the Marine Virosphere Using Metagenomics. In: PLoS Genetics. 9, Nr. 12, 2013, S. e1003987. doi:10.1371/journal.pgen.1003987. PMID 24348267. PMC 3861242 (freier Volltext).
- K. Holmfeldt, N. Solonenko, M. Shah, K. Corrier, L. Riemann, N. C. Verberkmoes, M. B. Sullivan: Twelve previously unknown phage genera are ubiquitous in global oceans. In: Proceedings of the National Academy of Sciences. 110, Nr. 31, 2013, S. 12798–12803. doi:10.1073/pnas.1305956110. PMID 23858439. PMC 3732932 (freier Volltext).
- unclassified Myoviridae. NCBI
- Synechococcus phage syn9 (species). NCBI
- Cyanophage Syn9 (species). NCBI
- Synechococcus phage S-RSM2 (species). NCBI
- Cyanophage S-BM4 (species). NCBI
- Synechococcus phage S-WHM1 (species). NCBI
- Cyanophage S-RSM88 (species). NCBI
- Prochlorococcus phage P-SSM4 (species). NCBI
- Rabbit fibroma virus (species). NCBI
- Enterobacteria phage SfV (species). NCBI
- Yunfen Hua, Tingting Luo, Yiqi Yang, Dong Dong, Rui Wang, Yanjun Wang, Mengsha Xu, Xiaokui Guo, Fupin Hu, Ping He: Phage Therapy as a Promising New Treatment for Lung Infection Caused by Carbapenem-Resistant Acinetobacter baumannii in Mice. In: Front. Microbiol., 9. Januar 2018; doi:10.3389/fmicb.2017.02659
- Natalia Bagińska, Anna Pichlak, Andrzej Górski1, Ewa Jończyk-Matysiak: Specific and Selective Bacteriophages in the Fight against Multidrug-resistant Acinetobacter baumannii (PDF) in: Virologica Sinica, Band 34, S. 347–357; doi:10.1007/s12250-019-00125-0, Tbl. 1
- Haemophilus phage Aaphi23 (species). NCBI
- Alteromonas phage AltPT11-V22 (species). NCBI
- Ochrobactrum phage vB_OspM_OC (species). NCBI
- Przemyslaw Decewicz, Piotr Golec, Mateusz Szymczak, Monika Radlinska, Lukasz Dziewit: Identification and Characterization of the First Virulent Phages, Including a Novel Jumbo Virus, Infecting Ochrobactrum spp. In: MDPI Int. J. Mol. Sci, Band 21, Nr. 6, Special Issue Bacteriophage Molecular Studies, 18. März 2020, 2096; doi:10.3390/ijms21062096
- Kaitlyn E. Kortright, Rachel E. Done, Benjamin K. Chan, Valeria Souza, Paul E. Turner: Selection for phage resistance reduces virulence of Shigella flexneri, in: ASM Appl. and Env. Microbiol. (AEM), 17. November 2021, doi:10.1128/AEM.01514-21, PDF. Dazu: Erin Garcia de Jesús: A bacteria-virus arms race could lead to a new way to treat shigellosis, auf: ScinceNews vom 14. Dezember 2021.
- Sphingomonas phage PAU (species). NVBI
- Richard Allen White III, Curtis A. Suttle: The Draft Genome Sequence of Sphingomonas paucimobilis Strain HER1398 (Proteobacteria), Host to the Giant PAU Phage, Indicates That It Is a Member of the Genus Sphingobacterium (Bacteroidetes). In: Genome Announc., 1(4), Juli-August 2013, e00598-13, doi:10.1128/genomeA.00598-13, PMID 23929486, PMC 3738902 (freier Volltext)
- Enterobacteria phage phi92 (species). NCBI
- Basem Al-Shayeb, Rohan Sachdeva, L. Chen, Jillian F. Banfield et al.: Clades of huge phages from across Earth’s ecosystems. In: Nature, 12. Februar 2020, doi:10.1038/s41586-020-2007-4
- Ed Yong: A Huge Discovery in the World of Viruses. The Atlantic, 20. Februar 2020
- Audra E. Devoto, Joanne M. Santini et al.: Megaphages infect Prevotella and variants are widespread in gut microbiomes. In: Nature Microbiology, Band 4, S. 693–700, 28. Januar 2019, doi:10.1038/s41564-018-0338-9, insbes. Table 1 und Supplementary Figure 11 (PDF; 9,2 MB)
.png.webp)