Alternatives Spleißen
Das alternative Spleißen (auch differenzielles Spleißen oder gewebespezifisches Spleißen genannt) stellt einen besonderen Vorgang im Rahmen der Transkription bei Eukaryoten dar. Dieser Vorgang wurde erstmals 1977 beobachtet.[1] Auch Viren, die Eukaryoten befallen, nutzen diesen Mechanismus. Aus ein und derselben DNA-Sequenz und dementsprechend ein und derselben prä-mRNA können mehrere verschiedene reife mRNA-Moleküle und durch deren Translation auch mehrere unterschiedliche Polypeptide oder Proteine gebildet werden. Die meisten Gene von Säugetieren unterliegen dem alternativen Splicing.[2] Fehlregulationen hierbei sind häufige Ursache von unterschiedlichen Krankheiten.[3][4][5][6]
Formen des alternativen Spleißens
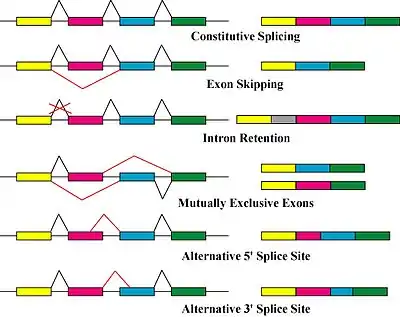
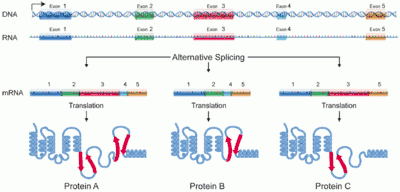
Beim alternativen Splicing entscheidet sich erst während des Spleißvorgangs, welche RNA-Sequenzen Introns und welche Exons sind. Die Regulation erfolgt über Splicefaktoren (Proteine, die Signale auf der RNA erkennen und die Auswahl der splice sites beeinflussen). Dabei können unterschiedliche Formen des alternativen Splicings unterschieden werden:
- Kassettenexons (mutually exclusive exons),
- das Überspringen von Exons (exon skipping) oder
- das Benutzen unterschiedlicher 5' oder 3' Splice Sites (alternative 5'/3' splice site), vgl. nebenstehende Abbildung.
Viele Proteine aus nur einem Gen
Die Entdeckung des alternativen Splicings bedeutet, dass die Ein-Gen-ein-Enzym-Hypothese für Eukaryoten nicht streng gilt. Eine DNA-Sequenz, also ein Gen, kann unterschiedliche Proteine codieren. Auf diese Weise ist z. B. eine menschliche Zelle in der Lage, mit ihren rund 20.000 Genen viele hunderttausend verschiedene Proteine herzustellen – aus relativ wenigen Genen entsteht so ein außerordentlich komplexes Proteom von 500.000 bis 1.000.000 Proteinspezies. Die Informationsdichte der DNA wird also durch Superposition erheblich erhöht.
Demnach kann der Grundsatz „ein Gen codiert für ein Protein“ nicht stimmen, da die Zahl der Proteine die Zahl der Gene weit übersteigt (siehe vorherigen Abschnitt). Weil die Information über die Sekundärstruktur der Proteine in der DNA enthalten sein muss, müssen daher einzelne DNA-Abschnitte für mehrere Proteine „zuständig“ sein, beziehungsweise codieren.
Ein Extrembeispiel hierzu: DSCAM, ein Gen in Drosophila melanogaster, welches das Richtungswachstum von Nervenzellen steuert, hat mehrere Kassettenexons, die miteinander kombiniert werden können, wodurch sich rechnerisch eine Zahl von insgesamt 38.016 verschiedenen Proteinen aus nur diesem einen Gen ergibt. Allerdings wurden nur wenige davon tatsächlich im Organismus nachgewiesen.[7] Im Gegensatz dazu erscheint die Zahl an Genen in diesem Organismus mit ca. 14.000 vergleichsweise klein. Dies unterstreicht eindrucksvoll, dass die Vielzahl an Proteinen in einem Organismus nicht primär durch die Zahl seiner Gene bestimmt ist, sondern vielmehr durch das alternative Splicing der prä-mRNAs.
Auswirkung auf die Genetik
Die Zahl der menschlichen Gene ist nach Sequenzierung des Genoms mit jetzt ca. 20.000 weit hinter den ursprünglichen Annahmen zurückgeblieben. Da jedoch fast jedes zweite Gen alternativ gespleißt werden kann, lässt sich trotz der gering erscheinenden Zahl an Genen eine viel höhere Vielfalt an Proteinen erklären. Daher stellt gerade das Verständnis des alternativen Splicings eine große Herausforderung bei der Erforschung der menschlichen Proteinvielfalt und somit auch dem Verständnis von vielen Erkrankungen (wie z. B. Krebs) und Erbkrankheiten dar.
Spleißen und Evolution
Das alternative Splicing stellt eine evolutionär besonders bedeutende Entwicklung bei den Eukaryoten dar:
- Die Entstehung neuer Proteine kann erheblich leichter erfolgen als bei Prokaryoten, nämlich durch eine veränderte Regulation des Splicings.
- Die Wahrscheinlichkeit, dass ein durch alternatives Splicing neu entstandenes Protein funktionsfähig ist, ist höher als bei einem durch Mutation der codierenden DNA-Sequenz entstandenen neuen Protein. Jedes auf diese Weise im Rahmen der Evolution entstehende Protein enthält zumindest mehrere bereits in anderen Proteinen funktionierende Aminosäure-Sequenzen.
- Dadurch wird die Anpassung von Eukaryoten an veränderte Lebensbedingungen erleichtert und beschleunigt. Dies könnte ein entscheidender Schritt für die Evolution von mehrzelligen Lebewesen mit längerer Generationsdauer gewesen sein. Während bei Bakterien zwischen zwei Generationen oft weniger als eine Stunde vergeht, kann diese Zeit bei Eukaryoten (Beispiel Mensch) auf mehrere Jahrzehnte anwachsen.
Einzelnachweise
- S. M. Berget, C. Moore, P. A. Sharp: Spliced segments at the 5' terminus of adenovirus 2 late mRNA. In: Proceedings of the National Academy of Sciences of the United States of America. Band 74, Nr. 8, 1977, S. 3171–3175, doi:10.1073/pnas.74.8.3171.
- Human Genes: Alternative Splicing Far More Common Than Thought. Abgerufen am 4. Dezember 2020 (englisch).
- O. Stoss u. a.: Misregulation of pre-mRNA splicing that causes human diseases. Concepts and therapeutic strategies. In: Gene Ther Mol Biol. Band 5, 2000, S. 9–30 (researchgate.net [PDF]).
- Y. Barash u. a.: Deciphering the splicing code. In: Nature. Band 465, Nr. 7294, 2010, S. 53–59, PMID 20445623.
- E. S. Lander u. a.: Initial sequencing and analysis of the human genome. In: Nature. Band 15, Nr. 409, 2001, S. 860–921, PMID 11237011.
- N. A. Faustino u. a.: Pre-mRNA splicing and human disease. In: Genes & Dev. Band 17, 2003, S. 419–437 (cshlp.org).
- Campbell-Biologiebuch, englische Ausgabe, S. 362.
Weblinks
- Alternatives Spleißen von Ionenkanälen des Gehirns in Bezug zu mentalen, psychischen und psychomotorischen Krankheiten
- fast-DB – sehr gute Datenbank zum Thema, mit vielen nützlichen Suchwerkzeugen
- TassDB – Datenbank für Tandem-Splice-Sites
- bio-pro.de: Alternatives Spleißen - eine Grundlage der Proteomvielfalt (Memento vom 10. Juli 2010 im Internet Archive), 23. Februar 2006