Phikzvirus
Phikzvirus (veraltet: Phikzlikevirus, PhiKZ-like viruses, PhiKZ-ähnliche Viren) ist eine Gattung von Viren der Familie Myoviridae in der Ordnung Caudovirales, deren natürliche Wirte Bakterien der Gattung Pseudomonas (Pseudomonaden) sind. Sie werden daher (nicht-taxonomisch) als Bakteriophagen klassifiziert. Derzeit (Stand 8. April 2020) sind vom International Committee on Taxonomy of Viruses (ICTV) in dieser Gattung drei Arten (Spezies) anerkannt, darunter die Typusart Pseudomonas-Virus PhiKZ (alias Pseudomonas-Phage PhiKZ, φKZ).[2][3][4]
| Phikzvirus | ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
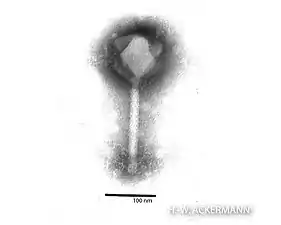
Elektronenmikroskopische Aufnahme eines | ||||||||||||||||
| Systematik | ||||||||||||||||
| ||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||
| ||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||
| Phikzvirus | ||||||||||||||||
| Links | ||||||||||||||||
|
Phagen dieser Gattung werden als große oder ‚Jumbo-Phagen‘ angesehen.[5]
Phagenkern
Von drei Spezies dieser Gattung – neben φKZ die bisher noch nicht vom ICTV bestätigten Spezies φPA3 und 201φ2-1 – ist bekannt, dass sie in der Wirtszelle eine ‚Phagenkern‘ (englisch phage nucleus) genannte Struktur ausbilden (siehe auch Viroplasma). Dieser ähnelt in der Funktion einem eukaryotischen Zellkern, indem er DNA sowie eine Replikations- und Transkriptionsmaschinerie beinhaltet. Die Phagenkern bestehen aus einem Tubulin-ähnlichen Protein PhuZ und einem Schalenprotein (englisch shell protein) gp105. Sie bieten dem Virus eine Immunität gegen die Abwehrkräfte des Wirts (Restriktionsenzyme und das CRISPR-Cas-System).[6][7]
Systematik
Mit Stand 8. April 2020 sind vom ICTV die folgenden Spezies offiziell bestätigt:[3]
Gruppe: dsDNA (Baltimore Gruppe 1)
- Ordnung: Caudovirales
- Familie: Myoviridae
- Gattung: Phikzvirus (veraltet Phikzlikevirus)
- Spezies Pseudomonas-Virus PhiKZ (wissenschaftlich Pseudomonas virus phiKZ, veraltet Pseudomonas phage phiKZ, φKZ oder ΦKZ, Typusspezies)[8][9]
- Pseudomonas-Phage KTN4 (wiss. Pseudomonas phage KTN4)[10]
- Spezies Pseudomonas-Virus PA7 (wiss. Pseudomonas virus PA7, veraltet Pseudomonas phage PA7, PA7)
- Spezies Pseudomonas-Virus SL2 (wiss. Pseudomonas virus SL2, veraltet Pseudomonas phage SL2 , SL2)
- Vom ICTV noch nicht bestätigte Kandidaten sind derzeit laut NCBI:[11]
- Spezies „Pseudomonas phage 201phi2-1“ (alias „Pseudomonas chlororaphis phage 201phi2-1“, 201φ2-1)[12][13][14][7][9]
- Spezies „Pseudomonas phage PA02“ (PA02)[15]
- Spezies „Pseudomonas phage PA1C“ (PA1C)[16]
- Spezies „Pseudomonas phage PaSz-1_45_270k“[17]
- Spezies „Pseudomonas phage PaZh_1“ (PaZh_1)[18]
- Spezies „Pseudomonas phage Phabio“[19]
- Spezies „Pseudomonas phage PhiPA3“ (φPA3)[20][8][9]
- Spezies „Pseudomonas phage Psa21“ (Psa21)[21]
- Spezies „Pseudomonas phage vB_PaeM_PS119XW“[22]
Die früher hier geführte Spezies Pseudomonas-Virus EL (wiss. Pseudomonas virus EL, veraltet Pseudomonas phage EL) wurde innerhalb der Myoviridae in die neue Gattung Elvirus verschoben, da die genetische Übereinstimmung mit der Typusspezies φKZ zu gering ist.[23]
Die früher ebenfalls hier geführte Spezies Pseudomonas phage Lin68 wurde mangels verfügbarer Genomdaten gelöscht, da eine Bestimmung der genetischen Verwandtschaft nicht möglich ist.[23]
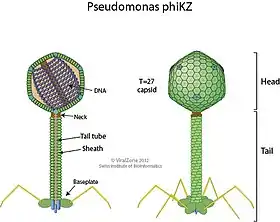
Aufbau
.jpg.webp)
.jpg.webp)
Die Virusteilchen (Virionen) in der Gattung Phikzvirus sind nicht umhüllt und haben einen Kopf und Schwanz, entsprechend ihrer Zugehörigkeit zur Ordnung Caudovirales („Schwanzviren“). Der Kopf hat eine ikosaedrische Symmetrie mit einer Triangulationszahl T=27, er hat einen relativ großen Durchmesser von etwa 140 nm. Der Schwanz ist ca. 160 nm lang und 35 nm breit.[2]
Genom
Das Genom besteht aus einem einzigen zirkulären Doppelstrang-DNA-Segment (dsDNA, monopartit). Einige der Spezies wurden vollständig sequenziert und sind beim ICTV abrufbar. Die Genom-Längen liegen zwischen 211 und 280 kbp (Kilo-Basenpaare). Es gibt 201 bis 306 kodierte Proteine.[24][2]
Vermehrungszyklus
Die Virusreplikation ist zytoplasmatisch, d. h. sie findet im Zellplasma statt. Das Virus bindet sich unter Verwendung seiner terminalen (endständigen, am Schwanzende befindlichen) Fasern an die Wirtszelle und stößt die virale DNA durch Kontraktion seiner Schwanzhülle in das Zellinnere des Wirtes aus. Die Transkriptionsmethode ist die DNA-gestützte Transkription.
Sobald die viralen Gene repliziert wurden, wird das Procapsid (Die Vorstufe des Kapsids) zusammengesetzt und die DNA verpackt. Danach wird der Schwanz zusammengesetzt und die reifen Virionen werden schließlich durch Lyse freigesetzt.
Die natürlichen Wirte sind Bakterien, die Übertragung geschieht durch passive Diffusion.[2]
Riesenphagen
.jpg.webp)
Doppelstrang-DNA-Phagen mit einer Genomgröße von mehr als 540 kbp werden als Megaphagen bezeichnet, kleinere mit mehr als 200 kbp als Jumbo-Phagen.[25] Von anderen Autoren wird die Grenze für Megaphagen bereits bei 500 kbp angesetzt.[26] Alle Phagen mit mehr als 200 kbp (also Jumbo-Phagen und Megaphagen) werden englisch als „huge phages“ (Riesenphagen) zusammengefasst.[26] Diese Bezeichnungen sind nicht-taxonomische Klassifizierungen, die per se keine Verwandtschaftsbeziehungen ausdrücken.
Forschungsgeschichte
Mit dem ICTV-Report von 2009 wurde die Gattung als PhiKZ-like viruses zusammen mit drei in ihr enthaltenen Arten erstmals als neue Gattung akzeptiert.[27] 2012 wurde der Name in Phikzlikevirus geändert, später in das aktuelle Phikzvirus.
Anmerkungen
- Das Material wurde von dieser Quelle kopiert, die unter einer Creative Commons Attribution 4.0 International License verfügbar ist.
Einzelnachweise
- ICTV: ICTV Taxonomy history: Enterobacteria phage T4, EC 51, Berlin, Germany, July 2019; Email ratification March 2020 (MSL #35)
- Viral Zone. ExPASy. Abgerufen am 6. April 2020.
- ICTV: ICTV Master Species List 2018b.v2, MSL #34v
- NCBI: Phikzvirus (genus)
- Victor Krylov, Maria Bourkaltseva, Elena Pleteneva, Olga Shaburova, Sergey Krylov, Alexander Karaulov, Sergey Zhavoronok, Oxana Svitich, Vitaly Zverev; Julie Thomas, Lindsay Black (Hrsg.): Phage phiKZ—The First of Giants. In: Viruses Band 13, Nr. 2, Sonderausgabe Giant or Jumbo Phages, 149, 20. Januar 2021, doi:10.3390/v13020149.

- V Chaikeeratisak, K Nguyen, ME Egan, ML Erb, A Vavilina, J Pogliano: The Phage Nucleus and Tubulin Spindle Are Conserved among Large Pseudomonas Phages.. In: Cell reports. 20, Nr. 7, 15. August 2017, S. 1563–1571. doi:10.1016/j.celrep.2017.07.064. PMID 28813669. PMC 6028189 (freier Volltext).
- Masaharu Takemura: Medusavirus Ancestor in a Proto-Eukaryotic Cell: Updating the Hypothesis for the Viral Origin of the Nucleus, in: Front. Microbiol. 11:571831, 3. September 2020, doi:10.3389/fmicb.2020.571831
- Annika Röcker: CRISPR-Cas: Gegen manche Viren ist die Genschere machtlos, auf: spektrum.de vom 10. Dezember 2019
- Victor Krylov, Olga Shaburova, Sergey Krylov, Elena Pleteneva: A Genetic Approach to the Development of New Therapeutic Phages to Fight Pseudomonas Aeruginosa in Wound Infections, in: MDPI Viruses Band 5, Nr. 1, 21. Dezember 2012, Special Issue Recent Progress in Bacteriophage Research, S. 15–53, doi:10.3390/v5010015
- NCBI: (no rank)
- NCBI: unclassified Phikzvirus
- NCBI: Pseudomonas phage 201phi2-1 (spezies)
- Pseudomonas phage 201phi2-1, auf: Virus-Host DB, TAX:198110
- Proteomes - Pseudomonas phage 201phi2-1, auf: UniProt
- NCBI: Pseudomonas phage PA02 (species)
- NCBI: Pseudomonas phage PA1C (spezies)
- NCBI: Pseudomonas phage PA1C (spezies)
- NCBI: Pseudomonas phage PaSz-1_45_270k (spezies)
- NCBI: Pseudomonas phage Phabio (spezies)
- NCBI: Pseudomonas phage PhiPA3 (spezies)
- NCBI: Pseudomonas phage Psa21 (spezies)
- NCBI: Pseudomonas phage vB_PaeM_PS119XW (spezies)
- ICTV: Elvirus
- NCBI: Phikzvirus Complete Genomes. Abgerufen am 8. April 2020.
- Audra E. Devoto, Joanne M. Santini et al.: Megaphages infect Prevotella and variants are widespread in gut microbiomes, in: Nature Microbiology, Band 4, S. 693–700, 28. Januar 2019, doi:10.1038/s41564-018-0338-9, insbes. Table 1 und Supplementary Figure 11
- Basem Al-Shayeb, Rohan Sachdeva, L. Chen, Jillian F. Banfield et al.: Clades of huge phages from across Earth’s ecosystems, in: Nature vom 12. Februar 2020, doi:10.1038/s41586-020-2007-4, zuvor bioRxiv: 10.1101/572362v1 (Preprint-Volltext). Die Autoren kürzen Kilobasenpaare bei Doppelstrang-Nukleinsäuren offenbar mit kb statt kbp ab.
- ICTV: ICTV Taxonomy history: Pseudomonas virus phiKZ
Weblinks
- SIB: ExPASy Viralzone: Phikzlikevirus
- ICTV: Virus Taxonomy
- NCBI: Caudovirales: Familien, Genera und Spezies