Cryptophyceae
Die Cryptophyceae (von altgriechisch κρύπτος kryptos, deutsch ‚geheim‘ und φύκιον phykion, deutsch ‚Alge‘) sind eine Klasse einzelliger, mikroskopisch kleiner Algen, die in Süß- und Meerwasser vorkommen. Die Cryptophyceen bewegen sich mit Hilfe von zwei Geißeln durch das Wasser und können rötlich, bläulich oder bräunlich gefärbt sein.[1] Einige Cryptophyceen bilden dickwandige und kugelige Dauerstadien, um ungünstige Umweltbedingungen zu überdauern. Cryptophyceen sind eine ökologisch sehr wichtige Algengruppe, dienen sie doch vielen Protisten als Nahrung. Da zu den Cryptophyceen farblose und photosynthetisch aktive Gattungen oder Arten gehören, gibt es botanische und zoologische Klassifizierungen. Sie können daher auch zoologisch als Cryptomonaden bezeichnet werden. Von ihrer Evolutionsgeschichte und ihren daraus resultierenden Verwandtschaften her gehören sie systematisch jedoch weder zu den Tieren noch zu den Pflanzen (nach momentanem Stand des Wissens werden im Reich Plantae sensu Archaeplastida die Glaucophyten, Rotalgen und Viridiplantae mit Grünalgen und Landpflanzen zusammengefasst).[2] Die nächsten Verwandten der Cryptophyceae sind die meist farblosen und phagotroph lebenden Katablepharidophyta.[3] Zusammen bilden sie das Taxon der Cryptophyta.
| Cryptophyceae | ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|
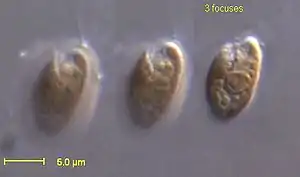
Rhodomonas salina | ||||||||||
| Systematik | ||||||||||
| ||||||||||
| Wissenschaftlicher Name | ||||||||||
| Cryptophyceae | ||||||||||
| Pascher 1913, emend. Schoenichen, 1925 |
Die meisten Cryptophyceen mit Ausnahme der basalen Gattung Goniomonas verfügen über zwei Zellkerne unterschiedlicher evolutionärer Herkunft und wurden daher für Evolutionsbiologen interessant. Die verschiedenen Zellkompartimente sind hierbei wie eine Matrjoschka ineinander verschachtelt. Das äußerste Kompartiment enthält den eigentlichen Zellkern und das Cytoplasma, in dem sich auch die Mitochondrien befinden. Das nächstkleinere sogenannte periplastidäre Kompartiment enthält den zweiten stark reduzierten Zellkern (Nucleomorph) und Stärkekörner. Das innerste Kompartiment ist das eigentliche Photosynthese-Organell, der Plastid. Da auch Mitochondrien und Plastiden über eigene Genome (Mitogenom, Plastom) verfügen, enthält eine Cryptophyceen-Zelle insgesamt vier Genome.
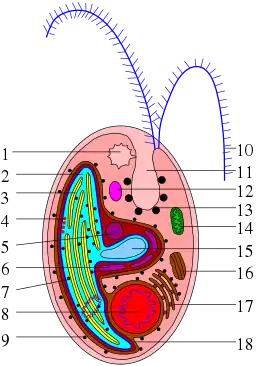
1: kontraktile Vacuole
2: Plastid bzw. Chloroplast
3: Thylakoid
4: Augenfleck (Stigma)
5: Nucleomorph
6: Stärkekörper (Granuli),
7: 70S-Ribosom
8: Zellkern (Nukleus)
9: 80S-Ribosom
10: Flagella
11: Zelleinstülpung (Invagination)
12: Lipid-Globuli
13: Ejektosome
14: Mitochondrium,
15: Pyrenoid
16: Golgi-Apparat
17: Endoplasmatisches Reticulum (ER)
18: ER des komplexen Plastiden/Chloroplasten
Erklärt wird die Vielzahl der Genome durch eine sekundäre Endosymbiose, bei der ein phagotropher Eukaryot einen photosynthetisch aktiven Eukaryoten aufnahm. Normalerweise werden aufgenommene Organismen verdaut. Bei einer Endosymbiose bleibt die aufgenommene Zelle jedoch erhalten und wandelt sich im Laufe der Zeit in ein unselbstständiges Organell um. Nur bei den Chlorarachniophyta und den mit ihnen nicht verwandten Cryptophyceen ging der Zellkern der aufgenommenen Alge nicht verloren.
Die marine Cryptophycee Guillardia theta wurde als Modellorganismus ausgewählt, um die Genome von Nucleomorph und Plastid zu sequenzieren.[4][5] Beide Genomsequenzierungen ergaben, dass der Plastid der Cryptophyceen ursprünglich eine Rotalge gewesen sein muss. Weitere Belege für diese Theorie sind die Stärkesynthese und die 80S-Ribosomen im periplastidären Raum (dem ehemaligen Cytoplasma der aufgenommenen Rotalge) und die vier Hüllmembranen, die den Plastiden umgeben. Alle Plastiden, die aus einer primären Endosymbiose stammen (die Chloroplasten der Grünalgen und Landpflanzen, die Cyanellen der Glaucocystophyten und die Rhodoplasten der Rotalgen), werden von nur zwei Hüllmembranen abgegrenzt und nicht von drei oder vier (= komplexe Plastiden).
Systematik der Cryptophyceae
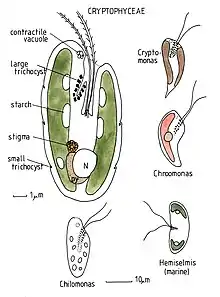
Cryptomonas, Chroomonas, Hemiselmis, Chilomonas.
Die systematische Einteilung der Cryptophyceen in verschiedene Gattungen erfolgte bisher hauptsächlich aufgrund von morphologischen Merkmalen und der Pigmentierung.
- Eines der wichtigsten Merkmale ist hierbei der Periplast. Der Periplast ist eine geschichtete Zellhülle – Cryptophyceen bilden keine Zellwand – bestehend aus einer inneren und einer äußeren Periplastenkomponente aus Proteinen. Dazwischen liegt die Plasmamembran der Zelle. Beide Periplastenkomponenten zeigen sehr feine Strukturierungen. Die innere Periplastenkomponente kann z. B. aus polygonalen Platten, sich überlappenden rektangulären Platten oder aus einer durchgehenden Schicht bestehen. Die äußere Periplastenkomponente kann sich auch aus Platten oder aus Rosettenschuppen und feinen Fibrillen zusammensetzen.
- Alle Cryptophyceen verfügen über eine Zelleinstülpung (Invagination), die mit explosiven Organellen, den sog. Ejekto- oder Ejektisomen, ausgekleidet ist. Die Öffnung dieser Zelleinstülpung kann entweder klein sein mit einem dahinterliegenden blind endenden Sack (Schlund) oder länglich gestreckt der Zelleinstülpung folgend (Furche). Auch Kombinationen aus Furche und Schlund sind möglich.
- Bei einigen Gattungen liegt der Nucleomorph nicht frei im periplastidären Raum, sondern ist in die Pyrenoid-Matrix eingebettet (räumlich abgetrennt durch die beiden inneren Hüllmembranen des Plastiden).
- Von den Phycobilisomen, den Lichtsammelkomplexen der Rotalgen (und Glaucocystophyceen und Cyanobakterien), die ursprünglich drei verschiedene blaue oder rote Pigmente enthielten, blieb bei den Cryptophyceen nur noch das Phycoerythrin übrig. Bei den Cryptophyceen evolvierten aus dem ursprünglich roten Phycoerythrin jedoch sieben verschiedene Phycoerythrin-Typen, von denen vier blau gefärbt sind und deshalb als Phycocyanine bezeichnet werden, obwohl sie mit den echten Phycocyaninen aus den Phycobilisomen nicht direkt verwandt sind.
- Die mikrotubulären Geißelwurzeln, mit denen die Geißeln in den Zellen verankert sind, weisen ebenfalls Unterschiede auf.
Aus der Kombination der verschiedenen Merkmale
- Struktur des Periplasten,
- Form der Zelleinstülpung,
- Position des Nucleomorphs,
- Pigment-Typ und
- Struktur des Geißelwurzelapparates
ergeben sich die verschiedenen Gattungen.
Die Erforschung der Verwandtschaftsverhältnisse innerhalb der Cryptophyceen mit Methoden der molekular-phylogenetischen Analyse (= Erstellung von Stammbäumen anhand von DNA-Sequenzen) ergab jedoch ein wesentlich komplexeres Bild. Cryptophyceen sind wahrscheinlich dimorph, das heißt, sie können zwei verschiedene Zelltypen ausbilden. Daher wurden wahrscheinlich fälschlicherweise mehrfach zwei Zellformen einer Gattung für zwei verschiedene Gattungen gehalten. Sicher nachgewiesen wurde Dimorphismus bei den Gattungen Proteomonas und Cryptomonas.[6][7]
Auch ein Leukoplast, ein farbloser Plastid, der die Fähigkeit zur Photosynthese verloren hat, ist kein sicheres Merkmal einer eigenständigen Gattung. Die ehemalige Gattung Chilomonas erwies sich als eine farblose Cryptomonas, von der außerdem mindestens drei verschiedene Evolutionslinien innerhalb von Cryptomonas existieren.[7][8] Auch die übrigen Gattungen der Cryptophyceen bedürfen wahrscheinlich einer Überarbeitung ihrer Systematik.[9][10][11]
Gattungen der Cryptophyceen nach vorläufigem Stand der Forschung sind: Chroomonas, Cryptomonas (enthält die ehemals eigenständigen Gattungen Campylomonas und Chilomonas), Geminigera, Goniomonas, Guillardia, Hanusia, Hemiselmis, Komma, Plagioselmis, Proteomonas, Rhinomonas, Rhodomonas (Pyrenomonas), Teleaulax.[12][13][14][6][15][16][17][7][18]
Eine von Tree of Life Web Project vorgeschlagene Taxonomie, ergänzt um Familien nach AlgaeBase, sowie um Falcomonas, lautet wie folgt:[19]
- Vertreter mit Plastiden:
- Cryptomonadaceae: Cryptomonas
- Chroomonadaceae: Chroomonas / Komma und Hemiselmis
- Chroomonadaceae: Falcomonas
- Pyrenomonadaceae: Rhodomonas / Rhinomonas / Storeatula
- Geminigeraceae: Guillardia / Hanusia
- Geminigeraceae: Geminigera / Plagioselmis / Teleaulax
- Geminigeraceae: Proteomonas
- basal:
- Goniomonadidae: Goniomonas
Literatur
- Karl-Heinz Linne von Berg, Michael Melkonian et al.: Der Kosmos-Algenführer. Die wichtigsten Süßwasseralgen im Mikroskop. Kosmos, Stuttgart 2004, ISBN 3-440-09719-6.
- Gary W. Saunders, Max H. Hommersand: Assessing red algal supraordinal diversity and taxonomy in the context of contemporary systematic data, in: American Journal of Botany. Band 91, Nr. 10, Oktober 2004, S. 1494–1507, PMID 21652305, doi:10.3732/ajb.91.10.1494
- Noriko Okamoto, Isao Inouye: The Katablepharids are a distant sister group of the Cryptophyta: A proposal for Katablepharidophyta divisio nova/Kathablepharida phylum novum based on SSU rDNA and beta-tubulin phylogeny, in: Protist, Band 156, Nr. 2, August 2005, PMID 16171184, doi:10.1016/j.protis.2004.12.003
- Susan E. Douglas, Susanne L. Penny: The plastid genome of the cryptophyte alga, Guillardia theta: complete sequence and conserved synteny groups confirm its common ancestry with red algae, in: Journal of Molecular Evolution, Band 48, Nr. 2, Februar 1999, S. 236–244, PMID 9929392, doi:10.1007/pl00006462
- Susan Douglas, Stefan Zauner, Martin Fraunholz, Margaret Beaton, Susanne Penny, Lang-Tuo Deng, Xiaonan Wu, Michael Reith, Thomas Cavalier-Smith, Uwe-G. Maier: The highly reduced genome of an enslaved algal nucleus, in: Nature, Band 410, London, 26. April 2001, S. 1091–1096, doi:10.1038/35074092
- David R. A. Hill, Richard Wetherbee: Proteomonas sulcata gen. et sp. nov. (Cryptophyceae), a cryptomonad with two morphologically distinct and alternating forms, in: Phycologia, Band 25, Nr. 4, 1986, S. 521–543, doi:10.2216/i0031-8884-25-4-521.1, Epub 6. März 2019, doi:10.2216/i0031-8884-25-4-521.1
- Kerstin Hoef-Emden, Michael Melkonian: Revision of the Genus Cryptomonas (Cryptophyceae): a Combination of Molecular Phylogeny and Morphology Provides Insights into a Long-Hidden Dimorphism, in: Protist, Band 154, Nr. 3–4, Oktober 2003, doi:10.1078/143446103322454130
- Kerstin Hoef-Emden: Multiple Independent Losses of Photosynthesis and Differing EvolutionaryRates in the Genus Cryptomonas (Cryptophyceae): Combined Phylogenetic Analyses of DNA Sequences of the Nuclear and the Nucleomorph Ribosomal Operons, in: Journal of Molecular Evolution, Band 60, Nr. 2, Februar 2005, S. 183–195, PMID 15785847, doi:10.1007/s00239-004-0089-5
- James A. Deane, Isabelle M. Strachan, Gary W. Saunders, David R. A. Hill, Geoffrey I. McFadden: Cryptomonad evolution: nuclear 18S rDNA phylogeny versus cell morphology and pigmentation, in: Journal of Phycology, Band 38, Nr. 6, S. 1236–1244, Dezember 2002, doi:10.1046/j.1529-8817.2002.01250.x
- Kerstin Hoef-Emden, Birger Marin, Michael Melkonian: Nuclear and nucleomorph SSU rDNA phylogeny in the Cryptophyta and the evolution of cryptophyte diversity, in: Journal of Molecular Evolution, Band 55, Nr. 2, August 2002, S. 161–179, PMID 12107593, doi:10.1007/s00239-002-2313-5
- Birger Marin, Max Klingberg, Martin Melkonian: Phylogenetic relationships among the Cryptophyta: analyses of nuclear-encoded SSU rRNA sequences support the monophyly of extant plastid containing lineages, in: Protist, Band 149, Nr. 3, September 1998, S. 265–276, PMID 23194638, doi:10.1016/S1434-4610(98)70033-1, Epub 13. Juli 2009
- James A. Deane, David R. A. Hill, Steven J. Brett, Geoffrey I. McFadden: Hanusia phi gen. et sp. nov. (Cryptophyceae): Characterization of ´Cryptomonas sp. Φ´, in: European Journal of Phycology, Band 33, Nr. 2, 1. Mai 1998, S. 149–154., doi:10.1080/09670269810001736643, Epub 3. Juni 2010
- David R. A. Hill: A revised circumscription of Cryptomonas (Cryptophyceae) based on an examination of Australian strains, in: Phycologia, Band 30, 1991, S. 170–188, doi:10.2216/i0031-8884-30-2-170.1, Epub 6. März 2019
- David R. A. Hill: Chroomonas and other blue-green cryptomonads, in: Journal of Phycology, Band 27, Nr. 1, Februar 1991, S. 133–145, Project: Taxonomic studies on cryptomonad algae (Cryptophyceae), doi:10.1111/j.0022-3646.1991.00133.x, Epub Oktober 2004
- David R. A. Hill, Richard Wetherbee: The structure and taxonomy of Rhinomonas pauca gen. et sp. nov. (Cryptophyceae), in: Phycologia, Band 27, Nr. .3, 1988, S. 355–365, doi:10.2216/i0031-8884-27-3-355.1, Epub 6. März 2019
- David R. A. Hill, Richard Wetherbee: A reappraisal of the genus Rhodomonas (Cryptophyceae), in: Phycologia, Band 28, Nr. 2, 1989, S. 143–158, doi:10.2216/i0031-8884-28-2-143.1, Epub 6. März 2019
- David R. A. Hill, Richard Wetherbee: Guillardia theta gen. et sp. nov. (Cryptophyceae), in: Canadian Journal of Botany, Band 68, Nr. 9, 1990, S. 1873–1876, Project: Taxonomic studies on cryptomonad algae (Cryptophyceae), doi:10.1139/b90-245, Epub Februar 2011, ResearchGate
- Geoffrey I. McFadden, Paul R. Gilson, David R. A. Hill: Goniomonas: rRNA sequences indicate that this phagotrophic flagellate is a close relative of the host component of cryptomonads, in: European Journal of Phycology, Band 29, Nr. 1, 1994, S. 29–32, doi:10.1080/09670269400650451
- Cryptomonads. In: Tree of Life Project. 2. April 2010. Abgerufen am 7. Dezember 2021.
Weblinks
- The Tree of Life Web Project: Cryptomonads
- Kerstin Hoef-Emden: Cryptophyte Morphology, Ecology & Evolution, Universität Köln