Lokiarchaeota
Die Lokiarchaeota (deutsch: Lokiarchaeen) sind eine systematische Gruppe von Mikroorganismen im taxonomischen Rang eines Stammes, die der Domäne der Archaeen zugeordnet wird. Die Errichtung des Taxons im Jahr 2015 fußte vor allem auf molekulargenetischen Daten.
| „Lokiarchaeota“[1] | ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|

Prometheoarchaeum syntrophicum, künstlerische Nachbildung in Plastilin. | ||||||||||
| Systematik | ||||||||||
| ||||||||||
| Wissenschaftlicher Name | ||||||||||
| „Lokiarchaeota“[1] | ||||||||||
| Spang et al. 2015[2] |

Obgleich die Lokiarchaeen Prokaryoten sind, finden sich in ihnen einige Gene, welche bislang nur bei Tieren, Pflanzen, Pilzen und anderen Eukaryoten gefunden wurden, sogenannte eukaryotische Signaturgene. Die Lokiarchaeen werden daher auch als eine Art Missing Link zwischen Archaeen und Eukaryoten diskutiert. Es sind jedoch weitere Untersuchungen nötig, um die Missing-Link-Hypothese zu festigen, beispielsweise der Nachweis eines dynamischen Zytoskeletts unter dem Mikroskop.
Das organische Material, in dem die spezielle genetische Signatur der Lokiarcheen identifiziert wurde, stammt aus Proben von marinen Sedimenten, die in einem Geothermalgebiet in der arktischen Tiefsee zwischen Nordwesteuropa und Grönland entnommen wurden. Dieses Geothermalgebiet wurde wegen der bizarren Formen der Schlote seiner Schwarzen Raucher von seinen Entdeckern „Lokis Schloss“ (englisch Loki’s Castle) genannt. Der Name „Lokiarchaeota“ nimmt zudem darauf Bezug, dass die Lokiarchaeen ihre Zellform ändern können und der nordische Gott Loki, der Sage nach, ein Formwandler war.[1]
Entdeckung und Beschreibung
„Lokis Schloss“ (73,55° N, 8,15° O) ist ein Geothermalgebiet in einer vulkanisch aktiven Tiefseeregion, in dem sich Schlote aus sulfidischen Mineralen über heißen Quellen, sogenannten Schwarzen Rauchern, gebildet haben. Es befindet sich in rund 2300 m Tiefe in der Übergangsregion vom Nordatlantik in den Arktischen Ozean auf dem Mittelozeanischen Rücken zwischen Grönland und Skandinavien (Übergang vom Mohn-Rücken in den Knipowitsch-Rücken).[4]
Im Jahr 2010 wurden dort Sedimentproben entnommen, deren metagenomische Analyse durch Teams aus Bergen, Uppsala in der Arbeitsgruppe von Christa Scheper an der Universität Wien[5] eine Fülle von Hinweisen auf bis dahin unbekannte Archaeen lieferte.[6][7] Dazu gehörte Lokiarchaeum, das bislang noch nicht im Labor in Reinkultur gezüchtet werden konnte.[8] Wegen der niedrigen Zelldichte (Zellen pro Gramm Sediment) stammt die ermittelte DNA-Sequenz von Lokiarchaeum eher nicht von einer isolierten Zelle, sondern eher aus der Kombination von Genfragmenten mehrerer Individuen. Das gefundene Genom ist zu 92 % komplett und 1,4-fach redundant. Die Fragmente stammen von drei Spezies eines neuen Taxons[1] (siehe Abbildung unten).
Das Genom von Lokiarchaeum setzt sich aus 5381 Protein codierenden Genen zusammen. Davon passen 32 % nicht zu bekannten Proteinen, 26 % sind eng mit den Proteinen bekannter Archaeen verwandt und 29 % mit bakteriellen Proteinen. Diese Zusammensetzung spricht für Folgendes:
- Es handelt sich um Proteine eines neuen Stammes, der der Domäne der Archaeen ein neues basales Taxon hinzufügt.
- Es hat ein besonders intensiver horizontaler Gentransfer von Bakterien zu Archaeen stattgefunden. (Zum Vergleich: bei Methanosarcinen wurde „nur“ ein Anteil von 5 % Genen bakteriellen Ursprungs gefunden.[9]).
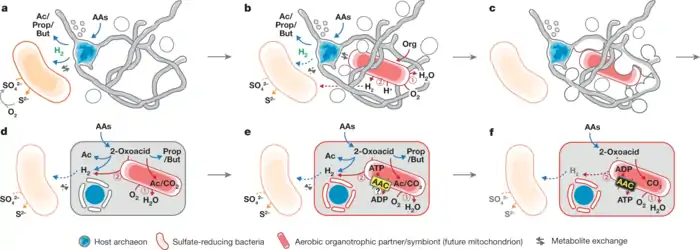
Ein kleiner, aber signifikanter Anteil der Gene (175 = 3,3 %) von Lokiarchaeum ähnelt stark den Genen von eukaryotischen Proteinen. Diese für Prokaryoten sehr ungewöhnlichen Gene stammen kaum aus Verunreinigungen der Proben, da sie stets von prokaryotischen Gensequenzen flankiert waren. Im Metagenom der Sedimentproben des thermophilen Biotops konnten wie erwartet keine Gene eukaryotischen Ursprungs entdeckt werden.[1] Proteine, die Lokiarchaeum mit Eukaryoten gemeinsam hat, sind bei letzteren Bestandteile des Cytoskeletts und dienen der Verformung der Zellmembran und der Zellform.[1][10][11] Anscheinend teilt Lokiarchaeum diese Fähigkeit.[1] Ein anderes gemeinsames Protein, nämlich Aktin, ist essenziell für die Phagocytose der Eukaryoten,[8][10] also der Fähigkeit der Organismen Partikel zu umfließen und in die Zelle aufzunehmen. Wenn sich bestätigt, dass auch Lokiarchaeen zur Phagocytose fähig sind, würde das gut erklären, wie es zur Symbiose von Archaeen und Bakterien kam. Lokiarchaeen oder enge Verwandte könnten Bakterien einverleibt und als Symbionten genutzt haben, welche sich dann nach der allgemein akzeptierten Endosymbiontentheorie zu Mitochondrien entwickelten.[1][12]
Innere und äußere Taxonomie der „Lokiarchaeota“ und Bedeutung in der Evolution

Eines der drei gefundenen Teilgenome (vgl. nebenstehende Abbildung) weist einen signifikant niedrigeren GC-Gehalt als die beiden anderen auf. Das bedeutet, dass sie einen unterschiedlichen Gehalt der DNA-Basen Guanin und Cytosin in ihrer DNA aufweisen. Dieser Unterschied kann nur aus einer erheblichen Menge an unterschiedlichen Punktmutationen der beiden Äste der Lokiarchaeota resultieren.
Die vergleichende Untersuchung der Genome von Lokiarchaeum und denen von bekannten Eukaryoten lässt stark vermuten, dass diese Organismen eine gemeinsame phylogenetische Vergangenheit und einen gemeinsamen monophyletischen Stammbaum haben.[1][13][14][15]
Das allgemein anerkannte Drei-Domänen-Modell von Carl Woese teilt alle zellulären Lebewesen ein in Archaea, Bakterien und Eukaryoten. Eukaryoten sind alle Vielzeller wie Tiere, Pilze, Pflanzen und die Protozoen. Sie sind gemeinsam charakterisiert durch ihre großen, hoch entwickelten Zellen, deren Energiehaushalt durchweg auf mitochondrialen ATP-Synthasen beruht und bei denen die DNA in eine Kernmembran eingebettet ist. Bakterien und Archaeen sind anscheinend ihre Vorfahren,[16] und es wurden 3,8 Milliarden Jahre alte fossile Spuren der Lipide von Archaeen gefunden.[17] Die Evolution der Eukaryoten war vermutlich erst vor 1,6 bis 2,1 Milliarden Jahren abgeschlossen.[18] Für diese Evolution aus prokaryotischen Archaeen[1][19][20] ist Lokiarchaeum anscheinend ein Missing Link. Dessen letzter gemeinsame Vorfahr mit den Eukaryoten hatte vor ca. zwei Milliarden Jahren vermutlich die Gene entwickelt, die unabdingbar für das Entstehen der komplexen eukaryotischen Zellstrukturen waren[21] und dafür wie ein „Starter-Kit“ wirkten.[5]
Die Lokiarchaeota werden selber in die 2014 von Céline Petitjean und Kollegen beschriebene Abteilung Proteoarchaeota gestellt.[22] Sie bilden zusammen mit ihren später gefundenen Schwestergruppen "Thorarchaeota", "Odinarchaeota" und "Heimdallarchaeota" die Untergruppe "Asgard" der Proteoarchaeota. "Asgard" erscheint dann selbst als Schwestergruppe von "TACK".[23] Entsprechend kladistischer Argumentation wären dann die Eukaryota (und damit der Mensch) ebenfalls der Asgard-Gruppe zugehörig.
Eine Zusammenfassung dieser Diskussion findet sich bei Traci Watson (2019).[24]
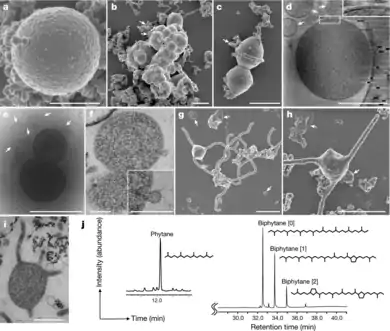
Nach 12 Jahren Forschungsarbeit berichten Mikrobiologen 2019 erfolgreich ein, sich nur sehr langsam vermehrendes, Lokiarchaeota aus Tiefseeschlamm isoliert und kultiviert zu haben. Das Prometheoarchaeum syntrophicum Strang MK-D1 hat lange „Tentakel“, in denen Partnermikroben nisten, welche ihm womöglich als „Protomitochondrien“ verbesserte Überlebenschancen bei steigendem Sauerstoff während der Großen Sauerstoffkatastrophe ermöglichen konnten und von den Tentakeln – als Vorfahren der Mitochondrien – umschlossen und endogenisiert wurden. Laut ihrem E3-Modell fand so die Eukaryogenese statt.[25][26][3]
Einzelnachweise
- Anja Spang, Jimmy H. Saw, Steffen L. Jørgensen, Katarzyna Zaremba-Niedzwiedzka, Joran Martijn, Anders E.Lind, Roel van Eijk, Christa Schleper, Lionel Guy, Thijs J. G. Ettema: Complex archaea that bridge the gap between prokaryotes and eukaryotes. In: Nature. Band 521, 2015, S. 173–179, doi:10.1038/nature14447, PMID 25945739, PMC 4444528 (freier Volltext).
- NCBI: Candidatus Lokiarchaeota (phylum, equivalent: Lokiarchaeota Spang et al. 2015); graphisch: Candidatus Lokiarchaeota, auf: Lifemap, NCBI Version
- Hiroyuki Imachi, Masaru K. Nobu, Nozomi Nakahara, Yuki Morono, Miyuki Ogawara, Yoshihiro Takaki, Yoshinori Takano, Katsuyuki Uematsu, Tetsuro Ikuta, Motoo Ito, Yohei Matsui, Masayuki Miyazaki, Kazuyoshi Murata, Yumi Saito, Sanae Sakai, Chihong Song, Eiji Tasumi, Yuko Yamanaka, Takashi Yamaguchi, Yoichi Kamagata, Hideyuki Tamaki & Ken Takai: Isolation of an archaeon at the prokaryote–eukaryote interface. In: Nature. Band 577, 2020, S. 519–525, doi:10.1038/s41586-019-1916-6 (nature.com).
- Rolf B. Pedersen, Hans Tore Rapp, Ingunn H. Thorseth, Marvin D. Lilley, Fernando J. A. S. Barriga, Tamara Baumberger, Kristin Flesland, Rita Fonseca, Gretchen L. Früh-Green, Steffen L. Jorgensen: Discovery of a black smoker vent field and vent fauna at the Arctic Mid-Ocean Ridge. In: Nature Communications. 1, Art.-Nr. 126, 2010. doi:10.1038/ncomms1124.
- Christa Schleper, Department für Ökogenomik und System Biologie der Universität Wien (Wissenschaftlicher Kontakt): Neue komplexe Archaea entdeckt. Nächste Verwandte der Eukaryoten. In: LABO Online, 7. Mai 2015. Abgerufen am 17. Februar 2016.
- Steffen Leth Jorgensen, Bjarte Hannisdal, Anders Lanzén, Tamara Baumberger, Kristin Flesland, Rita Fonseca, Lise Øvreås, Ida H. Steen, Ingunn H. Thorseth, Rolf B. Pedersen, Christa Schleper: Correlating microbial community profiles with geochemical data in highly stratified sediments from the Arctic Mid-Ocean Ridge. In: Proceedings of the National Academy of Sciences. Band 109, Nr. 42, 16. Oktober 2012, ISSN 0027-8424, S. E2846–E2855, doi:10.1073/pnas.1207574109 (pnas.org).
- Steffen Leth Jørgensen, Ingunn Hindenes Thorseth, Rolf Birger Pedersen, Tamara Baumberger, Christa Schleper: Quantitative and phylogenetic study of the Deep Sea Archaeal Group in sediments of the Arctic mid-ocean spreading ridge. In: Extreme Microbiology. Band 4, 4. Oktober 2013, S. 299, doi:10.3389/fmicb.2013.00299 (frontiersin.org).
- Paul Rincon: Newly found microbe is close relative of complex life. 6. Mai 2015.
- Sofya K. Garushyants, Marat D. Kazanov, Mikhail S. Gelfand: Horizontal gene transfer and genome evolution in Methanosarcina. In: BMC Evolutionary Biology. 15, Nr. 1, 2015, S. 1–14. doi:10.1186/s12862-015-0393-2.
- Umesh Ghoshdastider, Shimin Jiang, David Popp, Robert C. Robinson: In search of the primordial actin filament. In: Proceedings of the National Academy of Sciences. Band 112, Nr. 30, 28. Juli 2015, ISSN 1091-6490, S. 9150–9151, doi:10.1073/pnas.1511568112, PMID 26178194.
- Amina Khan: Meet Loki, your closest-known prokaryote relative. In: Los Angeles Times. 6. Mai 2015.
- Steven G. Ball, Debashish Bhattacharya, Andreas P. M. Weber: Pathogen to powerhouse. In: Science. 351, Nr. 6274, 2016, S. 659–660. doi:10.1126/science.aad8864.
- T. Martin Embley, William Martin: Eukaryotic evolution, changes and challenges. In: Nature. Band 440, Nr. 7084, 30. März 2006, ISSN 1476-4687, S. 623–630, doi:10.1038/nature04546, PMID 16572163.
- J. A. Lake: Origin of the eukaryotic nucleus determined by rate-invariant analysis of rRNA sequences. In: Nature. Band 331, Nr. 6152, 14. Januar 1988, ISSN 0028-0836, S. 184–186, doi:10.1038/331184a0, PMID 3340165.
- Lionel Guy, Thijs J. G. Ettema: The archaeal 'TACK' superphylum and the origin of eukaryotes. In: Trends in Microbiology. Band 19, Nr. 12, 1. Dezember 2011, ISSN 1878-4380, S. 580–587, doi:10.1016/j.tim.2011.09.002, PMID 22018741.
- Minglei Wang, Liudmila S. Yafremava, Derek Caetano-Anollés, Jay E. Mittenthal, Gustavo Caetano-Anollés: Reductive evolution of architectural repertoires in proteomes and the birth of the tripartite world. In: Genome Research. Band 17, Nr. 11, 1. November 2007, ISSN 1088-9051, S. 1572–1585, doi:10.1101/gr.6454307, PMID 17908824.
- Jürgen Hahn, Pat Haug: Traces of archaebacteria in ancient sediments. In: Systematic and Applied Microbiology. Band 7, Nr. 2, 1. Mai 1986, S. 178–183, doi:10.1016/S0723-2020(86)80002-9 (sciencedirect.com).
- A. H. Knoll, E. J. Javaux, D. Hewitt, P. Cohen: Eukaryotic organisms in Proterozoic oceans. In: Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. Band 361, Nr. 1470, 29. Juni 2006, ISSN 0962-8436, S. 1023–1038, doi:10.1098/rstb.2006.1843, PMID 16754612.
- Purificación López-García, David Moreira: Open Questions on the Origin of Eukaryotes. In: Trends in Ecology & Evolution. 30, Nr. 11, 2015, S. 697–708. doi:10.1016/j.tree.2015.09.005.
- Tom A. Williams, Peter G. Foster, Cymon J. Cox, T. Martin Embley: An archaeal origin of eukaryotes supports only two primary domains of life. In: Nature. 504, Nr. 7479, 2013, S. 231–236. doi:10.1038/nature12779.
- Carl Zimmer: Under the Sea, a Missing Link in the Evolution of Complex Cells. In: The New York Times. 6. Mai 2015, ISSN 0362-4331 (nytimes.com).
- Petitjean, C., Deschamps, P., López-García, P., and Moreira, D.: Rooting the Domain archaea by phylogenomic analysis supports the foundation of the new kingdom proteoarchaeota.. In: Genome Biol. Evol.. 7, 2014, S. 191–204. doi:10.1093/gbe/evu274.
- K Zaremba-Niedzwiedzka et al.: Asgard archaea illuminate the origin of eukaryotic cellular complexity. In: Nature. 541, 2017, S. 353–358. doi:10.1038/nature21031.
- Traci Watson: The trickster microbes that are shaking up the tree of life, in: Nature vom 14. Mai 2019 (englisch), Trickser-Bakterien schütteln den Stammbaum des Lebens, in: Spektrum.de vom 20. Juni 2019 (deutsch) – die Bezeichnung ‚Bakterien‘ ist nicht ganz korrekt, bei den betrachteten Mikroben handelt es sich um Archaeen oder (nach Ansicht mancher Forscher) jedenfalls um von den Bakterien verschiedene Proto-Eukaryonten.
- https://www.nytimes.com/2020/01/15/science/cells-eukaryotes-archaea.html
- Jonathan Lambert: Scientists glimpse oddball microbe that could help explain rise of complex life. In: Nature. Band 572, 2019, S. 294 (nature.com).