Methanosarcina barkeri
Methanosarcina barkeri ist eine Art von prokaryotischen Mikroorganismen.[1] M. barkeri ist anaerob, ein Methanbildner und gehört in die Domäne der Lebewesen Archaea.[A 1] Es ist die Typusart der Gattung Methanosarcina.[2]
| Methanosarcina barkeri | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|

Methanosarcina barkeri (Stamm Fusaro) | ||||||||||||
| Systematik | ||||||||||||
| ||||||||||||
| Wissenschaftlicher Name | ||||||||||||
| Methanosarcina barkeri | ||||||||||||
| Schnellen 1947 |
Fundorte und Morphologie
Der heutige Typstamm (Methanosarcina barkeri MST) wurde 1966 aus einem Faulbehälter für Klärschlamm aus Haushalten in Urbana (Illinois) isoliert.[3] Der Fusaro-Stamm vom M. barkeri wurde in Schlammproben aus dem Lago Fusaro, einem Süßwassersee in der Nähe von Neapel gefunden.[4][5] M. barkeri findet sich auch im Pansen von Rindern, wo es synergetisch mit anderen Mikroorganismen an der Verdauung von biogenen Polymeren wie Cellulose arbeitet.[6][7] M. barkeri bevorzugt Süßwassersysteme[8][9] und wurde vor allem aus Schlamm von Kläranlagen, Gräben und Seen isoliert.[9]
M. barkeri zeigt eine dichotome Morphologie: wenn diese Mikroben in Süßwassermedium gezüchtet werden, wachsen sie zu großen, vielzelligen Aggregaten heran, die in einer Matrix aus sogenannten Methanochondroitin eingebettet sind, während sie in einem marinen Milieu als einzelne, unregelmäßige Kokken wachsen,[10] die nur von einer Proteinschicht (S-Schicht), aber nicht vom Methanochondroitin umgeben sind.[4] M. barkeri hat zwar offene Leserahmen (ORFs) für die N-Acetylmuraminsäure-Synthese,[4] es wurde aber kein Murein in der Zellwand vorgefunden.[11] Während eine einzelne Zelle von M. barkeri lediglich die S-Schicht aufweist, die bei Archaeen sozusagen die „Standardausstattung“ der Zellwand ist, haben viele Archaeen zusätzliche Schichten, wobei die Ablagerung von Methanochondroitin über der S-Schicht eine Besonderheit von Methanosarcina-Zellaggregate ist.[12] Methanochondroitin ist ein heterogenes Polysaccharid auf der Basis von N-Acetyl-D-galactosamin und D-Glucuronsäure[13] und ähnelt in einigen Punkten dem Chondroitin im Bindegewebe von Wirbeltieren.[14][15]
M. barkeri (Stamm Fusaro) hat kein Flagellum, kann sich aber möglicherweise durch die Bildung von gasgefüllten Vesikeln bewegen.[4] Diese Vesikel bilden sich aber nur in Gegenwart von Wasserstoff und Kohlendioxid, wahrscheinlich als Antwort auf einen Wasserstoffgradienten.[4] Das Genom, das aus einem ringförmigen Chromosom und einem zusätzlichen, ebenfalls ringförmigen, extrachromosomalen Element besteht, bietet die bemerkenswerte Fähigkeit, eine breite Vielfalt an Molekülen zu metabolisieren.[4] Das bringt der Mikrobe wahrscheinlich den Vorteil, sich trotz ihrer Unbeweglichkeit oder eingeschränkten Beweglichkeit an die Umwelt anpassen zu können, je nachdem, welche Energiequelle zur Verfügung steht. Das Genom von M. barkeri weist 3680 offene Leserahmen auf, die vermutlich Gene darstellen.[4]
Physiologie und Stoffwechsel
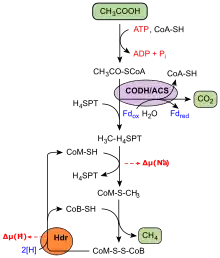
M. barkeri kann auf verschiedenen Stoffwechselwegen Methan produzieren und verschiedene Substrate zur Produktion von ATP nutzen, wie Methanol, Methylamin, Acetat sowie Wasserstoff mit Kohlendioxid.[16] Außerdem wurde schon 1947 stöchiometrisch bestimmt, dass M. barkeri aus vier mol Kohlenmonoxid und zwei mol Wasser drei mol Kohlendioxid und ein mol Methan produziert.[17][18] M. barkeri hat keine hohe Affinität für Wasserstoff und für Acetat, kann aber beides nutzen; dadurch erlangt es vermutlich Wachstumsvorteile gegenüber anderen Methanbildnern, wenn diese beiden Stoffe in hohen Konzentrationen vorhanden sind, z. B. in Kläranlagen.[19] Mindestens zwei Stämme von M. barkeri (Fusaro und MS) enthalten gleichzeitig DNA-Sequenzen für die unterschiedlichen ATP-Synthase-Typen A und F: der A-Typ ist für Archaeen typisch (zu denen Methanosarcina gehört) und der F-Typ ist für Bakterien, Mitochondrien und Chloroplasten typisch.[20] M. barkeri war der erste Organismus, in dem die Aminosäure Pyrrolysin gefunden wurde.[21]
Systematik
Methanosarcina barkeri wurde 1947 durch Schnellen beschrieben[22] und 1980 durch die Internationale Vereinigung mikrobiologischer Gesellschaften (IUMS) bestätigt.[1]
Seit 1986 ist „Methanosarcina barkeri Schnellen 1947“ die Typusart der Gattung Methanosarcina.[2]
Der Typstamm der Art Methanosarcina barkeri ist MS.[23] Der Stamm MST wurde in verschiedenen Sammlungen für Kulturstämme hinterlegt (DSM 800,[24] ATCC 43569[25] und JCM 10043[26]).
Eine frühe Klassifizierung nach phylogenetischen Verwandtschaftsverhältnissen, die M. barkeri einbezog, erfolgte 1979 durch die Analyse von 16S-RNA.[16] Die aktuelle Einteilung und die Nomenklatur sind in der LPSN einsehbar (Abruf 2019-05[27][28]).
Historische Zuordnung von Stämmen
Die Gattung Methanosarcina und ihre erste Art M. methanica wurden 1936 beschrieben[29] und 1980 als Gattung und Typusart bestätigt.[1] Da zur ursprünglichen Beschreibung von M. methanica kein passender Kulturstamm mehr vorhanden war und auch nicht entdeckt werden konnte, wurde 1984 vorgeschlagen, M. barkeri als neue Typusart der Gattung zu erwählen.[30] 1986 wurde M. barkeri als Typusart für die Gattung Methanosarcina eingesetzt.[2][31] Für M. barkeri lagen allerdings mehrere Stämme vor, die jeweils als Typstamm dieser Typusart in Frage kamen; das war zum einen der M.-barkeri-Stamm MS (DSM 800), der 1980 als Typstamm der Art bestätigt wurde[1] und zum anderen der M.-barkeri-Stamm 227 (DSM 1538), der zusammen mit dem Vorschlag,[30] M. methanica durch M. barkeri zu ersetzen, in die Debatte eingebracht wurde. 1987 erfolgte eine bessere Beschreibung des M.-barkeri-Stamms MS (DSM 800) und damit seine endgültige Bestätigung als Typstamm der Art (Methanosarcina barkeri MST).[23] 1992 wurden wichtige Kulturstämme der Gattung Methanosarcina charakterisiert und gegenübergestellt, so dass dies zu einer verbesserten Beschreibung der Art M. barkeri beitrug.[9]
Anwendungen und Bedeutung
Methanosarcina barkeri weist einige Eigenschaften auf, die für technische Anwendungen nutzbar sein können. Für das Archäon M. barkeri wurde in besonderen Maße untersucht, wie die Methanogenese durch syntrophische Beziehungen zu Bakterien gefördert werden kann; das trifft auf Pelobacter carbinolicus und Geobacter metallireducens zu.[32] In der Kombination mit P. carbinolicus kann das Archäon Wasserstoff (HIT, hydrogen interspecies transfer) und in der Kombination mit G. metallireducens direkt übertragene Elektronen (DIET, direct interspecies electron transfer) nutzen.[32] Allerdings finden Untersuchungen zur bioelektrochemischen Methanogenese noch im Labormaßstab statt.[33] M. barkeri wird an Orten mit extrem niedrigen Sauerstoffkonzentrationen wie dem Pansen von Rindern gefunden und als extremer Anaerobier eingestuft.[34] Das dort produzierte Methan bildet einen nennenswerten Beitrag zum Treibhauseffekt.[34] Bei kontrolliertem Auffangen lässt sich dieses Methan zur Energiegewinnung nutzen, ein Nebeneffekt ist beispielsweise die Reinigung von Abwasser.[35] Da M. barkeri unter extremen Bedingungen überleben kann, ist es möglich, das Archäon in Umgebungen mit sehr niedrigem pH-Wert einzusetzen, um die Säure effektiv zu neutralisieren und die Bedingungen für andere Methanbildner tolerabel zu gestalten.[34]
Datenbanken
- LPSN, Methanosarcina → http://www.bacterio.net/methanosarcina.html
- NCBI taxonomy browser, M. barkeri → https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=2208
- UCSC genome browser, M. barkeri → http://microbes.ucsc.edu/cgi-bin/hgGateway?db=methBark1
- KEGG, M. barkeri → https://www.genome.jp/dbget-bin/www_bfind_sub?mode=bfind&max_hit=1000&dbkey=alldb&keywords=methanosarcina+barkeri
- BacDive, M. barkeri → https://bacdive.dsmz.de/search?search=Methanosarcina+barkeri&submit=
- MicrobeWiki, M. barkeri → https://microbewiki.kenyon.edu/index.php/Methanosarcina_barkeri
Anmerkungen
- Die Zuordnung zur Domäne der Archaeen erfolgte nachträglich durch Wissenszuwachs (Woese et al. 1990, PMID 2112744). Zur Zeit der Artbeschreibung (Schellen 1947), bzw. zur Zeit der Bestätigung (Approved Lists 1980) wurde zwischen Bakterien und Archaeen noch nicht unterschieden. Siehe auch #Systematik in diesem Artikel.
Einzelnachweise
- P. H. A. Sneath, Vicki McGOWAN, V. B. D. Skerman: Approved Lists of Bacterial Names. In: International Journal of Systematic and Evolutionary Microbiology. Band 30, Nr. 1, 1. Januar 1980, ISSN 1466-5026, S. 225–420, doi:10.1099/00207713-30-1-225.
- International Union of Microbiological Societies: Opinion 63: Rejection of the Type Species Methanosarcina methanica (Approved Lists, 1980) and Conservation of the Genus Methanosarcina (Approved Lists, 1980) emend. Mah and Kuhn 1984 with Methanosarcina barkeri (Approved Lists, 1980) as the Type Species. In: International Journal of Systematic Bacteriology. 36, 1986, S. 492, doi:10.1099/00207713-36-3-492.
- M. P. Bryant, D. R. Boone: Emended Description of Strain MST(DSM 800T), the Type Strain of Methanosarcina barkeri. In: International Journal of Systematic Bacteriology. Band 37, Nr. 2, 1. April 1987, ISSN 0020-7713, S. 169–170, doi:10.1099/00207713-37-2-169.
- D. L. Maeder, I. Anderson, T. S. Brettin, D. C. Bruce, P. Gilna, C. S. Han, A. Lapidus, W. W. Metcalf, E. Saunders, R. Tapia, K. R. Sowers: The Methanosarcina barkeri genome: comparative analysis with Methanosarcina acetivorans and Methanosarcina mazei reveals extensive rearrangement within methanosarcinal genomes. In: Journal of bacteriology. Band 188, Nummer 22, November 2006, S. 7922–7931, doi:10.1128/JB.00810-06, PMID 16980466, PMC 1636319 (freier Volltext).
- O. Kandler, H. Hippe: Lack of peptidoglycan in the cell walls of Methanosarcina barkeri. In: Archives of Microbiology. Band 113, Nr. 1-2, 13. Mai 1977, ISSN 0302-8933, S. 57–60, PMID 889387.
- Suzanne C. Lambie, William J. Kelly, Sinead C. Leahy, Dong Li, Kerri Reilly: The complete genome sequence of the rumen methanogen Methanosarcina barkeri CM1. In: Standards in Genomic Sciences. Band 10, 2015, ISSN 1944-3277, S. 57, doi:10.1186/s40793-015-0038-5, PMID 26413197, PMC 4582637 (freier Volltext).
- G. N. Jarvis, C. Strömpl, D. M. Burgess, L. C. Skillman, E. R. Moore: Isolation and identification of ruminal methanogens from grazing cattle. In: Current Microbiology. Band 40, Nr. 5, Mai 2000, ISSN 0343-8651, S. 327–332, PMID 10706664.
- K. R. Sowers, J. E. Boone, R. P. Gunsalus: Disaggregation of Methanosarcina spp. and Growth as Single Cells at Elevated Osmolarity. In: Applied and Environmental Microbiology. Band 59, Nr. 11, November 1993, ISSN 0099-2240, S. 3832–3839, PMID 16349092, PMC 182538 (freier Volltext).
- G. M. Maestrojuan, J. E. Boone, R. A. Mah, J. A. G. F. Menaia, M. S. Sachs: Taxonomy and Halotolerance of Mesophilic Methanosarcina Strains, Assignment of Strains to Species, and Synonymy of Methanosarcina mazei and Methanosarcina frisia. In: International Journal of Systematic Bacteriology. Band 42, Nr. 4, 1. Oktober 1992, ISSN 0020-7713, S. 561–567, doi:10.1099/00207713-42-4-561.
- K. R. Sowers, J. E. Boone, R. P. Gunsalus: Disaggregation of Methanosarcina spp. and Growth as Single Cells at Elevated Osmolarity. In: Applied and Environmental Microbiology. Band 59, Nummer 11, November 1993, S. 3832–3839, PMID 16349092, PMC 182538 (freier Volltext).
- O. Kandler, H. Hippe: Lack of peptidoglycan in the cell walls of Methanosarcina barkeri. In: Archives of microbiology. Band 113, Nummer 1–2, Mai 1977, S. 57–60, PMID 889387.
- A. Klingl: S-layer and cytoplasmic membrane - exceptions from the typical archaeal cell wall with a focus on double membranes. In: Frontiers in Microbiology. Band 5, 2014, S. 624, doi:10.3389/fmicb.2014.00624, PMID 25505452, PMC 4243693 (freier Volltext) (Review).
- Peter Kreisl, Otto Kandler: Chemical structure of the cell wall polymer of methanosarcina. In: Systematic and Applied Microbiology. 7, 1986, S. 293, doi:10.1016/S0723-2020(86)80022-4.
- L. Kjellén, U. Lindahl: Proteoglycans: structures and interactions. In: Annual review of biochemistry. Band 60, 1991, S. 443–475, doi:10.1146/annurev.bi.60.070191.002303, PMID 1883201 (Review).
- S. V. Albers, B. H. Meyer: The archaeal cell envelope. In: Nature reviews. Microbiology. Band 9, Nummer 6, Juni 2011, S. 414–426, doi:10.1038/nrmicro2576, PMID 21572458 (Review).
- W. E. Balch: Methanogens:reevaluation of a unique biological group. In: Microbiology and Molecular Biology Reviews. Band 43, Nr. 2, 1979, S. 260–296, PMID 390357, PMC 281474 (freier Volltext) – (asm.org [PDF]).
- J. M. O'Brien, R. H. Wolkin, T. T. Moench, J. B. Morgan, J. G. Zeikus: Association of hydrogen metabolism with unitrophic or mixotrophic growth of Methanosarcina barkeri on carbon monoxide. In: Journal of Bacteriology. Band 158, Nr. 1, April 1984, ISSN 0021-9193, S. 373–375, PMID 6715282, PMC 215429 (freier Volltext).
- A. J. Kluyver, C. G. T. P. Schnellen: On the fermentation of carbon monoxide by pure cultures of methane bacteria. In: Archives of Biochemistry. Band 14, Nr. 1-2, Juli 1947, ISSN 0096-9621, S. 57–70, PMID 20251332.
- Derek R. Lovley: The Hydrogen Economy of Methanosarcina barkeri: Life in the Fast Lane. In: Journal of Bacteriology. Band 200, Nr. 20, 15. Oktober 2018, ISSN 1098-5530, doi:10.1128/JB.00445-18, PMID 30082458, PMC 6153660 (freier Volltext).
- Regina Saum u. a.: The F1FO ATP synthase genes in Methanosarcina acetivorans are dispensable for growth and ATP synthesis. In: FEMS Microbiology Letters. Vol. 300, Issue 2, November 2009, S. 230–236. doi:10.1111/j.1574-6968.2009.01785.x
- John Atkins, Ray Gesteland: The 22nd Amino Acid. In: Science Magazine. Band 296. American Association for the Advancement of Science, 24. Mai 2002, doi:10.1126/science.1073339 (sciencemag.org [PDF; abgerufen am 1. Juni 2014]).
- C. G. T. P. Schnellen: Onderzoekingen over de methaangisting. Inauguraldissertation. Delft 1947, S. 1–137 (niederländisch).
- M. P. Bryant, D. R. Boone: Emended Description of Strain MST(DSM 800T), the Type Strain of Methanosarcina barkeri. In: International Journal of Systematic Bacteriology. 37, 1987, S. 169, doi:10.1099/00207713-37-2-169.
- Methanosarcina barkeri, Typstamm MS (DSM 800). Leibniz-Institut DSMZ-Deutsche Sammlung von Mikroorganismen und Zellkulturen GmbH, abgerufen im Mai 2019 (englisch).
- Methanosarcina barkeri, Typstamm MS (ATCC® 43569™). ATCC®, abgerufen im Mai 2019 (englisch, Weitere Bezeichnung: ATCC 51582).
- Methanosarcina barkeri, Typstamm MS (JCM 10043). RIKEN-BRC, Microbe Division (JCM - Japan Collection of Microorganisms), abgerufen im Mai 2019 (englisch).
- LPSN in Zusammenarbeit mit der Ribocon GmbH: Classification of domains and phyla - Hierarchical classification of prokaryotes (bacteria), Version 2.1. Updated 19 July 2018. In: LPSN, List of prokaryotic names with standing in nomenclature. J. P. Euzéby, Juli 2018, abgerufen im Mai 2019 (englisch).
- LPSN in Zusammenarbeit mit der Ribocon GmbH: Abruf der Gattung mit ihren Arten. In: LPSN, List of prokaryotic names with standing in nomenclature. J. P. Euzéby, abgerufen im Mai 2019 (englisch).
- A. J. Kluyver & C. B. Van Niel: Prospects for a natural system of classification of bacteria. In: Zentralblatt für Bakteriologie Parasitenkunde Infektionskrankheiten und Hygiene. Abteilung II. Band 94, 1936, S. 369–403.
- R. A. Mah, D. A. Kuhn: Rejection of the Type Species Methanosarcina methanica (Approved Lists 1980), Conservation of the Genus Methanosarcina with Methanosarcina barkeri (Approved Lists 1980) as the Type Species, and Emendation of the Genus Methanosarcina: Request for an Opinion. In: International Journal of Systematic Bacteriology. 34, 1984, S. 266, doi:10.1099/00207713-34-2-266.
- Lawrence G.Wayne: Actions of the Judicial Commission of the International Committee on Systematic Bacteriology on Requests for Opinions Published in 1983 and 1984. In: International Journal of systematic Bacteriology. vol. 36, no. 2, 1986, S. 357–358.
- D. E. Holmes, A. E. Rotaru, T. Ueki, P. M. Shrestha, J. G. Ferry, D. R. Lovley: Electron and Proton Flux for Carbon Dioxide Reduction in Methanosarcina barkeri During Direct Interspecies Electron Transfer. In: Frontiers in Microbiology. Band 9, 2018, S. 3109, doi:10.3389/fmicb.2018.03109, PMID 30631315, PMC 6315138 (freier Volltext).
- F. Enzmann, F. Mayer, M. Rother, D. Holtmann: Methanogens: biochemical background and biotechnological applications. In: AMB Express. Band 8, Nummer 1, Januar 2018, S. 1, doi:10.1186/s13568-017-0531-x, PMID 29302756, PMC 5754280 (freier Volltext) (Review).
- Sarah E. Hook, André-Denis G. Wright, Brian W. McBride: Methanogens: Methane Producers of the Rumen and Mitigation Strategies. In: Archaea. Band 2010, 2010, ISSN 1472-3646, S. 1–11, doi:10.1155/2010/945785, PMID 21253540, PMC 3021854 (freier Volltext) – (hindawi.com [abgerufen am 23. Mai 2019]).
- Alexander Aivasidis, Christian Wandrey: Entwicklung und praktische Umsetzung eines Biogas-Hochleistungsverfahrens zur Reinigung organisch stark belasteter Abwässer: Entwicklung und praktische Umsetzung eines Biogas-Hochleistungsverfahrens zur Reinigung organisch stark belasteter Abwässer. In: Chemie Ingenieur Technik. Band 61, Nr. 6, 30. Juni 1989, S. 484–487, doi:10.1002/cite.330610613 (wiley.com [abgerufen am 23. Mai 2019]).
Siehe auch
- Amelia-Elena Rotaru, Pravin Malla Shrestha, Fanghua Liu, Beatrice Markovaite, Shanshan Chen, Kelly P. Nevin, Derek R. Lovley: Direct Interspecies Electron Transfer between Geobacter metallireducens and Methanosarcina barkeri. In: Applied and Environmental Microbiology. Band 80, Nr. 15, 16. Mai 2014, S. 4599–4605, doi:10.1128/AEM.00895-14.
- Jessica Brill: Methanosarcina barkeri fusaro. In: JGI Genome Portal. The Regents of the University of California (© 1997-2019), abgerufen am 22. Mai 2019 (englisch).