Asgard-Archaeen
Die Asgard-Supergruppe, vorschlagsgemäß auch als auch Asgardarchaeota bezeichnet,[1] ist eine Klade von Archaeen[2] im vorgeschlagenes Rang eines Superphylums, zu der (u. a.) die vorgeschlagenen Phyla „Lokiarchaeota“, „Thorarchaeota“, „Odinarchaeota“ und „Heimdallarchaeota“ gehören.[3] Während die frühen Hinweise nur aus Metagenom-Daten stammten, wurde inzwischen der erste Vertreter der Gruppe kultiviert.[4] Das Asgard-Superphylum stellt die nächsten prokaryotischen Verwandten der Eukaryoten dar,[5] die möglicherweise aus einer Vorfahrenlinie der Asgardarchaeota hervorgegangen sind, nachdem sie Bakterien durch den Prozess der Symbiogenese zu Mitochondrien oder mitochondrien-ähnliche Organellen (mitochondria-like organelles, MROs) assimiliert haben.[5][6]
| Asgardarchaeota | ||||||||
|---|---|---|---|---|---|---|---|---|

Prometheoarchaeum syntrophicum, künstlerische Nachbildung in Plastilin. | ||||||||
| Systematik | ||||||||
| ||||||||
| Wissenschaftlicher Name | ||||||||
| Asgardarchaeota | ||||||||
| Zaremba-Niedzwiedzka et al. 2017 |
Forschungsgeschichte
Im Sommer 2010 wurden Sedimente aus einem Bohrkern analysiert, der im Rifttal auf dem Knipowitsch-Rücken[Anm. 1] zwischen Grönland und Spitzbergen im Arktischen Ozean entnommen wurde. Der Entnahmeort war in der Nähe des Hydrothermalschlots namens Lokis Schloss[7] (Loki's Castle 73,55° N, 8,15° O), einem sog. „Schwarzen Raucher“. Weil vorherige Untersuchungen auf neuartige Archaeen-Linien hingedeutet hatten, wurden die Proben einer Metagenomanalyse unterzogen, die diese Vermutung bestätigten.[8][9]
Nach dem positiven Ergebnis der ersten Analysen wurden die Proben von einem Team unter Führung der Universität Uppsala einer phylogenetischen Analyse unterzogen, die eine Anzahl von hochkonservierter Protein-kodierender Gene zum Gegenstand hatte. Als Ergebnis schlug das Team im Jahr 2015 das neue Archaeenphylum „Lokiarchaeota“ für die aus der Metagenomik identifizierten Gensequenzen (Contigs) vor.[10]
Der Name ist ein Verweis auf den Schwarzen Raucher, von dem die erste Metagenomprobe stammte, und bezieht sich der Name auf Loki, einer der vielschichtigsten und wandlungsfähigsten Gestalten des nordischen Pantheons.[11] Der mythologische Loki wurde beschrieben als „eine atemberaubend komplexe, verwirrende und ambivalente Figur, die Ursache unzähliger ungelöster wissenschaftlicher Kontroversen war“,[12] ganz analog zur Rolle der Lokiarchaeota in den Debatten über den Ursprung der Eukaryoten.[10][13]
Im Jahr 2016 entdeckte ein anderes Team unter Leitung der University of Texas in Proben aus Sedimenten im Mündungsgebiet (Ästuarsedimenten) des White Oak River (34,8835° N, 77,2216° W) in North Carolina eine weitere, verwandte Gruppe von Archaeen, die Thorarchaeota benannt wurde nach Thor, einem weiteren nordischen Gott.[14]
Weitere Proben von Lokis Schloss, dem Yellowstone-Nationalpark, der Aarhus-Bucht, einem Grundwasserleiter (Aquifer) in der Nähe des Colorado River, dem Radiata Pool in Neuseeland (Ngatamariki, bei der Stadt Taupo und dem gleichnamigen Supervulkan, Nordinsel),[15][16] Hydrothermalquellen in der Nähe der Taketomi-Insel, Japan, und der Mündung des White Oak River in den Vereinigten Staaten führten dazu, dass weitere verwandte Gruppen entdeckt wurden, Odinarchaeota und Heimdallarchaeota,[3] und entsprechend der Namenskonvention nach Odin bzw. Heimdall benannt wurden. Das Superphylum, das diese Mikroben enthält, bekam dann konsequenterweise den Namen „Asgard“, nach dem Wohnort der Götter in der nordischen Mythologie.[3]
Im Jahr 2021 erweiterte die vergleichende Analyse von 162 Asgard-Genomen die phylogenetische Vielfalt dieser Supergruppe erheblich und führte zum Vorschlag von sechs zusätzlichen Phyla, einschließlich einer basalen Klade, die vorläufig Wukongarchaeota genannt wird. In mehreren dieser Phyla wurden weitere Homologe von Proteinen entdeckt, die für Eukaryoten charakteristisch sind. Die deutet auf eine dynamische Evolution durch horizontalen Gentransfer, Genverlust und -verdopplung und sogar Cross-Domain-Shuffling hin. Die Studie erlaubt jedoch noch nicht, zwischen den beiden möglichen Positionen des letzten gemeinsamen Vorfahren der Eukaryoten (LECA) zu entscheiden: entweder einer Schwesterklade der Heimdallarchaeota-Wukongarchaeota-Klade innerhalb von Asgard (wie im Kladogramm unten) oder einer Schwesterklade von Asgard selbst (innerhalb der Archaea).[17]
Beschreibung
Die Asgard-Mitglieder kodieren eine Vielzahl eukaryotischer Signaturproteine (ESPs),[18] darunter neuartige GTPasen, membranumbauende Proteine (en. membrane-remodelling proteins, wie ESCRT und SNF7), ein Ubiquitin-Modifizierungssystem und N-Glykosylierungspfad-Homologe.[3]
Asgard-Archaeen haben ein reguliertes Aktin-Zytoskelett, und die von ihnen verwendeten Profiline und Gelsoline können mit eukaryotischen Aktinen interagieren.[19][20][21] Sie scheinen auch Vesikel zu bilden, wie unter dem Kryoelektronenmikroskopie (Kryo-EM) zu erkennen ist. Einige scheinen S-Layer-Proteine mit einer PKD-Domäne (en. polycystic kidney disease domain) zu haben.[4] Außerdem haben sie wie Eukaryoten in der größten Untereinheit der ribosomalen RNA, der LSU-rRNA (large subunit of ribosomal RNA) eine dreifache Erweiterung ES39 (expansion segment 39)[22][23]
Die Vielfalt an CRISPR-Cas-verwandten Systemen ist ein spezielles Merkmal der Asgard-Archaeen, das bei Eukaryoten nicht vorkommt.[24]
Stoffwechsel
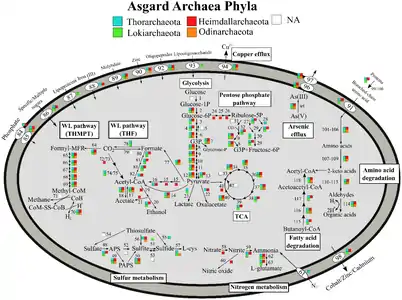 Stoffwechselwege der Asgard-Archaeen für einige der Phyla.[25]
Stoffwechselwege der Asgard-Archaeen für einige der Phyla.[25]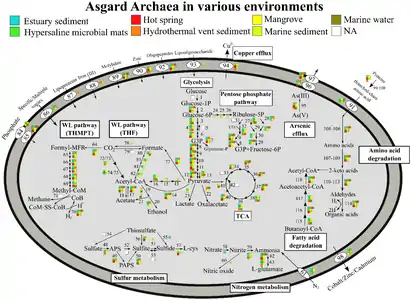 Stoffwechselwege von Asgard-Archaeen, je nach Umgebung.[25]
Stoffwechselwege von Asgard-Archaeen, je nach Umgebung.[25]
Asgard-Archaeen sind obligate Anaerobier. Sie haben einen Wood-Ljungdahl-Weg und führen Glykolyse durch. Die Mitglieder dieser Gruppe können autotroph, heterotroph oder phototroph mit Heliorhodopsin sein.[25] Ein Mitglied, Candidatus Prometheoarchaeum syntrophicum, führt Syntrophie mit einem schwefelreduzierenden Proteobakterium und einem methanogenen Archaeon durch.[4]
Ihre RuBisCO-Versionen sind nicht kohlenstofffixierend, sondern werden wahrscheinlich für das Nukleosid-Salvage (en. nucleoside salvaging) verwendet.[25]
Ähnlichkeiten mit Eukaryoten in Untergruppen
Im Jahr 2017 wurde entdeckt, dass die Vertreter des vorgeschlagenen Phylums Heimdallarchaeota N-terminale Core-Histon-Arme (corehistone tails) haben, ein Merkmal, von dem man zuvor annahm, dass es ausschließlich bei Eukaryoten vorkommt. Bei zwei weiteren Archaeen-Phyla, die nicht Asgard angehören, wurde 2018 ebenfalls dieses Merkmal gefunden, und zwar in „Huberarchaeota“ (DPANN) und „Bathyarchaeota“ (TACK).[26]
Im Januar 2020 fanden Wissenschaftler Candidatus Prometheoarchaeum syntrophicum, ein Mitglied der Lokiarchaeota, der eine Syntrophie mit zwei Bakterienarten eingeht. Dieser Befund zeigt, dass Asgard-Archaeen zu komplexen Syntrophien in der Lage sind, eine Voraussetzung für die von der Eozyten-Hypothese behauptete Entwicklung von den Archaeen hin zu komplexen eukaryotischen Mikroorganismen, wie sie vor etwa zwei Milliarden Jahren durch Symbiogenese entstanden sind.[27][4][Anm. 2]
Systematik
Die phylogenetischen Beziehungen innerhalb der Asgard-Supergruppe sind noch in der Diskussion.
Die „Heimdallarchaeota“ (und ggf. ihnen nahestehende Gruppen) gelten als die am tiefsten verzweigten Asgard-Archaea.[4] Die Eukaryoten können Schwesterklade Asgard-Archae als ganzes oder der Heimdallarchaeota bzw. der nahe verwandten Idunnarchaeota sein.[28][29] Ein bevorzugtes Szenario ist die Syntrophie, bei der ein Organismus auf die Ernährung des anderen angewiesen ist. In diesem Fall könnte die Syntrophie darauf zurückzuführen sein, dass die Asgard-Archaea in eine unbekannte Bakterienart inkorporiert wurden und sich zum Zellkern entwickelten. Ein α-Proteobakterium wurde inkorporiert, und entwickelte sich so zum Mitochondrium zu werden.[28]
Die verwandtschaftlichen Verhältnisse der Mitglieder sind ungefähr wie folgt:[5][6][17][29]
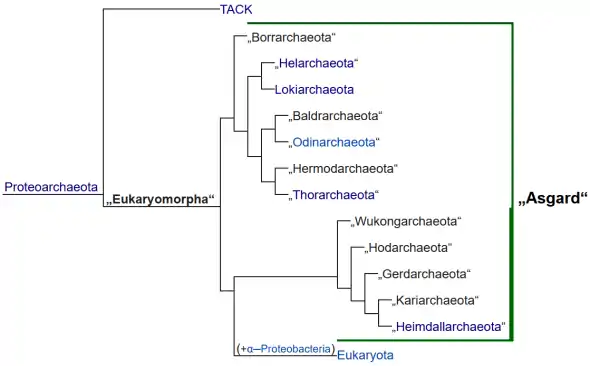
Die Asgard-Archaeen zerfallen danach in zwei Kladen (um die „Lokiarchaeota“ und um die „Heimdallarchaeota“). Nach Caceres (2019) sollten sich noch die Idunnarchaeota zur Klade aus Heimdall- und Kariarchaeota hinzugesellen.
Namensherkunft:
- „Baldrarchaeota“ Liu et al. 2020[17][30][29] ⇒ Balder
- „Borrarchaeota“ Liu et al. 2020[17][31] ⇒ Börr
- „Gerdarchaeota“ Cai et al. 2020[32] ⇒ Gerda
- „Heimdallarchaeota“ Zaremba-Niedzwiedzka et al. 2017[33][34] ⇒ Heimdall
- „Helarchaeota“ Seitz et al. 2019[35][36][37] ⇒ Hel
- „Hermodarchaeota“ Liu et al. 2020[17][38] ⇒ Hermodr
- „Hodarchaeota“ Liu et al. 2020[17][39] ⇒ Hödur
- „Kariarchaeota“ Liu et al. 2020[17][40] ⇒ Kari
- „Lokiarchaeota“ Spang et al. 2015[41][42] ⇒ Loki
- „Odinarchaeota“ Zaremba-Niedzwiedzka et al. 2017[43][44] ⇒ Odin
- „Thorarchaeota“ Seitz et al. 2016[14][45][46] ⇒ Thor
- „Wukongarchaeota“ Liu et al. 2020[17][47] ⇒ Sūn Wùkōng (chinesisch 孫悟空 / 孙悟空, W.-G. Sun Wu-k'ung)
Weitere vorgeschlagene Mitgiedsphyla:
- „Freyarchaeota“ Caceres 2019[48][29]
- „Gefionarchaeota“ Caceres 2019[29]
- „Friggarchaeota“ Caceres 2019[29]
- „Idunnarchaeota“ Caceres 2019[49] (Schwestergruppe der Heimdallarchaeota und möglicherweise die nächsten Verwandten der Eukaryoten)[29]
- „Sifarchaeota“ Farag et al. 2021[50][51] ⇒ Sif
- „Uncultured Archaeal Phylum 3 (UAP3/AAG)“ Parks et al. 2017,[52][53] mit
Nach Farag et al. (2021) scheinen die „Sifarchaeota“ am nächsten mit den „Thorarchaeota“ verwandt zu sein. Die „Thorarchaeota“ sind bei diesen Autoren (mit den „Sifarchaeota“) aber im Asgard-Zweig der „Heimdallarchaeota“ angesiedelt, und nicht bei den Lokiarchaeota.[50]
In beiden Fällen lässt sich die Asgard-Supergruppe (so wie im Kladogramm) im engeren Sinn auffassen, dann ist diese möglicherweise paraphyletisch, da die Eukaryoten von ihrem letzten gemeinsamen Vorfahren (last common ancestor, LCA) (und den α─Proteobacteria) abstammen. Alternativ Fasst man diese Gruppen im weiteren Sinn auf, dann sind die Eukaryoten ebenfalls (sehr weit entwickelte) Asgard-Mitglieder.
- „Eukaryomorpha“ Fournier & Poole 2018[58]
Diese Bezeichnung umfasst explizit alle Asgard-Archaeen zusammen mit den Eukaryoten.
Die Problematik setzt sich unter dieser Annahme in den höheren taxonomischen Rängen fort, bis hin zur Domäne. Man kann die Bezeichnung Archaea (Archaeen) im weiten Sinn verstehen (inklusive Eukaryoten), dann wären diese monophyletisch. Es gäbe nur zwei Domänen: Neben den so erweiterten Archaeen nur noch die Bacteria (Bakterien). Oder man versteht den Begriff Archaeen im engen Sinn und behält den bisherigen Sprachgebrauch bei, dann ist diese Gruppe paraphyletisch. Als taxonomischer Oberbegriff, der Archaeen und Eukaryoten umfasst, wurde 2020 von Cavalier-Smith und Chao die Bezeichnung „Neomura“ vorgeschlagen, dies wäre dann eine Schwestergruppe der Bacteria.[59][Anm. 3]
Alternative Bezeichnungsschemata
Der taxonomische Rang der Asgard-Klade und ihrer Teilgruppen ist derzeit (2019/2021) noch in Diskussion.[29] Je nach Rang tragen die bezeichneten Taxa dann Namen mit je nach Autor unterschiedlichen Endungen. Ein Beispiel die Synonyme:
- „Asgardarchaeota“ Violette Da Cunha et al. 2017
- „Asgardaeota“ Whitman 2018
Sun et al. (2021) sehen die oben bezeichneten Phyla eher als Klassen, was sich in der Endung „-archaia“ statt „-archaeota“ niederschlägt. So sind zum Beispiel Synonyme:
- „Sipharchaeia“
- „Sipharchaeota“
In dieser Notation würde der Stammbaum der Eukaryomorpha etwa so aussehen:[60]
| „Eukaryomorpha“ |
| ||||||||||||||||||||||||||||||||||||
Dieser Baum führt eine weitere von diesen Autoren vorgeschlagene Asgard-Subklade als Klasse auf:
- „Jordarchaeia“ Sun et al. 2021[61]
Anmerkungen
- benannt nach Nikolai Michailowitsch Knipowitsch
- Die Eozyten-Hypothese war in den 1980er Jahren – vor Entdeckung der ersten Asgard-Archaeen – aufgrund gewisser Übereinstimmungen der Crenarchaeota mit den Eukaryoten entwickelt worden. Diese gehören wir die Asgard-Archaeen den Proteoarchaeota an; „Eozyten“ ist eine veraltete Bezeichnung für die Crenarchaeota bzw. sensu lato ein Synonym für die Proteoarchaeota.
- Der Begriff „Neomura“ wurde von Cavalier-Smith bereits 2002 vorgeschlagen, damals im Rahmen eines zur Eozyten-Hypothese alternativen Szenarios. Inzwischen räumt der Autor aufgrund der neuen Ergebnisse aber auch die Möglichkeit einer Abstammung der Eukaryoten aus einer Gruppe der Archaeen ein. Die Bezeichnung „Neomura“ als Oberbegriff für Archaeen s. s. und Bakterien bleibt davon aber weitgehend unberührt. Referenz: Neomura Thomas Cavalier-Smith (2002): The phagotrophic origin of eukaryotes and phylogenetic classification of Protozoa, in: Int. J. Syst. Evol. Microbiol. Band 52, Nr. Pt2, März 2002, S. 297–354, doi:10.1099/00207713-52-2-297, PMID 11931142.
Einzelnachweise
- Violette Da Cunha, Morgan Gaia, Daniele Gadelle, Arshan Nasir, Patrick Forterre: Lokiarchaea are close relatives of Euryarchaeota, not bridging the gap between prokaryotes and eukaryotes. In: PLoS Genet., Band 13, Nr. 6, 12. Juni 2017, e1006810, doi:10.1371/journal.pgen.1006810.
- NCBI: Asgard group (clade); graphisch: Asgard group, auf: Lifemap, NCBI Version.
- Katarzyna Zaremba-Niedzwiedzka, Eva F. Caceres, Jimmy H. Saw, Disa Bäckström, Lina Juzokaite, Emmelien Vancaester, Kiley W. Seitz, Karthik Anantharaman, Piotr Starnawski: Asgard archaea illuminate the origin of eukaryotic cellular complexity. In: Nature. 541, Nr. 7637, 11. Januar 2017, ISSN 1476-4687, S. 353–358. bibcode:2017Natur.541..353Z. doi:10.1038/nature21031. PMID 28077874. PDF
- Hiroyuki Imachi, Masaru K. Nobu, Nozomi Nakahara, Yuki Morono, Miyuki Ogawara, Yoshihiro Takaki, Yoshinori Takano, Katsuyuki Uematsu, Tetsuro Ikuta, Motoo Ito, Yohei Matsui: Isolation of an archaeon at the prokaryote–eukaryote interface. In: Nature. 577, Nr. 7791, 23. Januar 2020, ISSN 1476-4687, S. 519–525. bibcode:2020Natur.577..519I. doi:10.1038/s41586-019-1916-6. PMID 31942073. PMC 7015854 (freier Volltext).
- Laura Eme, Anja Spang, Jonathan Lombard, Courtney W. Stairs, Thijs J. G. Ettema: Archaea and the origin of eukaryotes. In: Nature Reviews Microbiology. 15, Nr. 12, 10. November 2017, ISSN 1740-1534, S. 711–723. doi:10.1038/nrmicro.2017.133. PMID 29123225.
- Tom A. Williams, Cymon J. Cox, Peter G. Foster, Gergely J. Szöllősi, T. Martin Embley: Phylogenomics provides robust support for a two-domains tree of life. In: Nature Ecology & Evolution. 4, Nr. 1, 9. Dezember 2019, ISSN 2397-334X, S. 138–147. doi:10.1038/s41559-019-1040-x. PMID 31819234. PMC 6942926 (freier Volltext).
- Scientists Break Record By Finding Northernmost Hydrothermal Vent Field. 24. Juli 2008. Abgerufen am 30. April 2021. Auf: ScienceDaily
- Steffen Leth Jørgensen, Bjarte Hannisdal, Anders Lanzen, Tamara Baumberger, Kristin Flesland, Rita Fonseca, Lise Øvreås, Ida H. Steen, Ingunn H. Thorseth, Rolf B. Pedersen, Christa Schleper: Correlating microbial community profiles with geochemical data in highly stratified sediments from the Arctic Mid-Ocean Ridge. In: PNAS. 109, Nr. 42, 5. September 2012, S. E2846–55. doi:10.1073/pnas.1207574109. PMID 23027979. PMC 3479504 (freier Volltext).
- Steffen Leth Jørgensen, Ingunn H. Thorseth, Rolf B. Pedersen, Tamara Baumberger, Christa Schleper: Quantitative and phylogenetic study of the Deep Sea Archaeal Group in sediments of the Arctic mid-ocean spreading ridge. In: Frontiers in Microbiology. 4, 4. Oktober 2013, S. 299. doi:10.3389/fmicb.2013.00299. PMID 24109477. PMC 3790079 (freier Volltext).
- Anja Spang, Jimmy H. Saw, Steffen L. Jørgensen, Katarzyna Zaremba-Niedzwiedzka, Joran Martijn, Anders E. Lind, Roel van Eijk, Christa Schleper, Lionel Guy, Thijs J. G. Ettema: Complex archaea that bridge the gap between prokaryotes and eukaryotes. In: Nature. 521, Nr. 7551, Mai 2015, ISSN 1476-4687, S. 173–179. bibcode:2015Natur.521..173S. doi:10.1038/nature14447. PMID 25945739. PMC 4444528 (freier Volltext).
- Ed Yong: Break in the Search for the Origin of Complex Life (en-US). In: The Atlantic. Abgerufen am 30. April 2021.
- Stefanie von Schnurbein: The Function of Loki in Snorri Sturluson's "Edda". In: History of Religions. 40, Nr. 2, Oktober 2000, S. 109–124. doi:10.1086/463618.
- Anja Spang, Laura Eme, Jimmy H. Saw, Eva F. Caceres, Katarzyna Zaremba-Niedzwiedzka, Jonathan Lombard, Lionel Guy, Thijs J. G. Ettema, Antonis Rokas: Asgard archaea are the closest prokaryotic relatives of eukaryotes. In: PLOS Genetics. 14, Nr. 3, 18. März 2018, S. e1007080. doi:10.1371/journal.pgen.1007080. PMID 29596421. PMC 5875740 (freier Volltext).
- Kiley W. Seitz, Cassandre S. Lazar, Kai-Uwe Hinrichs, Andreas P. Teske, Brett J. Baker: Genomic reconstruction of a novel, deeply branched sediment archaeal phylum with pathways for acetogenesis and sulfur reduction. In: The ISME Journal. 10, Nr. 7, 29. Januar 2016, ISSN 1751-7370, S. 1696–1705. doi:10.1038/ismej.2015.233. PMID 26824177. PMC 4918440 (freier Volltext).
- Matthew Stott, Craig Cary, Melissa Climo: The 1000 Project – A microbial inventory of geothermal ecosystems, New Zealand Geoteral Workshop, 22. November 2012; Radiata Pool
- Nukki Macdonald: What lives in New Zealand's hot and hostile volcanic springs?, auf: stuff vom 7. Mai 2015
- Yang Liu, Kira S. Makarova, Wen-Cong Huang, Yuri I. Wolf, Anastasia Nikolskaya, Xinxu Zhang, Mingwei Cai, Cui-Jing Zhang, Wei Xu, Zhuhua Luo, Lei Cheng, Eugene V. Koonin, Meng Li: Expanded diversity of Asgard archaea and their relationships with eukaryotes, in: Nature, 28. April 2021, doi:10.1038/s41586-021-03494-3.
Preprint: Expanding diversity of Asgard archaea and the elusive ancestry of eukaryotes, in: bioRχiv, 20. Oktober 2020, doi:10.1101/2020.10.19.343400 - eukaryotic signature proteins (ESPs), ein Satz von Proteinen, die Verwandtschaft zu Eukaryoten anzeigen kann.
- Caner Akıl, Robert C. Robinson: Genomes of Asgard archaea encode profilins that regulate actin. In: Nature. 562, Nr. 7727, 3. Oktober 2018, S. 439–443. bibcode:2018Natur.562..439A. doi:10.1038/s41586-018-0548-6. PMID 30283132.
- Caner Akıl, Linh T. Tran, Magali Orhant-Prioux, Yohendran Baskaran, Edward Manser, Laurent Blanchoin, Robert C. Robinson: Complex eukaryotic-like actin regulation systems from Asgard archaea, bioRxiv 10.1101/768580, 14. September 2019, doi:10.1101/768580
- Caner Akıl, Linh T. Tran, Magali Orhant-Prioux, Yohendran Baskaran, Edward Manser, Laurent Blanchoin, Robert C. Robinson: Insights into the evolution of regulated actin dynamics via characterization of primitive gelsolin/cofilin proteins from Asgard archaea. In: PNAS. 117, Nr. 33, 18. August 2020, S. 19904–19913. doi:10.1073/pnas.2009167117. PMID 32747565.
- Odd Nygård, Gunnar Alkemar, Sofia L. Larsson: Analysis of the secondary structure of expansion segment 39 in ribosomes from fungi, plants and mammals, in: J Mol Biol, Band 357, Nr. 3, 31. März 2006, S. 904–916, doi:10.1016/j.jmb.2006.01.043, PMID 16473366, Epub 30. Januar 2006
- Petar I Penev, Sara Fakhretaha-Aval, Vaishnavi J. Patel, Jamie J. Cannone, Robin R. Gutell, Anton S. Petrov, Loren Dean Williams, Jennifer B. Glass: Supersized Ribosomal RNA Expansion Segments in Asgard Archaea. In: Genome Biology and Evolution. 12, Nr. 10, 1. Oktober 2020, S. 1694–1710. doi:10.1093/gbe/evaa170.
- Kira S. Makarova, Yuri I. Wolf, Sergey A. Shmakov, Yang Liu, Meng Li, Eugene V. Koonin: Unprecedented Diversity of Unique CRISPR-Cas-Related Systems and Cas1 Homologs in Asgard Archaea, in: The CRISPR Journal Band 3, Nr. 3, 1. Juni 2020, S. 156–163, doi:10.1089/crispr.2020.0012, ISSN 2573-1599, PMC 7307682 (freier Volltext)
- Fraser MacLeod, Gareth S. Kindler, Hon Lun Wong, Ray Chen, Brendan P. Burns: Asgard archaea: Diversity, function, and evolutionary implications in a range of microbiomes. In: AIMS Microbiology. 5, Nr. 1, 2019, ISSN 2471-1888, S. 48–61. doi:10.3934/microbiol.2019.1.48. PMID 31384702. PMC 6646929 (freier Volltext).
- B. Henneman, C. van Emmerik, H. van Ingen, R. T. Dame: Structure and function of archaeal histones. In: PLOS Genetics. 14, Nr. 9, September 2018, S. e1007582. bibcode:2018BpJ...114..446H. doi:10.1371/journal.pgen.1007582. PMID 30212449. PMC 6136690 (freier Volltext).
- Carl Zimmer: This Strange Microbe May Mark One of Life's Great Leaps - A organism living in ocean muck offers clues to the origins of the complex cells of all animals and plants.. In: The New York Times, 15. Januar 2020. Abgerufen im 2020-01-2020.
- Purificación López-García, David Moreira: Eukaryogenesis, a syntrophy affair. In: Nature Microbiology. 4, Nr. 7, 1. Juli 2019, ISSN 2058-5276, S. 1068–1070. doi:10.1038/s41564-019-0495-5. PMID 31222170. PMC 6684364 (freier Volltext).
- Eva F. Caceres: Genomic and evolutionary exploration of Asgard archaea, Doctoral thesis, Uppsala University, Disciplinary Domain of Science and Technology, Biology, Department of Cell and Molecular Biology, 12. November 2019
- NCBI: Candidatus Baldrarchaeota (phylum)
- NCBI: Candidatus Borrarchaeota (phylum)
- NCBI: Candidatus Gerdarchaeota (phylum)
- LPSN: Phylum "Candidatus Heimdallarchaeota"
- NCBI: Candidatus Heimdallarchaeota (phylum)
- Kiley W. Seitz, Nina Dombrowski, Laura Eme, Anja Spang, Jonathan Lombard, Jessica R. Sieber, Andreas P. Teske, Thijs J. G. Ettema, Brett J. Baker: Asgard archaea capable of anaerobic hydrocarbon cycling, in: Nature Communications, 23. April 2019, doi:10.1038/s41467-019-09364-x
- NCBI: Candidatus Helarchaeota (phylum)
- LPSN: Phylum "Candidatus Helarchaeota"
- NCBI: Candidatus Hermodarchaeota (phylum)
- NCBI: Candidatus Hodarchaeota (phylum)
- NCBI: Candidatus Kariarchaeota (phylum)
- LPSN: Phylum "Candidatus Lokiarchaeota"
- NCBI: Candidatus Lokiarchaeota (phylum)
- LPSN: Phylum "Candidatus Odinarchaeota"
- NCBI: Candidatus Odinarchaeota (phylum)
- LPSN: Phylum "Candidatus Thorarchaeota"
- NCBI: Candidatus Thorarchaeota (phylum)
- NCBI: Candidatus Wukongarchaeota (phylum)
- NCBI: "Candidatus Freyarchaeota" Caceres 2019 (phylum)
- NCBI: "Candidatus Idunnarchaeota" Caceres 2019 (phylum)
- Ibrahim F. Farag, Rui Zhao, Jennifer F. Biddle: “Sifarchaeota,” a Novel Asgard Phylum from Costa Rican Sediment Capable of Polysaccharide Degradation and Anaerobic Methylotrophy, in: ASM Appl Environ Microbiol 87:e02584-20, Epub 13. April 2021, doi:10.1128/AEM.02584-20, PMID 33608286.
Preprint: “Sifarchaeota” a novel Asgard phylum capable of polysaccharide degradation and anaerobic methylotrophy, auf: CSH bioRχiv vom 15. Oktober 2020, doi:10.1101/2020.10.14.339440, ResearchGate, PDF (14. Oktober 2020). Siehe insbes. Fig. 1 (Kladogramm) - NCBI: "Candidatus Sifarchaeota" Farag et al. 2021 (phylum)
- DRA: Sample Candidatus UAP3 archaeon UBA460
- NCBI: https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=1917434 archaeon UBA460 (species)
- Alla L. Lapidus, Anton I. Korobeynikov: Metagenomic Data Assembly – The Way of Decoding Unknown Microorganisms, in: Front. Microbiol., Band 12, 23. März 2021, Seiten: 653, doi:10.3389/fmicb.2021.613791, ISSN 1664-302X
- Nina Dombrowski, Tom A. Williams, Jiarui Sun, Benjamin J. Woodcroft, Jun-Hoe Lee, Bui Quang Minh, Christian Rinke, Anja Spang: Undinarchaeota illuminate DPANN phylogeny and the impact of gene transfer on archaeal evolution, in: Nature Communications, Band 11, Nr. 3939, 7. August 2020, doi:10.1038/s41467-020-17408-w
- Donovan H. Parks, Christian Rinke, Maria Chuvochina, Pierre-Alain Chaumeil, Ben J. Woodcroft, Paul N. Evans, Philip Hugenholtz, Gene W. Tyson: Recovery of nearly 8,000 metagenome-assembled genomes substantially expands the tree of life, in: Nature Microbiology, Band 2, S. 1533–1542, 11. September 2017, doi:10.1038/s41564-017-0012-7, mit Korrektur vom 12. Dezember 2017, doi:10.1038/s41564-017-0083-5
- Taxonoicon: Paraphyletic taxon: Candidate superphylum Asgard
- G. P. Fournier, A. M. Poole: A Briefly Argued Case That Asgard Archaea Are Part of the Eukaryote Tree. In: Front. Microbiol.. 9, 2018, S. 1896. doi:10.3389/fmicb.2018.01896. PMID 30158917. PMC 6104171 (freier Volltext).
- Thomas Cavalier-Smith & Ema E-Yung Chao: Multidomain ribosomal protein trees and the planctobacterial origin of neomura (eukaryotes, archaebacteria), in: Protoplasma Band 257, S. 621–753, 3. Januar 2020, doi:10.1007/s00709-019-01442-7
- Jiarui Sun, Paul N. Evans, Emma J. Gagen, Ben J. Woodcroft, Brian P. Hedlund, Tanja Woyke, Philip Hugenholtz: Recoding of stop codons expands the metabolic potential of two novel Asgardarchaeota lineages, in: Nature, ISME Commun. Band 1, Nr. 30, 28. Juni 2021, doi:10.1038/s43705-021-00032-0
- NCBI: "Candidatus Jordarchaeia" Sun et al. 2021 (class)
Weblinks
- Traci Watson: The trickster microbes that are shaking up the tree of life, in: Nature, 14. Mai 2019
- NCBI: Asgard group (clade)