Kryoelektronenmikroskopie
Die Kryoelektronenmikroskopie (Kryo-EM) ist eine Form der Transmissionselektronenmikroskopie (TEM), bei welcher biologische Proben bei kryogenen Temperaturen (≲ −150 °C) untersucht werden.
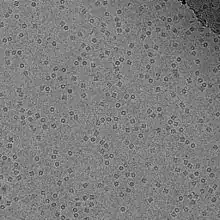
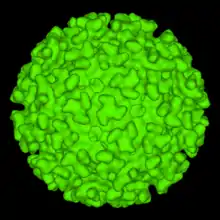
Prinzip
Bei der herkömmlichen Elektronenmikroskopie einer biologischen Probe bei Raumtemperatur muss vor der Untersuchung das Wasser aus der Probe entfernt werden. Dabei muss die Probe einen langwierigen und fehleranfälligen Prozess durchlaufen, bei welchem sie mit verschiedenen Probenvorbereitungen behandelt und das in ihr enthaltene Wasser sukzessive durch ein Kunststoff-Einbettmedium ersetzt wird. Im Rahmen dieser Behandlung können sich in der Probe Artefakte (Strukturveränderungen) durch die Behandlung bilden, welche letztendlich das Ergebnis verfälschen oder zu Fehlinterpretationen führen können.
Die Kryoelektronenmikroskopie ermöglicht eine Abbildung des untersuchten Objektes nahe am nativen Zustand. Daher kann im Vergleich zur klassischen TEM auf die Verwendung von Fixierungs- und Kontrastmitteln verzichtet werden.[1] Ohne die Kontrastmittel sind biologische Proben aufgrund der gleichmäßigen, niedrigen Elektronendichte für die verwendeten Strahlen fast transparent.[1] Aufgrund des hohen Anteils an Eis kann zudem nur eine kurz dosierte Strahlungsintensität (unter 1 bis 10 Elektronen pro Å2) verwendet werden.[1] Dadurch ist das Signal-Rausch-Verhältnis vergleichsweise gering.[1] Durch Berechnung der Strukturen aus vielen tausend Aufnahmen desselben Objekts aus unterschiedlichen Perspektiven kann ein 3D-Modell des Objekts erstellt werden (single particle method „Einzelpartikelmethode“).[1] Durch Mittelung mehrerer 3D-Modelle des gleichen Objekts können weitere Verbesserungen erzielt werden (subtomogram averaging „Untertomogrammmittelung“).[1] Die Auflösung der Kryoelektronenmikroskopie liegt normalerweise zwischen der Transmissionselektronenmikroskopie und der Röntgen-Strukturanalyse.[1] In einigen Fällen können Auflösungen um die 0,2 nm (2 Å) erreicht werden.[2] Ebenfalls ist es möglich von größeren Strukturen dreidimensionale Aufnahmen mittels der Kryoelektronentomographie zu machen.
Das Kryoelektronenmikroskop
Kryoelektronenmikroskope sind besonders angepasste Transmissionselektronenmikroskope. Die meisten Transmissionselektronenmikroskope sind mit einer Kühlfalle ausgestattet. Diese schützt die Probe während der Exposition gegenüber dem Elektronenstrahl vor Kontaminationen und sorgt für eine Verbesserung des Feinvakuums im Bereich der Probe. Die Kombination einer extrem leistungsfähigen Kühlfalle (im Besonderen sogenannte Kryoboxen) mit einem gekühlten Probenhalter, der einen wärmeleitfähig verbundenen Tank für flüssigen Stickstoff besitzt, ergibt ein Kryoelektronenmikroskop. Durch die vermehrte Verwendung der Forschungsmethode seit 2005 bauen die führenden Hersteller von Transmissionselektronenmikroskopen auch dedizierte Kryoelektronenmikroskope. Durch die Verwendung einer direkten CMOS-Aufnahme können trotz der geringen Strahlungsintensität noch Bilder aufgenommen werden. Der Kontrast der Aufnahme kann durch die Verwendung von Phasenplatten und Energiefiltern verstärkt werden.[1]
Methode
Probenvorbereitung
In der Kryoelektronenmikroskopie wird die Probe ultraschnell schockgefroren bzw. präziser formuliert: vitrifiziert, also in einen amorphen, glasartigen Zustand versetzt. Es können typischerweise Kühlraten von >10.000 K/s erreicht werden. Vorhandenes Wasser erstarrt zu amorphem Eis. Insbesondere werden Kryoelektronenmikroskope verwendet, um komplexe Proteinstrukturen zu analysieren. Dabei werden die Strukturen innerhalb von Sekundenbruchteilen durch tiefkalte Flüssigkeiten wie flüssigen Stickstoff, flüssiges Helium oder bevorzugt mit flüssigem Ethan[4] auf Temperaturen unter −150 °C (123 K) gekühlt.
Datenanalyse
Über eine im Gerät verbaute Verstelleinrichtung wird das Präparat aus verschiedenen Winkeln analysiert.
Datenverarbeitung
Aus den erhaltenen Daten kann mit Hilfe von Computerprogrammen eine dreidimensionale Elektronendichte errechnet werden. Die grundlegenden Programme wurden durch Joachim Frank maßgeblich mitentwickelt. In weiteren Schritten können dann atomare 3-D-Modelle der entsprechenden Biomoleküle in die Elektronendichte eingebaut werden, ähnlich wie bei der Röntgenstrukturanalyse.
Die Auflösung kryoelektronenmikroskopisch ermittelter 3-D-Strukturen kann derzeit 0,2 Nanometer (2 Å) erreichen. Die höchste Auflösung wurde bislang für eine Glutamatdehydrogenase (GDH) mit 1,8 Å erhalten. Im Gegensatz zur Röntgenstrukturanalyse gibt es für die Kryo-EM-Methode ein unteres theoretisches Limit der Molekülgröße, das bei etwa 30 kDa liegt. Ein weiteres Problem mancher Kryo-EM-Strukturen ist die unterschiedliche Auflösung der Elektronendichte innerhalb eines Biomoleküls.[5]
Varianten
Verschiedene Techniken und Varianten zur Kryoelektronenmikroskopie wurden entwickelt, z. B. Elektronenkristallographie,[6] Einzelpartikelanalyse,[7] Kryoelektronentomographie,[8] MicroED[9] und zeitaufgelöste Kryo-EM.[10][11][12]
Geschichte
Die Kryoelektronenmikroskopie wurde von dem Schweizer Chemiker Jacques Dubochet am European Molecular Biology Laboratory entwickelt und von Joachim Frank und Richard Henderson weiterentwickelt. 2017 wurden alle drei für ihre Arbeit mit dem Nobelpreis für Chemie ausgezeichnet.[13]
Einen Vergleich unterschiedlicher elektronenmikroskopischer Arbeitstechniken mit der Kryoelektronenmikroskopie zeigt eine Arbeit zur Charakterisierung des Mycobacterium smegmatis.[14]
Literatur
- Kira Welter, Kryo-Elektronenmikroskopie: Coole Bilder in 3D Chemie in unserer Zeit Vol. 6, 2017, S. 366–368 doi:10.1002/ciuz.201770604
Einzelnachweise
- Maria Mulisch: Romeis - Mikroskopische Technik. Springer-Verlag, 2015, ISBN 978-3-642-55190-1, S. 169.
- Resolution advances in cryo-EM enable application to drug discovery. In: Curr Opin Struct Biol. Band 41, Dec 2016, S. 194–202. doi: 10.1016/j.sbi.2016.07.009.
- J. Vonck, D. N. Parcej, D. J. Mills: Structure of Alcohol Oxidase from Pichia pastoris by Cryo-Electron Microscopy. In: PloS one. Band 11, Nummer 7, 2016, S. e0159476, doi:10.1371/journal.pone.0159476, PMID 27458710, PMC 4961394 (freier Volltext).
- Jaques Dubochet et al. Electron microscopy of frozen-hydrated bacteria Journal of Bacteriology, July 1983, p. 381-390 (Seite 381) abgerufen am 3. Januar 2018
- J. Vonck & D. J. Mills: Advances in high-resolution cryo-EM of oligomeric enzymes . In: Curr Opin Struct Biol. Band 46, Oct 2017, S. 48–54. doi: 10.1016/j.sbi.2017.05.016.
- Xiaodong Zou: Electron Crystallography. OUP Oxford, 2011, ISBN 978-0-199-58020-0. S. 4.
- Grant J. Jensen: Cryo-EM Part B: 3-D Reconstruction. In: Methods in Enzymology. Band 482, Academic Press, 2010, ISBN 978-0-123-84992-2. S. 211.
- Joachim Frank: Electron Tomography. Springer Science & Business Media, 2008, ISBN 978-0-387-69008-7, S. 50
- R. A. Crowther: The Resolution Revolution: Recent Advances In cryoEM. In: Methods in Enzymology, Band 579, Academic Press, 2016, ISBN 978-0-128-05435-2, S. 369.
- Ziao Fu, Sandip Kaledhonkar, Anneli Borg, Ming Sun, Bo Chen, Robert A. Grassucci, Måns Ehrenberg, Joachim Frank: Key Intermediates in Ribosome Recycling Visualized by Time-Resolved Cryoelectron Microscopy. In: Structure. 24, Nr. 12, 2016, S. 2092–2101. doi:10.1016/j.str.2016.09.014. PMID 27818103. PMC 5143168 (freier Volltext).
- Xiangsong Feng, Ziao Fu, Sandip Kaledhonkar, Yuan Jia, Binita Shah, Amy Jin, Zheng Liu, Ming Sun, Bo Chen, Robert A. Grassucci, Yukun Ren, Hongyuan Jiang, Joachim Frank, Qiao Lin: A Fast and Effective Microfluidic Spraying-Plunging Method for High-Resolution Single-Particle Cryo-EM. In: Structure. 25, Nr. 4, 2017, S. 663–670.e3. doi:10.1016/j.str.2017.02.005. PMID 28286002. PMC 5382802 (freier Volltext).
- Bo Chen, Sandip Kaledhonkar, Ming Sun, Bingxin Shen, Zonghuan Lu, David Barnard, Toh-Ming Lu, Ruben L. Gonzalez, Joachim Frank: Structural Dynamics of Ribosome Subunit Association Studied by Mixing-Spraying Time-Resolved Cryogenic Electron Microscopy. In: Structure. 23, Nr. 6, 2015, S. 1097–105. doi:10.1016/j.str.2015.04.007. PMID 26004440. PMC 4456197 (freier Volltext).
- Informationen der Nobelstiftung zur Preisverleihung 2017 an Jacques Dubochet, Joachim Frank, Richard Henderson (englisch)
- C. K. Bleck, A. Merz, M. G. Gutierrez, P. Walther, J. Dubochet, B. Zuber, G. Griffiths: Comparison of different methods for thin section EM analysis of Mycobacterium smegmatis. In: J Microsc. Band 237, Nr. 1, Januar 2010, S. 23–38, PMID 20055916