Contig
Ein Contig (von engl. contiguous = angrenzend, zusammenhängend) ist ein Satz überlappender DNA- oder Protein-Stücke (reads), die von derselben genetischen Quelle stammen.[1] Ein solches Contig kann dazu genutzt werden, die Original-DNA-Sequenz dieser genetischen Quelle (z. B. die Sequenz eines Chromosoms) abzuleiten.
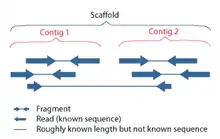
Eine Contig-Karte zeigt die relative Reihenfolge einer zusammengehörenden Contig-Bibliothek, die z. B. ein komplettes Chromosom darstellen.
MAGs
Ein damit zusammenhängender Begriff ist ein MAG (metagenome-assembled genome ‚Metagenom-assembliertes Genom‘), das ein gesamtes aus solchen Bruchstücken zusammengesetztes vermutetes (vorhergesagtes) Genom bezeichnet.[2][3]
Sequenzierung
Bei der DNA-Sequenzierung und insbesondere beim Shotgun Sequencing muss die DNA-Sequenz bzw. bei der De-Novo-Peptidsequenzierung muss die Aminosäuresequenz durch Aneinanderreihung der verschiedenen Contigs ermittelt werden.[4] Für eine Genomsequenzierung wird zur Vorbereitung oftmals die genomische DNA fragmentiert und die Bruckstücke anschließend vervielfältigt. Die einzelnen DNA-Stränge besitzen dann unterschiedliche, teilweise überlappende Sequenzen, die durch die Aneinanderreihung die vollständige Sequenz ergeben.[5][6]
Ein Contig entsteht unter der Annahme, dass die zugehörigen Reads korrekt sind. Es gibt aber bei allen bisher genutzten Sequenziermethoden Schwächen, die oft auch bekannt sind und deren Einfluss von den Herstellern zu minimieren versucht wird. Ein Contig kann auch schon aus einem einzelnen Read bestehen, aber jeder überlappende Read (am besten aus verschiedenen Richtungen oder sogar durch eine andere Sequenziermethode) erhöht die Wahrscheinlichkeit, dass hier die Wirklichkeit zu sehen ist.
Einzelnachweise
- A. Guthals, K. R. Clauser, N. Bandeira: Shotgun protein sequencing with meta-contig assembly. In: Molecular & cellular proteomics : MCP. Band 11, Nummer 10, Oktober 2012, ISSN 1535-9484, S. 1084–1096, doi:10.1074/mcp.M111.015768, PMID 22798278, PMC 3494147 (freier Volltext).
-
Ibrahim F. Farag, Rui Zhao, Jennifer F. Biddle: “Sifarchaeota,” a Novel Asgard Phylum from Costa Rican Sediment Capable of Polysaccharide Degradation and Anaerobic Methylotrophy, in: ASM Appl Environ Microbiol 87:e02584-20, Epub 13. April 2021, doi:10.1128/AEM.02584-20, PMID 33608286.
Preprint: “Sifarchaeota” a novel Asgard phylum capable of polysaccharide degradation and anaerobic methylotrophy, auf: CSH bioRxiv vom 14. Oktober 2020, doi:10.1101/2020.10.14.339440, ResearchGate. - Eva F. Caceres: Genomic and evolutionary exploration of Asgard archaea, Doctoral thesis, Uppsala University, Disciplinary Domain of Science and Technology, Biology, Department of Cell and Molecular Biology, 12. November 2019. Siehe insbesondere §Genome binning.
- Rodger Staden: A strategy of DNA sequencing employing computer programs. In: Nucleic Acids Research. 7, 1979, S. 2601–2610. PMC 327874 (freier Volltext).
- S. H. Lin, Y. C. Liao: CISA: contig integrator for sequence assembly of bacterial genomes. In: PloS one. Band 8, Nummer 3, 2013, ISSN 1932-6203, S. e60843, doi:10.1371/journal.pone.0060843, PMID 23556006, PMC 3610655 (freier Volltext).
- Z. Frenkel, E. Paux, D. Mester, C. Feuillet, A. Korol: LTC: a novel algorithm to improve the efficiency of contig assembly for physical mapping in complex genomes. In: BMC Bioinformatics. Band 11, 2010, ISSN 1471-2105, S. 584, doi:10.1186/1471-2105-11-584, PMID 21118513, PMC 3098104 (freier Volltext).