Ribosomale DNA
Als Ribosomale DNA (rDNA) werden jene Abschnitte der Desoxyribonukleinsäure (DNA) bezeichnet, die die Gene für ribosomale RNA (rRNA) enthalten. Solche Ribonukleinsäuren (RNA) sind ein funktionell wesentlicher Bestandteil der Ribosomen, an denen die Proteinbiosynthese stattfindet. Die einzelnen rRNA-Stränge entstehen jeweils durch Transkription von rDNA-Abschnitten. Es gibt verschiedene Arten von rRNA, die zentrale Funktionselemente von Ribosomen sind; sie werden nicht wie mRNA in Proteine übersetzt (translatiert).
In eukaryotischen Zellen gibt es rDNA nicht nur im Zellkern, sondern auch in den Mitochondrien, und bei Pflanzen zusätzlich in den Plastiden. Die von ihnen abgelesene rRNA wird nur in den Ribosomen dieser Organellen verwendet, nicht im Cytoplasma. Mitochondrien- und Plastiden-rDNA ähnelt von der Sequenz her der rDNA in Prokaryoten, wodurch die Endosymbiontentheorie gestützt wird.
Eukaryotische Zellen benötigen große Mengen rRNA. Daher kommt rDNA in vielen Kopien vor. Für rDNA sind tandemartig hintereinander liegende Kopien typisch, eine Form repetitiver DNA. Der Mengenanteil der rDNA am Genom ist für verschiedene Organismen unterschiedlich. Beim Menschen sind es etwa 0,4 %, auf 10 der 46 Chromosomen, bei Drosophila melanogaster etwa 17 %.
Es werden nicht in allen Zellen immer alle rDNA enthaltenden chromosomalen Abschnitte abgelesen und für die rRNA-Synthese verwendet. Die aktiven rDNA-Abschnitte sind im Zellkern auf einen oder wenige dichte Bereiche konzentriert, die Nucleoli (auch: Nukleoli; Einzahl: Nucleolus) oder Kernkörperchen. Die rDNA enthaltenden Abschnitte werden daher auch als "Nucleolus organisierende Regionen" (NORs) bezeichnet. Prokaryoten, Mitochondrien und Plastiden bilden keinen Nukleolus aus.
rDNA bei Prokaryoten
Obwohl Archaeen und Bakterien eigene Reiche bilden, ist ihre rRNA und auch ihre rDNA ähnlich organisiert. Beide Typen von Prokaryoten besitzen typischerweise nur drei verschiedene rRNAs: 16S-, 23S- und 5S-rRNA. Deren Gene liegen häufig als Cluster zusammen und werden gemeinsam transkribiert, sie bilden also ein Operon.
Bakterien
Die Anzahl der rDNA Operons bei Bakterien kann variieren, z. B. zwei bei Mollicutes, sieben bei Escherichia coli oder zehn bei Bacillus subtilis. Die Operons werden meist mit durchlaufenden Buchstaben bezeichnet (bei E. coli: rrnA bis rrnG). Oft, etwa bei E. coli enthält der rDNA-Cluster auch Gene für tRNAs.[1] Welche und ob tRNA-Gene enthalten sind, ist sehr variabel.
Das Primärtranskript der prokaryotischen rDNA wird als 30S-prä-rRNA bezeichnet. Die 16S- und die 23S-rRNA werden durch die Ribonuclease III herausgeschnitten. Der „Feinschnitt“ und die Bearbeitung der 5S-rRNA und der tRNAs erfolgt durch weitere RNA schneidende Enzyme. Damit nicht auch die funktionellen rRNA-Bereiche geschnitten werden, werden diese gleich nach der Transkription methyliert und somit geschützt.
Durch die große Vielfalt der Bakterien gibt es auch Sonderentwicklungen. Bei Mykoplasmen liegt zum Beispiel die 5S-rDNA separiert von der 16S- und 23S-rDNA vor.[2] Beim Planctomycet Planctomyces limnophilus sind zwei Sätze ribosomaler Gene vorhanden, die 16S-rDNA liegt jeweils separat von den nahe beieinander liegenden Clustern aus 23S- und 5S-rDNA.[3]
Archaeen
Bei einigen Archaeen liegt die 5S-rDNA etwas weiter entfernt von den anderen beiden rDNAs. Mitunter (z. B. bei Thermococcus celer) gibt es auch eine weitere, wenige 1000 Basenpaare entfernte 5S-rDNA.[4]
rDNA bei eukaryotischen Organellen
Die rDNAs von Mitochondrien und von Plastiden sind prinzipiell prokaryotenähnlich,[5] weisen jedoch viele Sonderentwicklungen auf.
Plastiden
Der Ribosomenaufbau, die Grundreihenfolge der rRNA-Gene (16S-23S-5S) und deren Sedimentationskoeffizient (70S) sind prokaryotenähnlich. Es gibt auch in der Chloroplasten-rDNA tRNA-Gene in der Transkriptionseinheit. Die rDNA-Cluster können hinsichtlich der Länge, der tRNA-Gene und auch der Introns innerhalb der rRNA-Gene erhebliche Unterschiede aufweisen. Mitunter wird spekuliert, dass die Chloroplastengenome der pflanzlichen Großgruppen unterschiedlicher Herkunft sein könnten.
In höheren Pflanzen wie Tabak (Nicotiana tabacum) und Reis (Oryza sativa) wurde abweichend von der Grundstruktur noch eine 4,5S-rDNA zwischen der 5S- und der 23S-rDNA beschrieben. Diese ist homolog mit dem 3'-Ende der 23S-rDNA.[6] Die 4,5S-rRNA ist mit der 23S- und der 5S-rRNA Teil der großen Ribosomenunterheit. Die Grünalge Chlamydomonas reinhardtii besitzt zwischen der 16S- und 23S-rDNA noch eine 7S- und eine 3S-rDNA. Diese Kombination ist in dieser Alge einzigartig.
Die rDNA befindet sich häufig in den sogenannten „Inverted Repeats“, zwei langen, gegenläufigen Wiederholungseinheiten, z. B. bei der ursprünglichen Glaucocystaceae Cyanophora paradoxa, dem Olisthodiscus luteus (Raphidophyceae) und den meisten grünen Pflanzen. Bei vielen Schmetterlingsblütengewächsen und bei Kiefern ist der inverted repeat sekundär verloren gegangen.[7] Bei der Rotalge Porphyra purpurea und bei Euglena gracilis gibt es keine Inverted Repeats.[8] Bei Euglena liegen drei Repeats und eine zusätzliche 16S-rDNA in direkten Wiederholungseinheiten zusammen. Bei manchen Chromalveolata, z. B. bei Dinoflagellaten[9] und Braunalgen, wie Pylaiella littoralis, gibt es mehrere Plastidenchromosomenringe. Bei P. littoralis liegt die rDNA auf einem Ring wie bei grünen Pflanzen in inverted repeats vor. Im kleinen Chromosomenring liegen ein 16S-Pseudogen und davon entfernt ein zweigeteiltes 23S-rDNA-Gen vor.
Mitochondrien
Die Anordnung der rDNA in Mitochondrien ist sehr heterogen. Sie ist häufig stark von der prokaryotischen Form abgewandelt, sodass zeitweise die Hypothese vertreten wurde, dass die Endosymbiose von Mitochondrien während der Evolution mehrfach stattgefunden habe.
Üblicherweise, so auch beim Menschen, sind nur noch zwei rRNA-Gene vorhanden, je eines für die rRNA der großen und der kleinen Untereinheit der Ribosomen. Beide Gene können mitunter entfernt voneinander liegen. Die gebildeten rRNAs sind bedeutend kleiner als die der Prokaryoten: Bei Tieren 12S in der kleinen Ribosomenuntereinheit und 16S in der großen, bei Pilzen 15S in der kleinen und 21S in der großen Untereinheit.
Eine eigenständige, der 5S-rDNA ähnliche Version wurde nur in einigen Grünalgen (z. B. Prototheca wickerhamii), in Landpflanzen und in dem Einzeller Reclinomonas americana beschrieben. Reclinomonas gilt als ursprünglichster mitochondrienbesitzender Eukaryot. In Rotalgen hingegen, die den grünen Pflanzen evolutionär näher stehen als dieser, ist die 5S-rDNA wiederum nicht mehr nachzuweisen.
Nur bei den Landpflanzen liegt die rDNA noch in Form eines Operons vor.[10] Bei Grünalgen, wie Chlamydomonas reinhardtii sind die rDNA-Gene gestückelt und auch von anderen Genen unterbrochen. Auch bei Reclinomonas ist die Organisation als Operon nicht mehr deutlich.
rDNA im Zellkern der Eukaryoten
Die cytoplasmatischen Ribosomen der Eukaryoten enthalten vier verschiedene rRNAs. Diese können bis zu 90 % der Gesamt-RNA einer Zelle ausmachen. Für die Produktion derart großer Mengen werden für jede der vier rRNAs viele Gene benötigt. Die Gene von dreien (die der 18S-, 5,8S- und 28S-rRNA) liegen jeweils direkt hintereinander und werden gemeinsam transkribiert, und zwar von der RNA-Polymerase I. Solche Transkriptionseinheiten liegen in großer Zahl als tandemartige Wiederholungseinheiten (engl.: Repeats) vor, sie bilden die eigentliche rDNA (siehe Abbildung). Im mikroskopischen Bild der Metaphase können die Wiederholungseinheiten als sekundäre Einschnürungen (Konstriktionen) gesehen werden[11], da die rDNA besonders dicht verpackt vorliegt.
Die Gene der 5S-rRNA bilden eine eigene Genfamilie, die strenggenommen nicht zur rDNA gehört. Sie werden von der RNA-Polymerase III transkribiert. Bei einigen Hefen, wie Hansenula polymorpha oder Saccharomyces cerevisiae, liegen sie direkt hinter den 18S-, 5,8S-, 28S-Repeats, jedoch auf dem Gegenstrang, also in entgegengesetzter Leserichtung. Bei den meisten anderen Hefen und den restlichen Eukaryoten liegen die 5S-rRNA-Gene abseits der 18S-, 5,8S-, 28S-Repeats vor. Sie bilden ebenfalls häufig Tandem-Repeats.
Für die Synthese der Ribosomen wird von allen rRNAs die genau gleiche Anzahl benötigt. Durch die räumliche Abtrennung der 5S-rRNA-Gene wird bei den Eukaryoten (im Gegensatz zu den Prokaryoten) aber nicht automatisch gleich viel hergestellt, sodass eine besondere Regulation erforderlich ist (siehe unten).
Die rDNA ist üblicherweise Bestandteil der Chromosomen. Nur bei einigen Protisten, wie der Schleimpilzgattung Physarum liegt sie extrachromosomal vor.[12]
18S-, 5,8S-, 28S-rDNA-Cluster
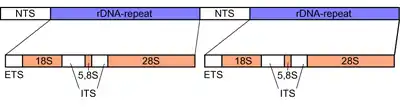
Die 18S-, 5,8S und 28S-rRNA Gene einer Transkriptionseinheit werden auf DNA-Ebene durch zwei sogenannte internal transcribed spacer (ITS) getrennt und gemeinsam von einem external transcribed spacer (ETS) angeführt (siehe Schemazeichnung). Mitunter gibt es auch am Repeat-Ende noch einen ETS. Aufeinander folgende Transkriptionseinheiten werden durch non-transcribed spacer (NTS) getrennt.
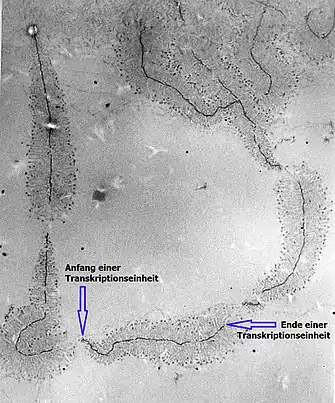
Die nichtranskribierten Spacer vor einzelnen Repeatblöcken bestehen aus einer Vervielfachung von Promotoren (insgesamt 2300 bis 5300 bp), sodass sie eine hohe Bindekraft für die RNA-Polymerasen besitzen. Die hohe Promotoreffektivität bewirkt, dass die rDNA-Gene gleichzeitig vielfach transkribiert werden, was sich in der sogenannten „Miller-Spreitung“ durch „Tannenbaum-Strukturen“ zeigt (siehe Bild).
Von der 45S-prä-rRNA wird als erstes der external transcribed spacer abgetrennt, es entsteht der 41S-rRNA-Vorläufer. Dieser wird im ersten ITS in eine 20S-prä-rRNA und einen 32S-Vorläufer gespalten. Im dritten Schritt, wird vom 20S-Vorläufer der kleine ITS-Teil abgetrennt und die reife 18S-rRNA entsteht. Gleichzeitig werden vom 32S-Vorläufer die ITS herausgeschnitten und die 5,8S-rRNA an den komplementären Zentralteil der 28S-rRNA gebunden. Diese Reaktionen werden von den sogenannten small nucleolar ribonucleoprotein particles (snoRNP) katalysiert.
In Drosophila, aber auch in vielen anderen Organismen, kann die 28S-rDNA eine sogenannte intervening sequence (IVS) enthalten. Diese wird den „Gruppe-I-Introns“ zugerechnet, welche autokatalytisch spleißen. Dabei knüpft ein externes Guanosinmonophosphat an das 3'-OH des Intronanfangs und das nun freigewordene 3'-Ende des ersten Exons an das 5'-Ende des zweiten Exons. Dadurch wird das Intron freigesetzt, abgebaut und die Exons verknüpft. Wann dieses Spleißen während der rRNA-Prozession stattfindet, ist bisher jedoch nicht geklärt.
Je nach Spezies kann die Anordnung, Verteilung und die Anzahl der Transkriptionseinheiten stark schwanken. Drosophila-Männchen haben etwa 150 Kopien, die Weibchen 250. Für die Ackerbohne (Vicia faba) wurden 22.000 Kopien beschrieben.[13]
Beim Menschen sind die etwa 43 kb langen 18S-5,8S-28S-Transkriptionseinheiten tandemartig zu 30 oder mehr als Cluster angeordnet. Die insgesamt zehn Cluster liegen auf den akrozentrischen Chromosomenpaaren 13, 14, 15, 21 und 22. Die Gesamtzahl der Transkriptionseinheiten wird auf 500 Kopien geschätzt[14]. Jede Transkriptionseinheit besitzt einen eigenen komplex regulierten Promotor. Zwischen den Wiederholungseinheiten pro Cluster gibt es in regelmäßigen Abständen sogenannte Spacer-Promoter in NTS-Bereichen. Diese transkribieren eine noncoding RNA, welche die Transkription der einzelnen Transkriptionseinheiten über einen Proteinkomplex mit Namen NoRC reguliert.
5S-rRNA-Gene
5S-rRNA-Gene liegen ebenfalls in Tandemwiederholungen vor. Beim Menschen ist ein Block auf dem langen Arm von Chromosom 1 nahe dem Telomer vorhanden, bei Drosophila melanogaster ein Block auf Chromosom 3. Beim Krallenfrosch (Xenopus laevis) sind etwa 24.000 Kopien nahe den Enden der meisten Chromosomen verteilt. Die DNA-Abschnitte zwischen 5S-rRNA-Genen heißen ebenfalls non-transcribed spacer. Sie können sowohl zwischen einzelnen Genkopien als auch zwischen nahe verwandten Arten stark variieren.
5S-rRNA-Gene werden im Unterschied zu den anderen rRNA-Genen, aber ebenso wie die tRNAs, von RNA-Polymerase III transkribiert. Sie besitzen keine Introns und werden auch nicht prozessiert. Der Promoter liegt innerhalb der codierenden Sequenz und wird internal control region genannt. Er besteht aus einer Box A, einem intermediate element und einer Box C (von 5' nach 3').
Die 5S-rRNA-Gene des Krallenfrosches unterscheiden sich innerhalb eines Individuums dadurch, dass einige nur in Eizellen, andere hingegen nur in Somazellen aktiv sind.
Regulation des rRNA-Gehalts
Eukaryotische Zellen müssen dafür sorgen, dass 5S-RNA und die anderen RNAs in den gleichen Mengen produziert werden. Eine unterschiedliche Prozessivität der RNA-Polymerasen I und III wird unter anderem durch unterschiedliche Kopienanzahlen der rRNA Gene ausgeglichen. Zudem kann auch die unterschiedliche Struktur der jeweiligen NTS-Regionen zu einer differentiellen Regulation führen. So wird in bestimmten Situationen nur ein Teil der zellulären rDNA transkribiert.
In manchen Situationen muss jedoch der Gesamtgehalt der rDNA-Gene verändert werden. Bei den Polytänchromosomen von Drosophila wird die rDNA zum Beispiel unterrepliziert. Häufiger jedoch ist es nötig, mehr Ribosomen zu produzieren, etwa in Eizellen. Dies wird durch eine intrazelluläre Vervielfältigung (Amplifikation) der Gene ermöglicht. Dabei werden rDNA-Gene aus dem Chromosom herausgeschnitten, zirkularisiert und durch rolling circle-Replikation amplifiziert. Diese stark anfärbbaren, 1901 erstmals beschriebenen DNA-Ringe werden nach ihrem Entdecker Giardina-bodies genannt. Sie sind etwa beim Gelbrandkäfer, beim Heimchen, beim Krallenfrosch und beim Hamster beobachtet worden. Der molekulare Mechanismus ist noch ungeklärt.
Ribosomale DNA und Evolution
Ribosomen sind komplexe und lebensnotwendige Bestandteile einer jeden Zelle, dementsprechend ist die Konservierung recht hoch. Bei Eukaryoten gibt es im Gegensatz zu den Prokaryoten mehrere hundert Kopien, sodass eine unabhängige Evolution zu erwarten wäre. Die Cluster werden allerdings durch inäquales Crossing over homogenisiert und somit wie ein Einzelgen vererbt.[15][16] Bei Hybriden, die beide Genome der Stammeltern besitzen (allotetraploid), wird nur die rDNA eines Elters transkribiert, was als „nukleoläre Dominanz“ bezeichnet wird.[17][18]
Evolution der rDNA
Auffällig ist insbesondere die Repeatstruktur der rDNA. Sie findet sich in Bakterien, Archaeen, im eukaryotischen Cytoplasma und manchen (pflanzlichen) mitochondrialen DNAs. So ist durchaus eingänglich, dass die 16S-rDNA mit der 18S-rDNA[19] und die 23S-rDNA mit der 28S-rDNA homologisierbar sind. Die 5.8S-rDNA ist homolog zum Ende der 23S-rDNA von Prokaryoten[20] Die 5S-rDNA der Prokaryoten ist homolog zur 5S-rDNA der Eukaryoten.
Wie vor allem die rDNA in Mitochondrien zeigt, kann es zu diversen Veränderungen in der Anordnung kommen, ohne dass die Funktionalität der Ribosomen darunter leiden muss. Da die genauen Wechselwirkungen der rRNAs noch nicht genug verstanden sind, bleiben viele Fragen noch offen.
Evolutionsforschung mit der rDNA

Die zentrale Bedeutung der rDNA im Ribosom ist auch für Evolutionsforscher von Interesse, denn defekte Genkopien sind ganz überwiegend schädlich – und das für alle Proteinsynthesen der Zelle – und werden ausselektiert. Zudem sind Ribosomen in allen Lebewesen vorhanden, sogar der Brückenschlag zwischen Prokaryoten und Eukaryoten ist möglich. So wird beispielsweise die 16S-rDNA genutzt, um Großgruppen von Bakterien zu systematisieren. Aber auch bei der Großsystematik der Eukaryoten sind die Sequenzen der reifen rRNA der Ribosomenuntereinheiten durchaus ein beliebter Marker. Die rDNA der Plastiden und Mitochondrien wird ebenso häufig verwendet.
Ein Vergleich zwischen Prokaryoten und Eukaryoten hinsichtlich ihrer ribosomalen DNA zeigt nur wenige kurze Sequenzen, die gleich sind. Doch zeigen die rRNA-Moleküle trotz unterschiedlicher Basenzusammensetzung morphologisch in Form und Faltung von Sekundärstrukturen eine Ähnlichkeit.
Die weniger stark durch Selektion beeinflussten ITS-Sequenzen spielen bezüglich der Phylogenie auf Gattungs- und Artebene eine Rolle. Üblicherweise wird die 5,8S-rDNA gleich mitsequenziert, da sie größenmäßig kaum ins Gewicht fällt. Um statistisch eine größere Sicherheit zu bekommen, werden die ITS-Sequenzen durch mindestens einen weiteren Marker ergänzt. Die Nutzung von ITS-Sequenzen ist mittlerweile zurückgegangen – einerseits, da andere Marker des Zellkerns verfügbarer werden und andererseits, da die Nutzung von ITS größere Probleme für die molekular basierte Phylogenie mit sich bringt. So sind zwar die einzelnen Basen nicht konserviert, sehr wohl aber gewisse sekundäre Strukturelemente wie Faltungen, sodass die Mutationen in dieser Hinsicht nicht frei sind. Auch kann die Angleichung verschiedener ITS-Kopien bei Hybriden in nur wenigen Generationen erfolgen, womit ITS sich ähnlich den Chloroplasten verhalten kann. Dennoch ist die hohe Kopieanzahl und somit die bessere Verfügbarkeit labortechnisch ein Argument für ITS-Vergleiche.
Geschichte
Ribosomen von Eukaryoten bestehen aus vier verschiedenen rRNAs, die je zu gleicher Anzahl vorkommen. Cyrus Levinthal et al. fanden 1962 heraus, dass rRNAs und tRNAs von der DNA transkribiert werden und sich nicht selbständig replizieren können.[21] Im Jahre 1965 zeigten Sol Spiegelman und Ferrucio Ritossa durch die Hybridisierung von rRNA mit Drosophila-Zellkernen mit unterschiedlicher Anzahl an Nukleolusorganisatorregionen, dass diese Regionen die codierende rDNA enthalten.[22]
Carl Woese veröffentlichte 1977[23] seinen tree of life, einen Stammbaum, der alle Großgruppen erstmals in Zusammenhang bringen konnte. Dieser basierte auf Sequenzen der 16S-rDNA aus Mitochondrien und stellte die Archaeen als eigenes Reich heraus.
Thomas R. Cech entdeckt 1982 die intervening sequence in der 28S-rRNA des Ciliaten Tetrahymena thermophila, ein autokatalytisch spleißendes Intron. Für diese bahnbrechende Entdeckung, dass RNA auch enzymatische Funktionen übernehmen und sich selbst spleißen kann, bekam er mit Sidney Altman 1989 den Nobelpreis für Chemie.
Einzelnachweise
- J. Brosius, T. J. Dull, D. D. Sleeter, H. F. Noller: Gene Organization and Primary Structure of a Ribosomal RNA Operon from „Escherichia coli“. In: J. Mol. Biol. 148/1981, S. 107–127
- C. Taschke, M.-Q. Klinkert, J. Wolters, R. Herrmann: Organization of the ribosomal RNA genes in „Mycoplasma hyopneumoniae“: The 5S rRNA gene is separated from the 16S and 23S rRNA genes. In: Mol Gen Genet. 205/1986, S. 428–433
- N. Ward-Rainey, F. A. Rainey, E. M. H. Wellington, E. Stackebrandt: Physical Map of the Genome of „Planctomyces limnophilus“, a Representative of the Phylogenetically Distinct Planctomycete Lineage. In: Journal of Bacteriology. April 1996, S. 1908–1913
- H. Neumann, A. Gierl, J. Tu, J. Leibrock, D. Staiger, W. Zillig: Organization of the Genes for Ribosomal RNA in Archaebacteria. In: Mol Gen Genet. 192/1983, S. 66–72
- Z. Schwarz, H. Koessel: The primary structure of 16 S rDNA from „Zea mays“ chloroplast is homologous to „E. coli“ 16-S rRNA. In: Nature. 283/1980, S. 739–742
- K. Edwards, J. Bedbrook, T. Dyer, H. Kössel: 4.5S rRNA from „Zea mays“ chloroplasts shows structural homology with the 3’-end of prokaryotic 23S rRNA. In: Biochem. Int. 2/1981, S. 533–538
- M. Sugiura: The chloroplast genome. In: Plant Molecular Biology. 19/1992, S. 149–168
- J. D. Palmer: Comparative organization of chloroplast genomes. In: Ann. Rev. Genet. 19/1985, S. 325–354
- C. J. Howe, R. Ellen, R. Nisbet, A. C. Barbrook: The remarkable chloroplast genome of dinoflagellates. In: Journal of Experimental Botany. 59(5), März 2008, S. 1035–1045, doi:10.1093/jxb/erm292
- C. Leblanc, O. Richard, B. Kloareg: Origin and evolution of mitochondria: what have we learnt from red algae? In: Current Genetics. 31/1997, S. 193–207
- T. Schmidt, T. Schwarzacher, J. S. Heslop-Harrison: Physical mapping of rRNA genes by fluorescent in-situ hybridization and structural analysis of 5S rRNA genes and intergenic spacer sequences in sugar beet (Beta vulgaris). in: Theoretical and Applied Genetics Ausgabe. 88, S. 629–636 (1994)
- S. Johansen, T. Johansen, F. Haugli: Extrachromosomal ribosomal DNA of „Didymium iridis“: sequence analysis of the large subunit ribosomal RNA gene and sub-telomeric region. In: Curr Genet. 22/1992, S. 305–312
- S. O. Rogers, A. J. Bendich: Ribosomal RNA genes in plants: variability in copy number and in the intergenic spacer. In: Plant Molecular Biology. 9(5)/1987, S. 509–520
- Liao Daiqing, in: Encyclopedia of the human genome, 2003, Macmillan Publishers LTd, Nature Publishing Group. pdf (Memento vom 30. April 2006 im Internet Archive)
- N. Arnheim, M. Krystal, R, Schmickel, G. Wilson, O. Ryder, E. Zimmer: „Molecular evidence for genetic exchanges among ribosomal genes on nonhomologous chromosomes in man and apes“ In: Proc. Natl. Acad. Sci. USA. 77(12)/1980, S. 7323–7327
- D. M. Hillis, M. T. Dixon: Ribosomal DNA: Molecular Evolution and Phylogenetic Inference. In: The Quarterly Review of Biology. 66(4)/1991, S. 411–453
- T. Honjo, R. H. Reeder: Preferential transcription of Xenopus laevis ribosomal RNA in interspecies hybrids between Xenopus laevis and Xenopus mulleri. In: J. Mol. Biol. 80/1973, S. 217–228
- Z. J. Chen, C. S. Pikaard: Transcriptional analysis of nucleolar dominance in polyploid plants: biased expression/silencing of progenitor rRNA genes is developmentally regulated in Brassica. In: Proc. Natl. Acad. Sci. USA. 94/1997, S. 3442–3447
- P. M. Rubtsov, M. M. Musakhanov, V. M. Zakhaiyev, A. S. Krayev, K. G. Skryabin, A. A. Bayev: The structure of the yeast ribosomal RNA genes. I. The complete nudeotide sequence of the 18S ribosomal RNA gene from „Saccharomyces cerevisiae“. In: Nucleic Acids Research. 8/23/1980, S. 5779–5794
- G. J. Olsen, D. J. Lane, S. J. Giovannoni, N. R. Pace: Microbial ecology and evolution: a ribosomal RNA approach. In: Ann. Rev. Microbiol. 40/1986, S. 337–365
- C. Levinthal, A. Heynan, A. Higa: Messenger mRNA turnover and protein synthesis in „B. subtilis“ inhibited by Actinomycin D. In: Proc. Nat. Acad. Sci. 48/1962, S. 1631–1638
- F. Ritossa, S. Spiegelman: Localization of DNA complementary to ribosomal RNA in the nucleolus oreganizer region of „Drosophila melanogaster“. In: Proc. Nat. Acad. Sci. 53/1965, S. 737–745
- C. R. Woese, G. E. Fox: Phylogenetic Structure of the Prokaryotic Domain: The Primary Kingdoms. In: Proc. Nat. Acad. Sci. 74(11)/1977, S. 5088–5090
Literatur
Prokaryoten
- A. M. Nedelcu: Fragmented and scrambled mitochondrial ribosomal RNA coding regions among green algae: a model for their origin and evolution. In: Mol Biol Evol. 14(5)/1997, S. 506–517; PMID 9159928.
- Seyffert: Lehrbuch der Genetik. Spektrum, 2. Aufl., 2003.
Eukaryoten
- Graw: Genetik.; 4. Auflage, 2006 (ehem. Hennig)
- L. Klabunde: Koproduktion pharmazeutischer Proteine und Hilfsfaktoren zur Optimierung mikrobieller Expressionssysteme bei Beschränkung auf ein einziges integratives Vektorsystem. 1996
- N. Neves, M. Delgado: Ribosomal DNA heterochromatin in plants. Cytogenet Genome Res 109:104–111 (2005); PMID 15753565.