Nukleolusorganisatorregion
Als Nukleolusorganisatorregion (NOR) wird in der Genetik der Abschnitt eines Chromosoms bezeichnet, der für die Bildung des Nukleolus oder Kernkörperchens im Zellkern (Nukleus) einer (eukaryotischen) Zelle wesentlich ist. Die NOR des menschlichen Genoms sind in den kurzen Armen der fünf akrozentrischen Chromosomen 13, 14, 15, 21 und 22 lokalisiert. Diese Chromosomenabschnitte enthalten jeweils ribosomale DNA (rDNA), die in ribosomale RNA (rRNA) umgeschrieben werden kann, was vornehmlich während der Interphase eines Zellzyklus geschieht.
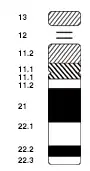
Eine NOR gibt sich in der Mitose als dünner Faden, als Sekundäreinschnürung zu erkennen. (Die häufigeren Einschnürungen an den Stellen der Zentromere gelten als primäre Konstriktionen.)
Von der rDNA zur rRNA
Die Transkription der rDNA-Abschnitte nukleolusorganisierender Regionen erfolgt durch RNA-Polymerase I in den sich bildenden Nukleolen. Aus dem Transkript, einer 45S-prä-rRNA, werden durch anschließende Prozessierung drei verschiedene (18S- sowie 5,8S- und 28S-) rRNA-Molküle für die beiden Untereinheiten von Ribosomen gebildet.
Im Interphasekern liegen innerhalb eines Nukleolus die Gene für rRNA auf den entfalteten (dekondensierten) Chromosomen gut zugänglich vor. Sie werden mit hoher Genaktivität abgelesen, sodass zahlreiche 28S-, 18S- und 5,8S-rRNA-Stränge entstehen, die sich hier nun mit 5S-rRNA und verschiedenen ribosomalen Proteinen passend zu zwei Arten von Ribosomuntereinheiten zusammenfügen.
Die Gene für die 5S-rRNA liegen allerdings in den (meisten) eukaryotischen Genomen außerhalb einer NOR. Das gilt auch für das menschliche Genom: Es enthält die vielfach wiederholten Sequenzen im Chromosom 1, und zwar in dessen langen Arm, in den Regionen 1q42.11–q42.13d[1] sowie 1q31.[2]
Eine Ausnahme wurde kürzlich bei einer Schildkrötenart entdeckt, bei der es neben dem „regulären“ Genort einige zusätzliche rDNA-Sequenzen für 5S-rRNA in der NOR gibt.[3]
Vom Zellkern ins Zytoplasma
Die kleinen und großen Untereinheiten gelangen durch Kernporen vom Kernplasma ins Cytosol, wo sie sich erst zu Ribosomen zusammensetzen. Dann stehen sie für die Proteinbiosynthese bereit; in diesem Vorgang wird eine mRNA in ein Protein übersetzt (translatiert).
NOR in der Mitose
Mitten in der Interphase (in der S-Phase) wird der DNA-Strang eines Chromatids verdoppelt. Zur mitotischen Teilung des Zellkerns werden ab der Prophase die beiden Chromatide eines Chromosoms dichter gepackt. Mit ihren Chromosomen kondensieren auch die Nukleolusorganisatorregionen: die rDNA wird verpackt und damit stillgelegt. Wenn eine NOR bis zur Metaphase nicht die Verpackungsdichte ihres umgebenden Chromosomenarms erreicht, bleibt sie als eine Art Einschnürung (Konstriktion) lichtmikroskopisch sichtbar.[4] Durch derartige Einschnürungen können Endbereiche der Nukleolus-Chromosomen so abgegliedert werden, dass sie als eine Art Anhängsel erscheinen, Satellit oder Trabant genannt.
Die NOR-Anzahl der Metaphase korreliert nicht unbedingt mit der Anzahl der Nukleolen in der Interphase; manche NOR wird nicht aktiv, manche formt mit anderen NOR einen gemeinsamen Nukleolus. Und nicht jede Einschnürung stellt eine NOR dar, denn jedes Metaphase-Chromosom hat an der Stelle des Zentromers eine (primäre) Konstriktion.
Geschichte
Emil Heitz untersuchte die Kernteilungen bei mehren Pflanzen und war der Meinung: „Einzelne, bestimmte Chromosomen stehen mit der Bildung je eines Nukleolus irgendwie in ursächlichem Zusammenhang.“ Mit Mitosen von Vicia faba lieferte er dann den „Nachweis der Entstehung des Kernkörperchens am Faden des [einzigen] Trabantenchromosoms.“ Er formulierte die allgemeine Hypothese: „Die Nukleolen entstehen an bestimmten Chromosomen („Nukleolenchromosomen“) und nur an diesen.“[5] Den Zusammenhang zwischen einem Nukleolus und den ihn organisierenden Regionen erklärte auch die Maisgenetikerin Barbara McClintock 1934.[6]
Dass eine sekundäre Konstriktion in Chromosomen von Altweltaffen zusammen mit NOR auftritt, fiel Chiarelli 1971 auf.[7]
Literatur
- Marco Di Stefano, Hans-Wilhelm Nützmann, Marc A Marti-Renom, Daniel Jost: Polymer modelling unveils the roles of heterochromatin and nucleolar organizing regions in shaping 3D genome organization in Arabidopsis thaliana. In: Nucleic Acids Res 49, 4, 2021: 1840–1858. PDF.
- Qinyu Hao, Kannanganattu V Prasanth: Regulatory roles of nucleolus organizer region-derived long non-coding RNAs. In: Mamm Genome 26. August 2021. doi: 10.1007/s00335-021-09906-z.
- Vera Hemleben, Donald Grierson, Nikolai Borisjuk, Roman A Volkov, Ales Kovarik: Personal perspectives on plant ribosomal RNA genes research: From precursor-rRNA to molecular evolution. In: Front Plant Sci 12, 2021: 797348. PDF.
- Hazel Mangan, Brian McStay: Human nucleoli comprise multiple constrained territories, tethered to individual chromosomes. In: Genes Dev 35, 7–8, 2021: 483–488. PDF.
- Lisa M Ogawa, Amber F Buhagiar, Laura Abriola, Bryan A Leland, Yulia V Surovtseva, Susan J Baserga: Increased numbers of nucleoli in a genome-wide RNAi screen reveal proteins that link the cell cycle to RNA polymerase I transcription. In: Mol Biol Cell 32, 9, 2021: 956–973. PDF.
- Joseph G Gall: The human nucleolus organizer regions. In: Genes Dev 33, 23–24, 2019: 1617–1618. PDF.
- Cristiana Bersaglieri, Raffaella Santoro: Genome organization in and around the nucleolus. In: Cells 8, 6, 2019: 579. PDF.
- Marjolein van Sluis, Michael Ó Gailín, Joseph G W McCarter, Hazel Mangan, Alice Grob, Brian McStay: Human NORs, comprising rDNA arrays and functionally conserved distal elements, are located within dynamic chromosomal regions. In: Genes Dev 33, 23–24, 2019: 1688–1701. PDF.
Einzelnachweise
- Pernille D Sørensen, B Lomholt, Sune Frederiksen, N Tommerup: Fine mapping of human 5S rRNA genes to chromosome 1q42.11-q42.13. In: Cytogenet Cell Genet 57, 1991: 26–29, 1991. Abstract.
- B Lomholt, Sune Frederiksen, J Nederby Nielsen, C Hallenberg: Additional assignment of the human 5S rRNA genes to chromosome region 1q31. In: Cytogenet Cell Genet 70, 1995: 76–79. Abstract.
- Asya G Davidian, Alexander G Dyomin, Svetlana A Galkina, Nadezhda E Makarova, Sergey E Dmitriev, Elena R Gaginskaya: 45S rDNA repeats of turtles and crocodiles harbor a functional 5S rRNA gene specifically expressed in oocytes. In: Mol Biol Evol 39, 1, 2022: msab324. PDF.
- A. S. Henderson, D. Warburton, K. C. Atwood: Localization of rDNA in the chromosome complement of the Rhesus (Macaca mulatta). In: Chromosoma. Band 44, Nr. 4, Dezember 1974, S. 367–370, doi:10.1007/BF00284896.
- Emil Heitz: Die Ursache der gesetzmäßigen Zahl, Lage, Form und Größe pflanzlicher Nukleolen. In: Planta 12, 1931: 775–844. Zitate: 781, 839.
- Barbara McClintock: The relation of a particular chromosomal element to the development of the nucleoli in Zea mays. In: Zeitschrift für Zellforschung und mikroskopische Anatomie. Band 21, Nr. 2, Januar 1934, S. 294–326, doi:10.1007/BF00374060.
- A. B. Chiarelli: Comparative cytogenetics in primates and its relevance in human cytogenetics. In: A. B. Chiarelli (Hrsg.): Comparative genetics in monkeys, apes and man. Academic Press, 1971, S. 273–308.