Internal transcribed spacer
Ein internal transcribed spacer ist eine Nukleotidsequenz zwischen den rRNA-Genen (rDNA) oder zwischen den rRNA-Abschnitten in den aus der rDNA während der Transkription entstehenden Transkripten.
Eigenschaften
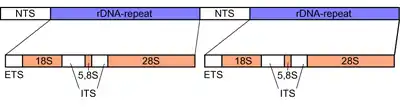
Die RNA des Ribosoms (rRNA) besteht aus verschiedenen RNA-Molekülen, die in Eukaryoten aus unterschiedlichen Transkripten der rRNA-Gene (rDNA) erzeugt wurden. Innerhalb dieser Sequenzen liegen die ITS, die als spacer im Gegensatz zur rRNA nicht im fertigen Ribosom vorkommen. Im Gegensatz zu ITS liegen die external transcribed sequences (ETS) außerhalb der ribosomalen Untereinheiten, kommen aber ebenso wenig im späteren Ribosom vor. Die ITS und ETS werden nach der Transkription herausgeschnitten und anschließend abgebaut,[1][2] teilweise unter Beteiligung von snoRNA.
In Bakterien und Archaeen befinden sich die ITS zwischen der 16 S und der 23 S rRNA.[3]
In Eukaryoten existieren zwei verschiedene ITS: ITS1 liegt zwischen der 18 S und der 5,8 S rRNA und ITS2 zwischen der 5,8 S und der 26 S (in Pflanzen) bzw. der 28 S rRNA (in Tieren und Pilzen[4]). Während ITS1 der bakteriellen ITS entspricht, entstand ITS2 später in der 23 S rRNA durch Duplikation.[5]
Verwendung
Die 5,8 S rRNA von Eukaryoten und die umgebenden ITS werden zur Untersuchung der Phylogenetik verwendet, da ITS vergleichsweise häufig mutieren und die Gene der rRNA in vielen Kopien im Genom vorkommen und dadurch tendenziell weniger zu falsch negativen Ergebnissen führen, z. B. in Bakterien,[6] in Pflanzen,[7] in Stechmücken[8] und in Pilzen.[9][10] Innerhalb der ribosomalen DNA der Pilze sind die ITS die Sequenzen mit der deutlichsten Unterscheidbarkeit innerhalb einer Art oder zwischen Arten.[11] Standard-Primerpaare für die Polymerase-Kettenreaktion zur Unterscheidung von Pilzen sind ITS1 mit ITS4.[12] Für die Unterscheidung von Basidiomyceten und Mycorrhiza werden andere Primerpaare verwendet.[13]
| Taxonomische Gruppe | Reich | Taxonomische Stufe | Jahr | Referenz |
|---|---|---|---|---|
| Pilze | Pilze | Genera | 1990 | White et al.[12] |
| Asteraceae: Compositae | Pflanzen | Spezies | 1992 | Baldwin et al.[7] |
| Basidiomyceten/Ascomyceten | Pilze | Genera | 1993 | Gardes et al.[13] |
| Viscaceae: Arceuthobium | Pflanzen | Spezies | 1994 | Nickrent et al.[14] |
| Poaceae: Zea | Pflanzen | Spezies | 1996 | Buckler & Holtsford[15] |
| Leguminosae: Medicago | Pflanzen | Spezies | 1998 | Bena et al.[3] |
| Orchidaceae: Diseae | Pflanzen | Genera | 1999 | Douzery et al.[16] |
| Odonota: Calopteryx | Tiere | Spezies | 2001 | Weekers et al.[17] |
| Hefen von klinischer Bedeutung | Pilze | Genera | 2001 | Chen et al.[18] |
| Poaceae: Saccharinae | Pflanzen | Genera | 2002 | Hodkinson et al.[19] |
| Plantaginaceae: Plantago | Pflanzen | Spezies | 2002 | Rønsted et al.[20] |
| Jungermanniopsida: Herbertus | Pflanzen | Spezies | 2004 | Feldberg et al.[21] |
| Anophelinae Südamerikas | Tiere | Spezies | 2006 | Marelli et al.[8] |
| Pinaceae: Tsuga | Pflanzen | Spezies | 2008 | Havill et al.[22] |
| Brassicaceae | Pflanzen | Stämme | 2010 | Warwick et al.[23] |
| Ericaceae: Erica | Pflanzen | Spezies | 2011 | Pirie et al.[24] |
| Diptera: Bactrocera | Tiere | Spezies | 2014 | Boykin et al.[25] |
| Scrophulariaceae: Scrophularia | Pflanzen | Spezies | 2014 | Scheunert & Heubl[26] |
| Potamogetonaceae: Potamogeton | Pflanzen | Spezies | 2016 | Yang et al.[27] |
Einzelnachweise
- A. W. Coleman: Nuclear rRNA transcript processing versus internal transcribed spacer secondary structure. In: Trends in genetics : TIG. Band 31, Nummer 3, März 2015, S. 157–163, doi:10.1016/j.tig.2015.01.002, PMID 25648500.
- Bernard Michot, Jean-Pierre Bachellerie, Francoise Raynal: Structure of mouse rRNA precursors. Complete sequence and potential folding of the spacer regions between 18S and 28S rRNA. In: Nucleic Acids Research. 11, Nr. 10, 25. Mai 1983, ISSN 0305-1048, S. 3375–3391. doi:10.1093/nar/11.10.3375.
- Gilles Bena, Marie-France Jubier, Isabelle Olivieri, Bernard Lejeune: Ribosomal External and Internal Transcribed Spacers: Combined Use in the Phylogenetic Analysis of Medicago (Leguminosae). In: Journal of Molecular Evolution. 46, Nr. 3, 1998, ISSN 0022-2844, S. 299–306. doi:10.1007/PL00006306.
- Ivo R. Horn, Peter A. Verleg, Nafiesa Z. Ibrahim, Khadiedjah Soeleman, Floris van Kampen: Mushroom DNA barcoding project: Sequencing a segment of the 28S rRNA gene. In: Biochemistry and Molecular Biology Education. Band 48, Nr. 4, 2020, ISSN 1539-3429, S. 404–410, doi:10.1002/bmb.21388, PMID 32585770, PMC 7497104 (freier Volltext) – (wiley.com [abgerufen am 11. Februar 2022]).
- Denis L.J. Lafontaine, David Tollervey: The function and synthesis of ribosomes. In: Nature Reviews Molecular Cell Biology. 2, 2001, S. 514, doi:10.1038/35080045.
- J. Trček, F. Barja: Updates on quick identification of acetic acid bacteria with a focus on the 16S-23S rRNA gene internal transcribed spacer and the analysis of cell proteins by MALDI-TOF mass spectrometry. In: International journal of food microbiology. Band 196, März 2015, S. 137–144, doi:10.1016/j.ijfoodmicro.2014.12.003, PMID 25589227.
- B. G. Baldwin: Phylogenetic utility of the internal transcribed spacers of nuclear ribosomal DNA in plants: an example from the compositae. In: Molecular phylogenetics and evolution. Band 1, Nummer 1, März 1992, S. 3–16, PMID 1342921.
- M. T. Marrelli, M. A. Sallum, O. Marinotti: The second internal transcribed spacer of nuclear ribosomal DNA as a tool for Latin American anopheline taxonomy - a critical review. In: Memorias do Instituto Oswaldo Cruz. Band 101, Nummer 8, Dezember 2006, S. 817–832, PMID 17293975.
- Y. C. Chen, J. D. Eisner, M. M. Kattar, S. L. Rassoulian-Barrett, K. Lafe, U. Bui, A. P. Limaye, B. T. Cookson: Polymorphic internal transcribed spacer region 1 DNA sequences identify medically important yeasts. In: Journal of clinical microbiology. Band 39, Nummer 11, November 2001, S. 4042–4051, doi:10.1128/JCM.39.11.4042-4051.2001, PMID 11682528, PMC 88485 (freier Volltext).
- Kabir G. Peay, Peter G. Kennedy, Thomas D. Bruns: Fungal Community Ecology: A Hybrid Beast with a Molecular Master. In: BioScience. 58, 2008, S. 799, doi:10.1641/b580907.
- C. L. Schoch, K. A. Seifert, S. Huhndorf, V. Robert, J. L. Spouge, C. A. Levesque, W. Chen: Nuclear ribosomal internal transcribed spacer (ITS) region as a universal DNA barcode marker for Fungi. In: Proceedings of the National Academy of Sciences. Band 109, Nummer 16, April 2012, S. 6241–6246, doi:10.1073/pnas.1117018109, PMID 22454494, PMC 3341068 (freier Volltext).
- T. J. White, T. Bruns, S. Lee, J. Taylor: Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics. In: PCR Protocols: a Guide to Methods and Applications (1990), Kapitel 18, S. 315–322.
- M. Gardes, T. D. Bruns: ITS primers with enhanced specificity for basidiomycetes–application to the identification of mycorrhizae and rusts. In: Molecular ecology. Band 2, Nummer 2, April 1993, S. 113–118, PMID 8180733.
- Daniel L. Nickrent, Kevin P. Schuette, Ellen M. Starr: A Molecular Phylogeny of Arceuthobium (Viscaceae) Based on Nuclear Ribosomal DNA Internal Transcribed Spacer Sequences. In: American Journal of Botany. 81, Nr. 9, 1. Januar 1994, S. 1149–1160. doi:10.2307/2445477.
- E. S. Buckler, T. P. Holtsford: Zea systematics: ribosomal ITS evidence.. In: Molecular Biology and Evolution. 13, Nr. 4, 1. April 1996, ISSN 0737-4038, S. 612–622. PMID 8882504.
- Emmanuel J. P. Douzery, Alec M. Pridgeon, Paul Kores, H. P. Linder, Hubert Kurzweil, Mark W. Chase: Molecular phylogenetics of Diseae (Orchidaceae): a contribution from nuclear ribosomal ITS sequences. In: American Journal of Botany. 86, Nr. 6, 1. Juni 1999, ISSN 0002-9122, S. 887–899. PMID 10371730.
- Peter H. H. Weekers, Johan F. De Jonckheere, Henri J. Dumont: Phylogenetic Relationships Inferred from Ribosomal ITS Sequences and Biogeographic Patterns in Representatives of the Genus Calopteryx (Insecta: Odonata) of the West Mediterranean and Adjacent West European Zone. In: Molecular Phylogenetics and Evolution. 20, Nr. 1, 1. Juli 2001, S. 89–99. doi:10.1006/mpev.2001.0947.
- Chen,Y-C, J. D. Eisner, M. M. Kattar, S. L. Rassoulian-Barrett, K. Lafe, A. P. Limaye, and B. T. Cookson: Polymorphic Internal Transcribed Spacer Region 1 DNA Sequences Identify Medically Important Yeasts. In: J. Clin. Microbiol.. 39, Nr. 11, 2001, S. 4042–4051. doi:10.1128/JCM.39.11.4042-4051.2001. PMID 11682528. PMC 88485 (freier Volltext).
- Trevor R. Hodkinson, Mark W. Chase, Dolores M. Lledó, Nicolas Salamin, Stephen A. Renvoize: Phylogenetics of Miscanthus, Saccharum and related genera (Saccharinae, Andropogoneae, Poaceae) based on DNA sequences from ITS nuclear ribosomal DNA and plastid trnL intron and trnL-F intergenic spacers. In: Journal of Plant Research. 115, Nr. 5, Februar, ISSN 0918-9440, S. 381–392. doi:10.1007/s10265-002-0049-3.
- Nina Rønsted, Mark W. Chase, Dirk C. Albach, Maria Angelica Bello: Phylogenetic relationships within Plantago (Plantaginaceae): evidence from nuclear ribosomal ITS and plastid trnL-F sequence data. In: Botanical Journal of the Linnean Society. 139, Nr. 4, 1. August 2002, ISSN 1095-8339, S. 323–338. doi:10.1046/j.1095-8339.2002.00070.x.
- K. Feldberg, H. Groth, R. Wilson, A. Schäfer-Verwimp, J. Heinrichs: Cryptic speciation in Herbertus (Herbertaceae, Jungermanniopsida): Range and morphology of Herbertus sendtneri inferred from nrITS sequences. In: Plant Systematics and Evolution. 249, Nr. 3–4, 4. November 2004, ISSN 0378-2697, S. 247–261. doi:10.1007/s00606-004-0221-4.
- Nathan P. Havill, Christopher S. Campbell, Thomas F. Vining, Ben LePage, Randall J. Bayer, Michael J. Donoghue: Phylogeny and Biogeography of Tsuga (Pinaceae) Inferred from Nuclear Ribosomal ITS and Chloroplast DNA Sequence Data. In: Systematic Botany. 33, Nr. 3, 1. Juli 2008, S. 478–489. doi:10.1600/036364408785679770.
- Suzanne I. Warwick, Klaus Mummenhoff, Connie A. Sauder, Marcus A. Koch, Ihsan A. Al-Shehbaz: Closing the gaps: phylogenetic relationships in the Brassicaceae based on DNA sequence data of nuclear ribosomal ITS region. In: Plant Systematics and Evolution. 285, Nr. 3–4, 13. April 2010, ISSN 0378-2697, S. 209–232. doi:10.1007/s00606-010-0271-8.
- Michael D. Pirie, E. G. H. Oliver, Dirk U. Bellstedt: A densely sampled ITS phylogeny of the Cape flagship genus Erica L. suggests numerous shifts in floral macro-morphology. In: Molecular Phylogenetics and Evolution. 61, Nr. 2, 1. November 2011, S. 593–601. doi:10.1016/j.ympev.2011.06.007.
- L. M. Boykin, M. K. Schutze, M. N. Krosch, A. Chomič, T. A. Chapman, A. Englezou, K. F. Armstrong, A. R. Clarke, D. Hailstones: Multi-gene phylogenetic analysis of south-east Asian pest members of the Bactrocera dorsalis species complex (Diptera: Tephritidae) does not support current taxonomy. In: Journal of Applied Entomology. 138, Nr. 4, 1. Mai 2014, ISSN 1439-0418, S. 235–253. doi:10.1111/jen.12047.
- Agnes Scheunert, Günther Heubl: Diversification of Scrophularia (Scrophulariaceae) in the Western Mediterranean and Macaronesia – Phylogenetic relationships, reticulate evolution and biogeographic patterns. In: Molecular Phylogenetics and Evolution. 70, 1. Januar 2014, S. 296–313. doi:10.1016/j.ympev.2013.09.023.
- Tao Yang, Tian-lei Zhang, You-hao Guo, Xing Liu: Identification of Hybrids in Potamogeton: Incongruence between Plastid and ITS Regions Solved by a Novel Barcoding Marker PHYB. In: PLOS ONE. 11, Nr. 11, 17. November 2016, ISSN 1932-6203, S. e0166177. doi:10.1371/journal.pone.0166177. PMID 27855191. PMC 5113904 (freier Volltext).