Bacillus subtilis
Bacillus subtilis (lat. bacillus, Stäbchen; subtilis, fein, schlicht) oder Heubazillus ist ein weitverbreitetes grampositives, stäbchenförmiges, begeißeltes Bodenbakterium.[2] Wie alle Bakterien der Gattung Bacillus ist B. subtilis ein fakultativ anaerob wachsender Endosporenbildner.
| Bacillus subtilis | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
Bacillus subtilis (Flagellen-Färbung) | ||||||||||||
| Systematik | ||||||||||||
| ||||||||||||
| Wissenschaftlicher Name | ||||||||||||
| Bacillus subtilis | ||||||||||||
| (Ehrenberg 1835) Cohn 1872[1] |
Merkmale
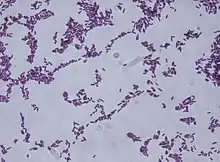

Der stäbchenförmige B. subtilis weist üblicherweise eine Größe von ca. 2 bis 3 µm auf, die Dicke liegt bei ca. 0,7 bis 0,8 µm. Seine Sporen sind elliptisch mit einer Länge von 1 bis 1,5 µm und einer Breite von 0,6 bis 0,9 µm.[3] Die Zellen sind peritrich, also mehrfach und über die ganze Zelle verteilt begeißelt und können sich dadurch schnell fortbewegen. Das Bakterium wird von einer grampositiven Zellwand umgeben.[4]
Lebensweise und Physiologie
B. subtilis ist ubiquitär verbreitet und kann aus Boden, Staub und Luft isoliert werden. Als Bodenkeim kommt er häufig auf Pflanzen und Pflanzenmaterial vor.[5]
Der Name Heubazillus weist darauf hin, dass er sich leicht in einem sogenannten Heuaufguss anreichern lässt. Die Generationszeit beträgt bei optimalem Nährstoffangebot, optimaler Sauerstoffversorgung und einer optimalen Wachstumstemperatur von 40 °C ca. 45 Minuten.[4]
B. subtilis ernährt sich chemoorgano-heterotroph, d. h., er nutzt von anderen Lebewesen erzeugte Nährstoffe, um Energie und körpereigene Substanz zu generieren. B. subtilis besiedelt sowohl die Rhizosphäre als auch die oberen Schichten des Bodens.[6] Dort hat er als typisches Fäulnisbakterium Anteil an der Rückführung organischer Stoffe in die Nahrungskreisläufe. Er besitzt ein großes Arsenal an Glukan- (polymer verkettete Zucker)[7] und Proteasen[8] (Protein-abbauenden Enzymen), die bei Bedarf aus der Zelle exportiert werden.
Als Kohlenstoff- und Energiequelle wird bevorzugt Traubenzucker (Glucose) genutzt. Bei ausreichender Konzentration verhindert Glucose die Aktivierung von Genen, deren Produkte andere Kohlenstoffquellen in den Stoffwechsel einschleusen. Bei Glucoseabwesenheit können auch andere Zucker oder kohlenstoffhaltige Substrate genutzt werden.(Katabolitrepression)[4]
Zur Energiegewinnung dient Sauerstoff als bevorzugter terminaler Elektronenakzeptor (Zellatmung). Auch hier wird die Nutzung alternativ in Frage kommender Substrate bei Sauerstoffzutritt unterdrückt. Unter anaeroben Bedingungen können die Zellen bei Glucose- und Nitrat-Anwesenheit noch genug Energie für langsames Wachstum erzeugen. Sind keine als Elektronenakzeptor nutzbaren Substrate verfügbar, dann ist B. subtilis in der Lage, auch ausschließlich durch Gärungsstoffwechsel bei Erzeugung von Milchsäure, Ethanol, Acetoin und 2,3-Butandiol zu überleben.[4]

Widrigen Umweltbedingungen versucht sich B. subtilis durch aktive Fortbewegung mithilfe seiner Geißel zu entziehen. Ferner kann sich B. subtilis über die sogenannte generelle Stressantwort als vegetativ aktive Zelle mit Schwankungen von Umweltfaktoren auseinandersetzen. In letzter Konsequenz kann B. subtilis durch ein atypisches Zellteilungsprogramm Endosporen bilden, die lange Perioden – allerdings unter Aufgabe der ökologischen Nische und Ausscheiden aus evolutionären Prozessen – überdauern. Im Lichtmikroskop sind Sporen bzw. Vorsporen in sporulierenden Zellen auch ohne Färbung als stark lichtbrechende, ovale Strukturen zu erkennen.
Eine weitere Eigenschaft ist die Ausbildung der Kompetenz. Kompetenz bei Bakterien heißt die Fähigkeit, extrazelluläre (Fremd)DNA aufzunehmen und diese zwecks Erweiterung des eigenen Genoms zu integrieren oder sie zur Ernährung zu nutzen.[9]
Systematik
Taxonomisch zählt man B. subtilis zu den Eubakterien (Bacteria), genauer zu den grampositiven Firmicutes. Dort wird er der Klasse der Bacilli zugeordnet (niedriger GC-Gehalt). Diese Klasse umfasst die Ordnung der Bacillales und der Lactobacillales (Milchsäurebakterien).[10] Zu der Ordnung der Bacillales gehört unter anderem die Familie der Bacillaceae mit der Gattung Bacillus, die etwa 150 Arten einschließt (Claus & Berkeley, 1986). Außerdem gehören auch die Familien der Staphylococcaceae und der Listeriaceae zu der Ordnung der Bacillales.[10] Die phylogenetische Nähe zu Pathogenen wie Staphylokokken oder Listerien macht B. subtilis für die molekularbiologische und -medizinische Forschung besonders interessant.
1999 erfolgte die Aufteilung in Unterarten (Subspezies), von denen zurzeit (Stand 2014) drei bekannt sind:[10]
- B. subtilis subsp. inaquosorum Rooney et al. 2009[11][12]
- B. subtilis subsp. spizizenii Nakamura et al. 1999[13]
- B. subtilis subsp. subtilis (Ehrenberg 1835) Nakamura et al. 1999[14][13]
B. subtilis wurde im Jahre 1835 durch Christian Gottfried Ehrenberg als Vibrio subtilis (Gekrümmtes Stäbchen) beschrieben. 1872 wurde es durch Ferdinand Julius Cohn in Bacillus subtilis (Stäbchen) umbenannt und von Fischer 1895 einer gleichnamigen Familie zugeordnet.[10]
Gruppe
Zur Bacillus subtilis Gruppe gehören Bacillus amyloliquefaciens, Bacillus atrophaeus, Bacillus mojavensis, Bacillus sonorensis, Bacillus tequilensis, Bacillus vallismortis und Bacillus velezensis. Aufgrund hoch konservierter DNA-Sequenzen innerhalb des Komplexes lassen sich diese nicht mit herkömmlicher 16S rRNA Sequenzierung unterscheiden.[12] Auch eine Differenzierung anhand physiologischer und biochemischer Tests ist nicht möglich. Mittels MALDI-TOF MS kann eine korrekte Klassifizierung erfolgen, wenn eine entsprechend spezialisierte Datenbank vorliegt.[15]
Bedeutung für den Menschen
In der Forschung dient B. subtilis als Modellorganismus zur Erforschung der Zellwand.[16]
B. subtilis wurde früher in der Humanmedizin zur Behandlung von chronischen Hautkrankheiten (Dermatosen) bzw. von Durchfall, Gärungs- und Fäulnisdyspepsien, Magen-Darm-Entzündung (Enteritis) und Enterocolitis angewandt. In der Roten Liste der in Deutschland verfügbaren Fertigarzneimittel von 2017 (Rote Liste, 2017) sind nur noch die homöopathischen Präparate Utilin und Utilin N ohne Indikation aufgeführt.
Aufgrund der hohen Hitzeresistenz der Sporen von B. subtilis werden diese auch als Indikator bei entsprechenden Sterilisationsprozessen in Pharmazie, Medizin und Lebensmittelindustrie eingesetzt.[17]
In der Landwirtschaft wird der B. subtilis Stamm QST 713 als biologisches Pflanzenschutzmittel kommerziell eingesetzt. Lipopeptide wirken fungizid und gegen bakterielle Pflanzenschädlinge. Sie sind im ökologischen Landbau zugelassen.[18]
Aufgrund seiner Fähigkeit zur Sekretion extrazellulärer Enzyme wird B. subtilis insbesondere für die Herstellung von Waschmittelenzymen (z. B. Subtilisin[19]), aber außerdem auch für die Synthese von Riboflavin[20] (Vitamin B2) und des Antibiotikums Bacitracin[21] in der biotechnologischen Industrie genutzt.
Der Stamm Bacillus subtilis var. natto wird zur Herstellung der japanischen Spezialität Nattō und ähnlichen Gerichten verwendet.[22]
B. subtilis kann unter äußerst seltenen Umständen auch pathogen wirken, z. B. kann es bei Augenverletzungen und Eindringen des Bakteriums zur Blindheit kommen (Panophthalmie).[23] Zudem kann B. subtilis das Toxin Amylosin bilden und zu Lebensmittelvergiftungen führen.[24]
Genetik
B. subtilis gilt als das bestuntersuchte grampositive Bakterium. In den Jahren 1990 bis 1997 wurde sein Genom erforscht und komplett sequenziert, wobei sich die Sequenzierungsstrategie an bereits vorhandenen Genkarten orientierte. Der zirkuläre DNA-Doppelstrang umfasst 4.214.810 Basenpaare; der GC-Gehalt liegt bei 43,5 %. Von der Gesamtsequenz haben 86,87 % der Nukleotide codierende Funktion, die restlichen Nukleotide befinden sich in z. T. regulatorisch bedeutsamen Regionen zwischen den Genen. Im Durchschnitt misst ein Gen 890 Nukleotide.[25]
Weblinks
Einzelnachweise
- Bacillus subtilis. doi:10.1601/nm.10618.
- Fuchs, Georg 1945-, Schlegel, Hans Günter 1924-2013, Eitinger, Thomas 1961-: Allgemeine Mikrobiologie. 8. Auflage. Thieme, Stuttgart New York 2007, ISBN 978-3-13-444608-1, S. 51.
- Grundlagen der Lebensmittelmikrobiologie: Eine Einführung. 6., verbesserteage Auflage. Steinkopff, Heidelberg 1986, ISBN 3-642-85350-1, S. 16.
- Antranikian, G. (Garabed): Angewandte Mikrobiologie. Springer-Verlag Berlin Heidelberg, Berlin, Heidelberg 2006, ISBN 3-540-29456-2, S. 14.
- Klein, Günter: Mikrothek: Bildatlas Mikrobiologie. Band 1. Behr, Hamburg 2007, ISBN 978-3-89947-372-8, II.17.1.
- Shoda, Makoto: Biocontrol of plant diseases by bacillus subtilis: basic and practical applications. CRC Press, Boca Raton, FL 2020, ISBN 978-0-429-63676-9, S. 4.
- Monika Wolf, Attila Geczi, Ortwin Simon, Rainer Borriss: Genes encoding xylan and β-glucan hydrolysing enzymes in Bacillus subtilis: characterization, mapping and construction of strains deficient in lichenase, cellulase and xylanase. In: Microbiology,. Band 141, Nr. 2, 1995, ISSN 1350-0872, S. 281–290, doi:10.1099/13500872-141-2-281.
- M. M. Kole, I. Draper, Donald F. Gerson: Production of protease by bacillus subtilis using simultaneous control of glucose and ammonium concentrations. In: Journal of Chemical Technology & Biotechnology. Band 41, Nr. 3, 1988, ISSN 1097-4660, S. 197–206, doi:10.1002/jctb.280410305.
- Patrícia H Brito, Bastien Chevreux, Cláudia R Serra, Ghislain Schyns, Adriano O Henriques, José B Pereira-Leal: Genetic Competence Drives Genome Diversity in Bacillus subtilis. In: Genome Biology and Evolution. Band 10, Nr. 1, 2017, ISSN 1759-6653, S. 108–124, doi:10.1093/gbe/evx270, PMID 29272410.
- Jean Euzéby, Aidan C. Parte: Genus Bacillus. In: List of Prokaryotic names with Standing in Nomenclature (LPSN). Abgerufen am 9. August 2014.
- Bacillus subtilis inaquosorum. doi:10.1601/nm.14741.
- Alejandro P. Rooney, Neil P. J. Price, Christopher Ehrhardt, James L. Swezey, Jason D. Bannan: Phylogeny and molecular taxonomy of the Bacillus subtilis species complex and description of Bacillus subtilis subsp. inaquosorum subsp. nov. In: International Journal of Systematic and Evolutionary Microbiology,. Band 59, Nr. 10, 2009, ISSN 1466-5026, S. 2429–2436, doi:10.1099/ijs.0.009126-0.
- L. K. Nakamura, Michael S. Roberts, Frederick M. Cohan: Note: Relationship of Bacillus subtilis clades associated with strains 168 and W23: A proposal for Bacillus subtilis subsp. subtilis subsp. nov. and Bacillus subtilis subsp. spizizenii subsp. nov. In: International Journal of Systematic and Evolutionary Microbiology,. Band 49, Nr. 3, 1999, ISSN 1466-5026, S. 1211–1215, doi:10.1099/00207713-49-3-1211.
- Taxonomy of the subspecies Bacillus subtilis subtilis (Ehrenberg 1835) Nakamura et al. 1999. doi:10.1601/tx.4858.
- Chien-Hsun Huang, Lina Huang, Mu-Tzu Chang, Kuo-Lung Chen: Establishment and application of an analytical in-house database (IHDB) for rapid discrimination of Bacillus subtilis group (BSG) using whole-cell MALDI-TOF MS technology. In: Molecular and Cellular Probes. Band 30, Nr. 5, 2016, ISSN 0890-8508, S. 312–319, doi:10.1016/j.mcp.2016.08.002.
- M. Leaver, P. Domínguez-Cuevas, J. M. Coxhead, R. A. Daniel, J. Errington: Life without a wall or division machine in Bacillus subtilis. In: Nature. Band 457, Nr. 7231, 2009, ISSN 1476-4687, S. 849–853, doi:10.1038/nature07742.
- T. E. Odlaug, R. A. Caputo, G. S. Graham: Heat Resistance and Population Stability of Lyophilized Bacillus subtilis Spores. In: Applied and Environmental Microbiology. Band 41, Nr. 6, 1981, ISSN 0099-2240, S. 1374–1377, doi:10.1128/AEM.41.6.1374-1377.1981, PMID 16345790.
- Zeller, Wolfgang: Proceedings of the 1st International Symposium on Biological Control of Bacterial Plant Diseases: Seeheim /Darmstadt, 23rd - 26th October 2005. Biologische Bundesanst. für Land- und Forstwirtschaft, Berlin Braunschweig 2006, ISBN 3-930037-29-7, S. 167 (researchgate.net [PDF]).
- Sara Lanigan‐Gerdes, Alek N. Dooley, Kym F. Faull, Beth A. Lazazzera: Identification of subtilisin, Epr and Vpr as enzymes that produce CSF, an extracellular signalling peptide of Bacillus subtilis. In: Molecular Microbiology. Band 65, Nr. 5, 2007, ISSN 1365-2958, S. 1321–1333, doi:10.1111/j.1365-2958.2007.05869.x.
- J B Perkins, A. Sloma, T. Hermann, K. Theriault, E. Zachgo, T. Erdenberger, N. Hannett, N P Chatterjee, V. Williams II, GA Rufo Jr, R. Hatch, J. Pero: Genetic engineering of Bacillus subtilis for the commercial production of riboflavin. In: Journal of Industrial Microbiology and Biotechnology. Band 22, Nr. 1, 1999, ISSN 1476-5535, S. 8–18, doi:10.1038/sj.jim.2900587.
- Balbina A. Johnson, Herbert Anker, Frank L. Meleney: Bacitracin: A New Antibiotic Produced by a Member of the B. Subtilis Group. In: Science. Band 102, Nr. 2650, 1945, ISSN 0036-8075, S. 376–377, doi:10.1126/science.102.2650.376, PMID 17770204.
- Masahito Hitosugi, Katsuo Hamada, Kazutaka Misaka: Effects of Bacillus subtilis var. natto products on symptoms caused by blood flow disturbance in female patients with lifestyle diseases. In: International Journal of General Medicine. Band 8, 2015, ISSN 1178-7074, S. 41–46, doi:10.2147/IJGM.S76588, PMID 25653551.
- Werner Köhler (Hrsg.): Medizinische Mikrobiologie. 8. Auflage. München/Jena 2001, ISBN 978-3-437-41640-8.
- C. Apetroaie-Constantin, R. Mikkola, M.A. Andersson, V. Teplova, I. Suominen, T. Johansson, M. Salkinoja-Salonen: Bacillus subtilis and B. mojavensis strains connected to food poisoning produce the heat stable toxin amylosin. In: Journal of Applied Microbiology. Band 106, Nr. 6, 2009, S. 1976–1985, doi:10.1111/j.1365-2672.2009.04167.x.
- F. Kunst, N. Ogasawara, I. Moszer, A. M. Albertini, G. Alloni, V. Azevedo, M. G. Bertero, P. Bessières, A. Bolotin, S. Borchert, R. Borriss, L. Boursier, A. Brans, M. Braun, S. C. Brignell, S. Bron, S. Brouillet, C. V. Bruschi, B. Caldwell, V. Capuano, N. M. Carter, S.-K. Choi, J.-J. Codani, I. F. Connerton, N. J. Cummings, R. A. Daniel, F. Denizot, K. M. Devine, A. Düsterhöft, S. D. Ehrlich, P. T. Emmerson, K. D. Entian, J. Errington, C. Fabret, E. Ferrari, D. Foulger, C. Fritz, M. Fujita, Y. Fujita, S. Fuma, A. Galizzi, N. Galleron, S.-Y. Ghim, P. Glaser, A. Goffeau, E. J. Golightly, G. Grandi, G. Guiseppi, B. J. Guy, K. Haga, J. Haiech, C. R. Harwood, A. Hénaut, H. Hilbert, S. Holsappel, S. Hosono, M.-F. Hullo, M. Itaya, L. Jones, B. Joris, D. Karamata, Y. Kasahara, M. Klaerr-Blanchard, C. Klein, Y. Kobayashi, P. Koetter, G. Koningstein, S. Krogh, M. Kumano, K. Kurita, A. Lapidus, S. Lardinois, J. Lauber, V. Lazarevic, S.-M. Lee, A. Levine, H. Liu, S. Masuda, C. Mauël, C. Médigue, N. Medina, R. P. Mellado, M. Mizuno, D. Moestl, S. Nakai, M. Noback, D. Noone, M. O'Reilly, K. Ogawa, A. Ogiwara, B. Oudega, S.-H. Park, V. Parro, T. M. Pohl, D. Portetelle, S. Porwollik, A. M. Prescott, E. Presecan, P. Pujic, B. Purnelle, G. Rapoport, M. Rey, S. Reynolds, M. Rieger, C. Rivolta, E. Rocha, B. Roche, M. Rose, Y. Sadaie, T. Sato, E. Scanlan, S. Schleich, R. Schroeter, F. Scoffone, J. Sekiguchi, A. Sekowska, S. J. Seror, P. Serror, B.-S. Shin, B. Soldo, A. Sorokin, E. Tacconi, T. Takagi, H. Takahashi, K. Takemaru, M. Takeuchi, A. Tamakoshi, T. Tanaka, P. Terpstra, A. Tognoni, V. Tosato, S. Uchiyama, M. Vandenbol, F. Vannier, A. Vassarotti, A. Viari, R. Wambutt, E. Wedler, H. Wedler, T. Weitzenegger, P. Winters, A. Wipat, H. Yamamoto, K. Yamane, K. Yasumoto, K. Yata, K. Yoshida, H.-F. Yoshikawa, E. Zumstein, H. Yoshikawa, A. Danchin: The complete genome sequence of the Gram-positive bacterium Bacillus subtilis. In: Nature. Band 390, Nr. 6657, 1997, ISSN 1476-4687, S. 249–256, doi:10.1038/36786.