Antisense-RNA
Die Antisense-RNA (aRNA), auch natürliches Antisense Transkript (NAT) genannt,[1] ist eine einzelsträngige RNA, die komplementär zu einer proteincodierenden Messenger-RNA (mRNA) ist. Man ordnet die aRNA den lncRNAs bzw. ncRNAs zu.[2] Sie spielt in Zellen bei Regulationsprozessen eine wichtige Rolle[3], wird aber auch in der Forschung immer häufiger als Tool für einen Gene-Knockdown eingesetzt.[4]
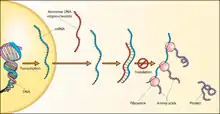
Struktur
Die Struktur von aRNA ist naturgemäß der von mRNA ähnlich. Allerdings weisen fast alle aRNAs Sekundärstrukturen, wie stem-loops und teilweise auch komplexere Tertiärstrukturen, wie Pseudoknoten zwischen ebendiesen Sekundärstrukutren auf. Diese Strukturelemente bestimmen die Abbaurate durch intrazelluläre Ribonukleasen und auch die Rate mit welcher sich die aRNA mit der komplementären RNA paart.[5][6]
Wirkungsweg
Die aRNA kann auf verschiedene Arten und Weisen wirken. Zumeist wirkt sie, indem sie die Translation eines Gens unterbindet. Sie kann aber auch epigenetisch oder aktivierend wirken.
Translation
Die mRNA wird vom Matrizenstrang der DNA transkribiert. Der Nicht-Matrizenstrang ist somit der codogene Strang. Wird auch dieser – der zum Matrizenstrang komplementäre Strang – transkribiert, entsteht eine zur mRNA komplementäre aRNA. Die aRNA inhibiert durch Basenpaarung mit der komplementären mRNA deren Translation in der Zelle.
Ein alternativer Wirkungsweg ist die Bindung der aRNA an die Bindestellen von miRNA und die damit einhergehende Stabilisierung der RNA. Es wird vermutet, dass dieser Mechanismus auch bei Alzheimer eine Rolle spielt.[7][8] Damit wird die Genexpression einzelner Gene reguliert.
Epigenetik
Es ist bekannt, dass aRNA auch epigenetisch wirken kann. Hierbei sind zumeist längere aRNAs zu beobachten.[9] Dies kann zum Beispiel durch die von aRNA induzierte Methylierung von CpG-Islands im Genom verursacht werden. Ein solcher Effekt wurde zum Beispiel im Zusammenhang mit der Erkrankung α-Thalassämie[10] oder dem Silencing von X-Chromosomen[11] nachgewiesen.
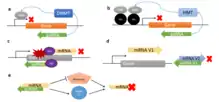
Aktivierung
Es ist auch möglich, dass durch aRNA ein aktivierender Effekt hervorgerufen wird.[12] Dies wird zum Beispiel erzielt, indem die aRNA an die RNA einer Hairpinstruktur bindet und so eine in dieser verborgene Shine-Dalgarno-Sequenz freilegt und so die Translation ermöglicht.
Klassifikation
Antisense-RNA kann sowohl ein cis- als auch trans-wirkendes Element darstellen. In ersterem Fall wird die aRNA vom komplementären DNA-Strang transkribiert. So hat man häufig einen sehr hohen Grad oder eine komplette Komplementarität und die aRNA besitzt nur eine einzige Ziel-RNA.[6][13] In letzterem Fall stammt die aRNA von einem weiter entfernten Gen. Sie zeigen meistens einen geringere Grad an Komplementarität, weshalb die gebildeten Komplexe instabiler sind und teilweise Chaperone zur Stabilisierung des Komplexes erforderlich sind und mehrere RNAs das Ziel sein können.[14]
Neben dieser Einteilung kann eine Einteilung auch entweder nach dem Typ der Interaktion (RNA-DNA, RNA-RNA oder RNA-Protein), der Länge der aRNA (die Grenze wird hier bei 100 bp gezogen) oder nach dem Typ des betroffenen Promotors erfolgen.[15][16] Zuletzt wird auch teilweise eine Klassifikation nach der Halbwertszeit der aRNA in der Zelle vorgenommen, auch wenn die Bedeutung dieser der der Paarungsrate zwischen aRNA und mRNA untergeordnet ist.[5]
Verwendung & Vorkommen
Antisense-RNA stellt eine natürliche Möglichkeit der Genregulation der Proteinbiosynthese dar. Beim Menschen gibt es mindestens 1600 Antisense-Gene, beispielsweise der Insulin-like growth factor 2 receptor (IGF-2). Bei diesem Gen kann, abhängig von genetischer Prägung, ein zweiter Promotor am 3'-Ende des Gens aktiv sein, über den Antisense-RNA transkribiert wird. Diese verhindert in der Folge die Translation beider Allele dieses Gens. Der Phänotyp folgt also nicht der mendelschen Vererbungslehre. Antisense-Transkript treten bei mehr als 70 % der Gene in cDNA-Datenbanken (Fantom-3, GenBank) auf.[17]
Eingesetzt wird Antisense-RNA zum Beispiel in der Biotechnologie, beispielsweise bei der kommerziell wenig erfolgreichen Flavr-Savr-Tomate. Hier wurde ein künstliches Gen in die Tomate eingebracht, das Antisense-RNA gegen ein am Reifungsprozess beteiligtes Gen produziert, das für das Enzym Polygalacturonase codiert. Hierdurch kann der Reifungsprozess der sogenannten Flavr-Savr-Tomate verzögert werden.[18] Ein weiteres Beispiel ist die Kartoffelsorte Amflora, bei der die Technik angewendet wurde, um die Amylose-Produktion der Kartoffel zu unterdrücken.[19]
Neben dem Einsatz in der Biotechnologie findet das System der Antisense-RNA zunehmend Verbreitung in der Medizin und Pharmakologie. Das erste Medikament, das auf der Antisense-Technik beruht und für den Verkehr zugelassen wurde, ist das Virostatikum Fomivirsen gegen das Cytomegalievirus. Für die Entwicklung von Antisense-RNA Medikamenten erhielten C. Frank Bennett (neurodegenerative Erkrankungen) und Adrian R. Krainer (bei Spinaler Muskelatrophie) für 2019 den Breakthrough Prize in Life Sciences.
Bei solchen therapeutischen Ansätzen wird zumeist versucht ein Gen durch aRNA stillzulegen. Hierbei gibt es zwei Ansätze. Zum einen kann eine aRNA für eine Art Anti-Gen erzeugt, dass dann an die mRNA des Zielgens bindet. Alternativ kann eine 15 bis 20 bp lange aRNA genutzt werden, die eine spezifische Sequenz ansteuert, was meistens bereits ausreichend ist. Eine Möglichkeit ist hierbei das 5'-Ende der mRNA zu attackieren und so eine Translation komplett zu unterbinden. Genauso gut kann an eine beliebige andere Stelle innerhalb der mRNA gebunden werden, was zumeist dieselbe Effektivität hat.[20]
In der Molekular- und Zellbiologie wird in vitro erzeugte Antisense-RNA für In-situ-Hybridisierungen eingesetzt.
Literatur
- Farzaneh Modarresi, Mohammad Ali Faghihi, Miguel A Lopez-Toledano, Roya Pedram Fatemi, Marco Magistri: Inhibition of natural antisense transcripts in vivo results in gene-specific transcriptional upregulation. In: Nature Biotechnology. Band 30, Nr. 5, Mai 2012, ISSN 1087-0156, S. 453–459, doi:10.1038/nbt.2158, PMID 22446693, PMC 4144683 (freier Volltext).
- Jian-zhong Xu, Jun-lan Zhang, Wei-guo Zhang: Antisense RNA: the new favorite in genetic research. In: Journal of Zhejiang University-SCIENCE B. Band 19, Nr. 10, Oktober 2018, ISSN 1673-1581, S. 739–749, doi:10.1631/jzus.B1700594, PMID 30269442, PMC 6194357 (freier Volltext).
- Yutaka Eguchi, Tateo Itoh, Jun-ichi Tomizawa: Antisense RNA. In: Annual Review of Biochemistry. Band 60, Nr. 1, Juni 1991, ISSN 0066-4154, S. 631–652, doi:10.1146/annurev.bi.60.070191.003215.
- João F. Menino, Agostinho J. Almeida, Fernando Rodrigues: Gene Knockdown in Paracoccidioides brasiliensis Using Antisense RNA. In: Host-Fungus Interactions. Band 845. Humana Press, Totowa, NJ 2012, ISBN 978-1-61779-538-1, S. 187–198, doi:10.1007/978-1-61779-539-8_12.
- R.W. Simons: Antisense RNA. In: Encyclopedia of Genetics. Elsevier, 2001, ISBN 0-12-227080-0, S. 83–84, doi:10.1006/rwgn.2001.0061 (elsevier.com [abgerufen am 21. Oktober 2019]).
- E Wagner, S Altuvia, P Romby: 12 Antisense RNAs in bacteria and their genetic elements. In: Advances in Genetics. Band 46. Elsevier, 2002, ISBN 0-12-017646-7, S. 361–398, doi:10.1016/s0065-2660(02)46013-0.
- Mohammad Ali Faghihi, Farzaneh Modarresi, Ahmad M. Khalil, Douglas E. Wood, Barbara G. Sahagan: Expression of a noncoding RNA is elevated in Alzheimer's disease and drives rapid feed-forward regulation of beta-secretase. In: Nature Medicine. Band 14, Nr. 7, Juli 2008, ISSN 1546-170X, S. 723–730, doi:10.1038/nm1784, PMID 18587408, PMC 2826895 (freier Volltext).
- Mohammad Ali Faghihi, Ming Zhang, Jia Huang, Farzaneh Modarresi, Marcel P. Van der Brug: Evidence for natural antisense transcript-mediated inhibition of microRNA function. In: Genome Biology. Band 11, Nr. 5, 2010, ISSN 1474-760X, S. R56, doi:10.1186/gb-2010-11-5-r56, PMID 20507594, PMC 2898074 (freier Volltext).
- Kevin V. Morris, Peter K. Vogt: Long antisense non-coding RNAs and their role in transcription and oncogenesis. In: Cell Cycle. Band 9, Nr. 13, Juli 2010, ISSN 1538-4101, S. 2544–2547, doi:10.4161/cc.9.13.12145, PMID 20581457, PMC 3040850 (freier Volltext).
- Cristina Tufarelli, Jackie A Sloane Stanley, David Garrick, Jackie A Sharpe, Helena Ayyub: Transcription of antisense RNA leading to gene silencing and methylation as a novel cause of human genetic disease. In: Nature Genetics. Band 34, Nr. 2, Juni 2003, ISSN 1061-4036, S. 157–165, doi:10.1038/ng1157.
- Jeannie Lee, Lance S Davidow, David Warshawsky: Tsix, a gene antisense to Xist at the X-inactivation centre. In: Nature Genetics. Band 21, Nr. 4, April 1999, ISSN 1061-4036, S. 400–404, doi:10.1038/7734 (nature.com [abgerufen am 22. Oktober 2019]).
- Vicent Pelechano, Lars M. Steinmetz: Gene regulation by antisense transcription. In: Nature Reviews Genetics. Band 14, Nr. 12, Dezember 2013, ISSN 1471-0056, S. 880–893, doi:10.1038/nrg3594.
- Koji Numata, Yuki Okada, Rintaro Saito, Hidenori Kiyosawa, Akio Kanai: Comparative analysis of cis-encoded antisense RNAs in eukaryotes. In: Gene. Band 392, Nr. 1-2, Mai 2007, S. 134–141, doi:10.1016/j.gene.2006.12.005 (elsevier.com [abgerufen am 20. Oktober 2019]).
- Fatemeh Saberi, Mehdi Kamali, Ali Najafi, Alavieh Yazdanparast, Mehrdad Moosazadeh Moghaddam: Natural antisense RNAs as mRNA regulatory elements in bacteria: a review on function and applications. In: Cellular & Molecular Biology Letters. Band 21, Nr. 1, Dezember 2016, ISSN 1425-8153, S. 6, doi:10.1186/s11658-016-0007-z, PMID 28536609, PMC 5415839 (freier Volltext).
- Marco Magistri, Mohammad Ali Faghihi, Georges St Laurent, Claes Wahlestedt: Regulation of chromatin structure by long noncoding RNAs: focus on natural antisense transcripts. In: Trends in Genetics. Band 28, Nr. 8, August 2012, S. 389–396, doi:10.1016/j.tig.2012.03.013, PMID 22541732, PMC 3768148 (freier Volltext) – (elsevier.com [abgerufen am 20. Oktober 2019]).
- S. K. Ballas, W. C. Sherwood: Rapid in vivo destruction of Yt(a+) erythrocytes in a recipient with anti-Yta. In: Transfusion. Band 17, Nr. 1, Januar 1977, ISSN 0041-1132, S. 65–66, doi:10.1046/j.1537-2995.1977.17177128889.x, PMID 841677.
- RIKEN Genome Exploration Research Group and Genome Science Group (Genome Network Project Core Group) and the FANTOM Consortium: Antisense Transcription in the Mammalian Transcriptome. In: Science. Band 309, Nr. 5740, 2. September 2005, ISSN 0036-8075, S. 1564–1566, doi:10.1126/science.1112009.
- R. A. Sanders, W. Hiatt: Tomato transgene structure and silencing. In: Nat. Biotechnol. Bd. 23, 2005, S. 287–289. PMID 15765076
- BASF: Amflora (Seite nicht mehr abrufbar, Suche in Webarchiven) Info: Der Link wurde automatisch als defekt markiert. Bitte prüfe den Link gemäß Anleitung und entferne dann diesen Hinweis.
- David P. Clark, Nanette J. Pazdernik: Inherited Defects and Gene Therapy. In: Biotechnology. Elsevier, 2016, ISBN 978-0-12-385015-7, S. 523–564, doi:10.1016/b978-0-12-385015-7.00017-x.
Siehe auch
Weblinks
- Hamburger Bildungsserver: Ein gentechnisch verändertes Lebensmittel: Die Flavr Savr-Tomate
- www.transgen.de: Veränderte Pflanzenentwicklung: Ausgereift - und dennoch länger haltbar. (Memento vom 10. Dezember 2008 im Internet Archive) vom 10. Dezember 2008.