Attenuation (Genexpression)
Als transkriptionelle Attenuation wird eine Form der Regulation der Genexpression bei Prokaryoten bezeichnet, mit der eine begonnene Transkription vorzeitig abgebrochen werden kann.[1]
Der Transkriptionsvorgang wird hierbei beendet, indem die transkribierende RNA-Polymerase von der DNA-Vorlage getrennt wird, wenn infolge von Wechselwirkungen innerhalb des soeben aufgebauten mRNA-Anfangsbereichs durch interne Basenpaarung eine Haarnadelstruktur gebildet wird an der Terminatorstelle, dem Attenuator. Allerdings ist diese terminierende Schleifenbildung in der Sekundärstruktur der mRNA nicht möglich bei bestimmten Positionen eines gleichzeitig voranschreitenden und die entstehende mRNA translatierenden Ribosoms. Diese Positionen nimmt das Ribosom aber nur ein bei einer verzögerten Translation, erst deren Verzögerung erlaubt somit die Fortsetzung der Transkription über den Attenuatorbereich hinaus. Verzögert wird der Translationsprozess etwa, wenn nicht prompt eine mit der spezifischen Aminosäure beladene tRNA verfügbar ist, da die betreffende Aminosäure nur in geringer Konzentration in der Zelle vorliegt. Unter solchen Bedingungen kann dann eine vollständige Transkription erfolgen – beispielsweise von Genen für die Synthese einer Aminosäure, wenn es an dieser mangelt.
Voraussetzung für diesen Regulationsmechanismus ist, dass die Vorgänge von Transkription und Translation gemeinsam und nahezu synchron stattfinden, dass also die RNA-Polymerase noch transkribiert und das mRNA-Molekül aufbaut, während bereits ein Ribosom an anderer Stelle desselben mRNA-Moleküls sitzt und gleichzeitig translatiert. Diese zeitliche und räumliche Kopplung ist nur bei Prokaryoten gegeben. Denn bei eukaryoten Zellen findet die Transkription im Zellkompartiment des Zellkerns statt, anschließend wird die mRNA aus dem Karyoplasma exportiert und die Translation läuft außerhalb des Kerns im Zytoplasma ab, zeitlich und räumlich getrennt.
Beispiel: Tryptophan-Operon in Escherichia coli
Das trp-Operon in Bakterien wie Escherichia coli ist ein klassisches Beispiel der transkriptionellen Attenuation als einer zusätzlichen Genregulationsweise, erstmals 1981 von Charles Yanofsky beschrieben.[2] Doch vorrangig wird auch bei E. coli der Zugriff auf das Operon durch Repression geregelt. Ein trpR-Gen codiert für ein Repressorprotein, das reversibel an einen im Promotorbereich (P) gelegenen Operator (O) bindet und damit der RNA-Polymerase den Zugang verlegt,[3] wenn es zuvor durch die Aminosäure Tryptophan (Trp) als Corepressor aktiviert wurde. Derart wird die Transkription abhängig vom intrazellulären Tryptophan-Spiegel negativ rückgekoppelt reguliert und mit ansteigendem Spiegel unterdrückt. Bei hoher Tryptophankonzentration ist das trp-Operon daher mit sehr hoher Wahrscheinlichkeit reprimiert und eine Transkription findet kaum statt.
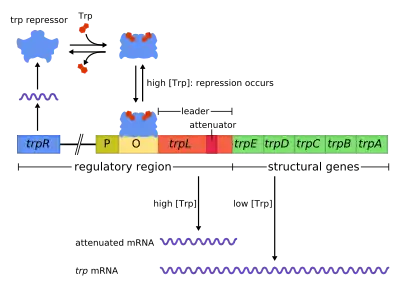
Kommt dennoch eine Transkription in Gang, so stellt die Attenuation zunächst einen zusätzlich sichernden Mechanismus dar, mit dem bei hohem Spiegel an mit Tryptophan beladener tRNA (tRNATrp) ein vorzeitiger Abbruch erzwungen werden kann – noch bevor die nachfolgenden Strukturgene des Operons transkribiert werden. Diese aufeinanderfolgenden Gene (trpE, trpG-D, trpC-F, trpB und trpA)[4] codieren für sieben Polypeptide, die in enzymatisch wirksamen Proteinen für die Biosynthese von Tryptophan gebraucht werden – falls an dieser Aminosäure ein Mangel besteht. Noch vor den offenen Leserahmen dieser Gene liegt nun ein sogenannter untranslatierter Bereich, die 5'-UTR oder Leader-Sequenz, mit jenen Sequenzen, die während der Bildung der mRNA eine Attenuation möglich machen.[3]
In diesem rund 160 Nukleotide umfassenden Bereich am 5'-Anfang der mRNA finden sich nämlich vier kürzere palindromische Sequenzen: als je etwa ein Dutzend Nukleotide lange einsträngige Sequenzabfolgen (1., 2., 3., 4.), die einander ergänzen können und so durch Basenpaarungen miteinander jeweils doppelsträngige Teilabschnitte einer Haarnadelstruktur in der mRNA auszubilden vermögen. Dabei sind verschiedene Stamm-Schleifen-Bildungen möglich (1:2, 2:3, 3:4) mit zueinander komplementären Palindromen. Eine Ausbildung der hinteren (3:4) mRNA-Schleife führt – wegen der schlaufennah starken Bindungen über Wasserstoffbrücken (GCGGGC... in 4.) – zum Anhalten des RNA-Polymerase-Moleküls, und – wegen der anschließend schwachen Bindungen (...UUUUUU) – zum Ablösen vom DNA-Strang, womit die Transkription vorzeitig beendet ist, noch bevor das erste Gen des Operons (trpE) erreicht wurde. Die für diese Rho-Faktor-unabhängige Termination nötige strukturtragende Sequenz wird als das (intrinsische) Terminationssignal der Attenuatorsequenz bezeichnet.
Die davorliegenden Sequenzen erlauben innerhalb des entstehenden mRNA-Transkripts eine alternative Schleifenbildung, womit der vorzeitige Transkriptionsabbruch unterlaufen werden kann. Denn wird die Sequenz 2 mit 3 gepaart (2:3), ist die Paarung von 3 mit 4 nicht möglich. Zur Bildung dieser Schleifenstruktur 2:3 kommt es jedoch erst bei einem deutlich verzögerten Aufenthalt des Ribosoms in Sequenz 1, das zunächst synchronisiert[Anm. 1] der RNA-Polymerase nachfolgt.[4]
Diese entscheidende Verzögerung kann auftreten, da die Sequenz 1 zugleich auch das Ende eines anderen regulatorischen Abschnitts darstellt, der trpL-Sequenz, eines – in einer 5'-UT-Region eher ungewöhnlichen – Abschnitts mit 14 Codons. Deren fünf letzte vor dem Stopcodon, darunter zweimal das Codon für Tryptophan, liegen in Sequenz 1. Das Ribosom nun translatiert diese codierende Nukleotidsequenz trpL der mRNA in ein sogenanntes Leader-Peptid,[Anm. 2] und bleibt hier dann etwas länger hängen, wenn beladene tRNATrp für das Trp-Tandem nicht prompt verfügbar ist, so etwa bei Tryptophan-Mangel im Zytosol. Damit aber wird zugleich Sequenz 1 als möglicher Partner für Sequenz 2 entzogen, die sich sodann mit 3 verkuppelt (2:3), womit 3:4 nicht mehr geht. Die vorzeitige Termination der Transkription wird so unterlaufen.[3]
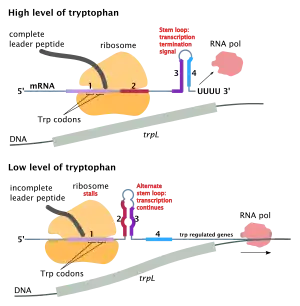
Bei niedrigem Trp-Spiegel wird das Leader-Peptid in Region 1 verzögert gebildet, wodurch die Struktur 2:3 möglich wird und so die vollständige Transkription.
Liegt dagegen genug Tryptophan vor, so kann ein Ribosom diesen Bereich der Leader-Sequenz rasch translatieren und der Polymerase zügig folgend über Sequenz 2 nachziehen. Es bildet sich dann keine Stamm-Schleifen-Struktur 2:3 aus; die Sequenz 3 kann daher mit 4, sobald vorliegend, die Struktur 3:4 bilden: das Terminationssignal für die RNA-Polymerase unterbricht die Transkription (und die Gene trpE bis trpA werden nicht transkribiert).
Ist zu wenig Tryptophan vorhanden, braucht das Ribosom zur Translation der Sequenz 1 in das Leader-Peptid deutlich mehr Zeit. Die verzögerte Translation führt zur Stamm-Schleifen-Struktur 2:3 im stromabwärts entstandenen Transkript, sie erlaubt damit die vollständige Transkription sämtlicher Gene trpE bis trpA durch die RNA-Polymerase in eine rund 6800 Nukleotide lange polycistronische mRNA – und ermöglicht somit die anschließende Translation dieser mRNA durch Ribosome in die Genprodukte TrpE bis TrpA: als enzymatisch wirksame Proteine machen sie eine gesteigerte Tryptophan-Synthese möglich, bei Tryptophanmangel vorteilhaft.
Ob es denn zur Attenuation kommt oder der Polymerase nicht der Strang entzogen wird, entscheidet sich also auf der Wanderung des ersten Ribosoms durch den Raum der Faltungsmöglichkeiten am Anfang der naszierenden mRNA. Die ungewöhnliche Aufgabe, im sogenannten untranslatierten Bereich ein kurzes tryptophanhaltiges Peptid aufzubauen, wird dabei zum Kriterium für den Transkriptionsverlauf und macht die Expression der trp-Gene von der aktuellen Trp-Konzentration im Zellmilieu abhängig. Nur bei niedrigem Spiegel kommt es dann zu jener kurzen Pause, mit der eine erfolgreiche Transkription erst möglich wird.
Weitere Beispiele transkriptioneller Attenuation
Inzwischen sind eine Reihe weiterer Operons bekannt, die transkriptionelle Attenuation als Regulationsweise nutzen. Neben Operons für die Aminosäurenbiosynthese beispielsweise von Phenylalanin, Histidin, Leucin, Threonin, Methionin oder Cystein ebenfalls solche für eine Aminosäurendegradation, und auch einige mit Abwandlungen des oben beschriebenen Attenuationmechanismus.[5]
So enthält etwa das tna-Operon in E. coli jene Gene, die für Enzyme des Tryptophanabbaus (Tryptophanase) codieren, und vornehmlich bei Tryptophanüberschuss in der Zelle transkribiert werden. Unter diesen Bedingungen unterbindet hier ein translatiertes tryptophanhaltiges Leader-Peptid die vorzeitige – von einem Rho-Faktor abhängige – Termination, indem es dann dafür nötige Bindungsstellen in der Leader-Region blockiert.[6]
Die Expression des Operons pyrBI in E. coli, das für ein Enzym der Biosynthese von Pyrimidin codiert, wird primär durch Attenuation reguliert. Hier ist es die Polymerase, die bei geringer Verfügbarkeit eines Pyrimidin-triphosphats (UTP) in einer bestimmten Region der Leadersequenz pausiert, und darüber auf Stellung und Bewegung des Ribosoms derart Einfluss nimmt, dass dieses die Bildung des Terminatorsignals verhindert.[7] Beim pyr-Operon in Bacillus subtilis ist es ein besonderes RNA-bindendes Protein, PyrR, das durch ein Pyrimidin-monophosphat (UMP) aktiviert wird und dann so an die mRNA binden kann, dass der intrinsische Terminator entsteht; bei niedrigem UMP-Spiegel bleibt es dagegen inaktiv, sodass eine alternative mRNA-Struktur gefaltet werden kann, die eine vollständige Transkription möglich macht.[8]
Beim trp-Operon in B. subtilis wird die Expression seiner sechs Strukturgene trpEDCFBA durch ein typtophan-aktiviertes RNA-bindendes Protein (TRAP) geregelt, das in der Leaderregion an die alternative Struktursequenz bindet, darüber mittelbar die Bildung des Terminatorsignals veranlasst und so zur transkriptionellen Attenuation führt. Daneben kann über hundert Nukleotide weiter stromabwärts noch eine Umfaltung der mRNA bewirkt werden, mit der die Ribosomen-Bindungsstelle vor trpE in eine Haarnadelstruktur eingelagert wird, sodass die Initiation der Translation dieses Gens blockiert ist. TRAP ist ein torusförmiger Komplex aus 11 identischen Proteinuntereinheiten, die je zwischen sich eine Nische für die Tryptophan-Bindung bilden. Sind diese besetzt, kann sich peripher um die Außenfläche des Gebildes herum einem Reifen ähnlich die mRNA-Sequenz legen, welche 11 (U/G)AG-Tripletts in Serie mit 2/3 Nukleotiden Abstand enthält, und wird von 11 KKR-Motiven (Lys-Lys-Arg) gegenüber erkannt.[4]
Die transkriptionelle Attenuation ist also eine nicht ungewöhnliche Form der Regulation der Genexpression in prokaryoten Zellen, bei der alternative Faltungsmuster im soeben gebildeten Anfangsbereich einer mRNA die Möglichkeit bieten, je nach den aktuell herrschenden metabolischen Bedingungen in der Zelle eine begonnene Transkription vorzeitig abzubrechen – noch bevor Strukturgene in mRNA umgeschrieben wurden – oder nicht.[5]
Charakteristika der Regulation über Attenuation
Die Möglichkeit der Attenuation stellt neben der Repression eine zusätzliche Regulationsweise der Genexpression dar. Inzwischen sind verschiedene Abwandlungen bekannt, mit denen sie realisiert werden kann.
Beim trp-Operon in E. coli liegen die Grenzen des Regelbereichs für die in Abhängigkeit von der zellulären Tryptophan-Konzentration exprimierten Strukturgene mit vorliegendem Repressor um etwa den Faktor 700 auseinander. Auch bei ausgeschaltetem Repressorgen trpR ist allein über Attenuation noch eine tryptophangeregelte Dämpfung der Genexpression um etwa den Faktor 10 möglich.
Voraussetzungen für diese Attenuation sind
- simultane Transkription und Translation mit
- synchronisierter Prozession von RNA-Polymerase und Ribosom
- bei unterschiedlichen Faltungsmöglichkeiten der naszierenden mRNA
- innerhalb des stromaufwärts der Strukturgene gelegenen Bereichs der 5'-UTR
- mit Wechselwirkung zwischen soeben entstehender Region und zuvor entstandenen,
- womit intrinsisch eine Strukturbildung möglich wird, die als ein Terminationssignal wirkt,
- zu der jedoch alternative Strukturbildungen möglich sind, die ebendieses ausschließen,
- wobei daneben weitere regulatorische Regionen oder regulierende Elemente vorliegen,
- mittels derer auf die Wahl zwischen den Alternativen entscheidend Einfluss genommen wird,
- und dies nun in Abhängigkeit von den aktuell herrschenden metabolischen Bedingungen,
- wobei dann vorrangig die Veränderlichkeit einer solchen Größe eine Rolle spielt,
- die zum Genprodukt oder dessen Wirkung in rückgekoppeltem Bezug steht.
Anmerkungen
- Die Prozession der transkribierenden RNA-Polymerase mit dem translatierenden Ribosom zu synchronisieren gelingt, indem die Polymerase nach Bildung der signalisierenden mRNA-Struktur 1:2 pausiert – und wartet, bis das erste Ribosom mit einer Translation beginnt, wobei diese Haarnadelstruktur dann aufgelöst wird.
- Von den Aminosäuren dieses Peptids mit der Aminosäuresequenz MKAIFVLKGWWRTS stop (siehe LPW ECOLI in UniProt-Datenbank) ist ein Siebtel somit Tryptophan (W). Doch üblicherweise ist diese Aminosäure auch bei E. coli ein recht selten gebrauchter Baustein in Proteinen, nur rund jeder hundertste.
Einzelnachweise
- Wilfried Janning, Elisabeth Knust: Genetik: Allgemeine Genetik – Molekulare Genetik – Entwicklungsgenetik. 2. Auflage. Georg Thieme, Stuttgart 2008, ISBN 978-3-13-151422-6, S. 211 ff.
- Charles Yanofsky: Attenuation in the control of expression of bacterial operons. In: Nature. 289, Nr. 5800, Februar 1981, S. 751–758. PMID 7007895.
- C. Yanofsky, T. Platt, I. Crawford, B. Nichols, G. Christie, H. Horowitz, M. Vancleemput, A. Wu: The complete nucleotide sequence of the tryptophan operon of Escherichia coli. In: Nucleic Acids Res.. 9, Nr. 24, Dezember 1981, S. 6647–6668. PMC 327632 (freier Volltext).
- C. Yanofsky: RNA-based regulation of genes of tryptophan synthesis and degradation, in bacteria. In: RNA. 13, Nr. 8, August 2007, S. 1141–1154. PMC 1924887 (freier Volltext).
- C. Yanofsky: Transcription Attenuation: Once Viewed as a Novel Regulatory Strategy. In: Journal of Bacteriology. 182, Nr. 1, Januar 2000, S. 1–8. PMC 94232 (freier Volltext).
- K. Konan, C. Yanofsky: Role of ribosome release in regulation of tna operon expression in Escherichia coli. In: Journal of Bacteriology. 181, Nr. 5, August 1999, S. 1530–1536. PMC 93543 (freier Volltext).
- J. Donahue, C. Turnbough: Nucleotide-specific transcriptional pausing in the pyrBI leader region of Escherichia coli K-12. In: Journal of Biological Chemistry. 269, Nr. 27, Juli 1994, S. 18185–18191. PMID 7517939.
- Y. Lu, R. Turner, R. Switzer: Function of RNA secondary structures in transcriptional attenuation of the Bacillus subtilis pyr operon. In: Proc Natl Acad Sci. 93, Nr. 25, Oktober 1996, S. 14462–14467. PMC 26155 (freier Volltext).
Literatur
- Nancy Trun, Janine Trempy: Gene expression and regulation. In: Nancy Trun, Janine Trempy: Fundamental Bacterial Genetics. Blackwell, Malden MA u. a. 2004, ISBN 0-632-04448-9, online (PDF; 500 kB).