Glutamatcysteinligase
Glutamatcysteinligasen (GCL) sind Enzyme aus der Gruppe der Ligasen. Diese Art der Ligase katalysiert die „Verknüpfung“ (Ligation) von Glutamat mit Cystein zu dem Dipeptid γ-Glutamylcystein. Das ist der erste Schritt bei der Bildung des Antioxidans Glutathion. Da dieser Reaktionsschritt in den meisten Lebewesen die Gesamtgeschwindigkeit der Glutathionsynthese bestimmt, können Mutationen in Genen, die den genetischen Bauplan für GCL enthalten, zu einem Glutathionmangel führen. Hierdurch kann bei Pflanzen und Tieren die Fähigkeit beeinträchtigt werden, giftige Stoffe und reaktive Sauerstoffspezies abzubauen. Bei völligem Ausfall der GCL sind die meisten höheren Organismen nicht lebensfähig.
| Glutamatcysteinligase (Brassica juncea) | ||
|---|---|---|
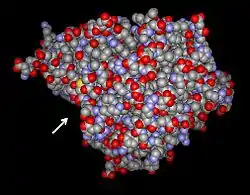 | ||
| Strukturmodell der Glutamatcysteinligase aus dem Braunen Senf (Brassica juncea). Der Pfeil weist auf den Zugang zum aktiven Zentrum. nach PDB 2GWD | ||
| Masse/Länge Primärstruktur | 459 Aminosäuren | |
| Sekundär- bis Quartärstruktur | Mono-/Homodimer | |
| Kofaktor | Mg2+ | |
| Bezeichner | ||
| Gen-Name(n) | GSH1 | |
| Externe IDs | ||
| Enzymklassifikation | ||
| EC, Kategorie | 6.3.2.2, Ligase | |
| Reaktionsart | Bildung einer Peptidbindung | |
| Substrat | ATP + L-Glutamat + L-Cystein | |
| Produkte | ADP + Phosphat + γ-L-glutamyl-L-cystein | |
| Vorkommen | ||
| Übergeordnetes Taxon | Cyanobakterien, Proteobacteria, Eukaryoten | |
| Glutamatcysteinligase (Homo sapiens) | ||
|---|---|---|
| Masse/Länge Primärstruktur | 910 = 636 + 274 Aminosäuren | |
| Sekundär- bis Quartärstruktur | Dimer (GCLC + GCLM) | |
| Kofaktor | Mg2+/(Mn2+) | |
| Bezeichner | ||
| Gen-Name(n) | GCLC, GCLM | |
| Externe IDs | ||
| Glutamatcysteinligase (E. coli K12) | ||
|---|---|---|
 | ||
| Bändermodell des Tetramers von E. coli nach PDB 1V4G | ||
| Masse/Länge Primärstruktur | 518 Aminosäuren | |
| Sekundär- bis Quartärstruktur | Tetramer | |
| Kofaktor | Mn2+/Mg2+/Cu2+ | |
| Bezeichner | ||
| Gen-Name(n) | gshA | |
| Externe IDs | ||
Bedeutung
Die Aktivität der Glutamatcysteinligase bestimmt in den meisten Lebewesen die Geschwindigkeit der Glutathionsynthese. Damit hat sie einen entscheidenden Einfluss auf die zelluläre Glutathionkonzentration und zahlreiche weitere Vorgänge im Primärstoffwechsel.[1][2] Glutathion erfüllt im Stoffwechsel vielfältige Funktionen, z. B. als Transport- und Speicherform reduzierten Schwefels, als Antioxidans und Redoxmetabolit, bei der Biotransformation (Phase II) von Schwermetallen und Xenobiotika sowie als Regulator zellulärer Prozesse.
Es konnte gezeigt werden, dass Mutanten der Acker-Schmalwand (Arabidopsis thaliana) mit verringerter Glutamatcysteinligase-Aktivität eine geringere Toleranz gegenüber Schwermetallen und Krankheitserregern aufwiesen. Bei fast vollständigem Ausfall kam die Bildung von Meristem in der Wurzelspitze zum Stillstand.[3] Mäuse, die aufgrund eines Knockouts der regulatorischen Untereinheit eine verringerte Glutamatcysteinligase-Aktivität entwickelten, waren empfindlicher gegenüber Xenobiotika.[4] Während höhere Eukaryoten wie Tiere und Pflanzen bei einem völligen Ausfall der Glutamatcysteinligase nicht mehr lebensfähig sind, bleiben Bakterien wie Escherichia coli trotz des Defekts lebens- und teilungsfähig sowie resistent gegen verschiedene Stressfaktoren.[5] Bei einigen mitochondrienlosen Eukaryoten (Entamoeba histolytica, Giardia, Trichomonas) sowie den meisten grampositiven Bakterien und Archaea fehlt die Glutamatcysteinligase und die Fähigkeit zur Glutathionsynthese völlig.[6]
Evolution, Vorkommen und Struktur
Es wird vermutet, dass die Glutamylcystein- und Glutathionsynthese ursprünglich einen evolutionären Vorteil bot, da beide Substanzen weniger empfindlich gegenüber spontaner Oxidation sind als Cystein und sich daher besser als zelluläre Speicherformen für reduzierten Schwefel eignen.[7]
Für Glutamatcysteinligase codierende Gene (meist als gshA oder GSH1 bezeichnet) wurden bei allen untersuchten aeroben Eukaryoten sowie bei Cyanobakterien und Alpha-, sowie Gamma-Proteobakterien gefunden. Die Gene können aufgrund von DNA-Sequenzanalysen in drei Gruppen unterteilt werden, die nur eine geringe Sequenzähnlichkeit aufweisen. Trotzdem weisen lokale Ähnlichkeiten von kurzen Teilstücken auf einen gemeinsamen evolutionären Ursprung aller Glutamatcysteinligase-Gene hin, der bei den Cyanobakterien vermutet wird. Von diesen ausgehend wurden die Gene von anderen Bakterien und Eukaryoten wahrscheinlich über lateralen Gentransfer erworben, wobei Sequenzanalysen für die Eukaryoten auch einen Erwerb über die Endosymbiose des proteobakteriellen Vorläufers der Mitochondrien nicht ausschließen.[6]

Gruppe 1 umfasst Gene aus Gamma-Proteobakterien. Die Kristallstruktur des etwa 55 kDa schweren Proteins aus Escherichia coli ist aufgeklärt worden.[8] Das nahezu globuläre Protein lag als Tetramer vor. Bei Streptokokken kommt darüber hinaus ein Gen vor, das für ein Protein codiert, welches die Aktivitäten von Glutamatcysteinligase und Glutathionsynthase vereinigt, also die gesamte Synthese von Glutathion aus den Aminosäuren katalysieren kann.[9] Dieses bifunktionale Enzym weist am amino-terminalen Ende Ähnlichkeit zu Gruppe-1-GCL-Proteinen auf.
Gruppe 2 umfasst die Gene aus Eukaryoten, mit Ausnahme der Chloroplastida (Pflanzen und Grünalgen). Neben nicht-photosynthetisch aktiven Eukaryoten schließt dies auch Kieselalgen und Rotalgen ein.[10] Diese GCL-Proteine umfassen als Holoenzym neben der katalytischen Untereinheit (GCLC) eine kleinere regulatorische Untereinheit (GCLM), die keine Homologie zur katalytischen Untereinheit oder anderen GCL-Proteinen aufweist. Dieses Heterodimer ist durch eine Disulfidbrücke kovalent verbunden, die auch bei der Regulation der Aktivität eine wichtige Rolle spielt. Beim Menschen haben die Untereinheiten eine Molekülmasse von etwa 70 kDa (GCLC) und 30 kDa (GCLM).[11][12]
Gruppe 3 umfasst Gene aus Alpha-Proteobakterien, Pflanzen und Grünalgen[13] sowie Gene aus einigen wenigen Gamma-Proteobakterien und, als distinkte Untergruppe, die Gene aus Cyanobakterien. Die Kristallstruktur konnte für das Protein aus dem Braunen Senf (Brassica juncea) aufgeklärt werden, das eine Molekülmasse von etwa 50 kDa besitzt.[14] Trotz sehr geringer Sequenzähnlichkeit konnte dabei eine strukturelle Ähnlichkeit zu dem Enzym aus Escherichia coli nachgewiesen werden. Das Enzym aus Brassica bildete nicht-kovalent verknüpfte Homodimere. Bei höheren Pflanzen handelt es sich um ein Chloroplastenprotein, bei Grünalgen und Moosen kommen möglicherweise auch cyytosolische Varianten vor.[13]
Biochemie
Die Glutamatcysteinligase katalysiert die Bildung einer Peptidbindung zwischen der Aminogruppe eines Cysteinmoleküls und der gamma-Carboxygruppe eines Glutamatmoleküls, wobei gamma-L-Glutamyl-L-Cystein (γ-Glutamylcystein) gebildet wird. Die Reaktion ist mit der Spaltung eines Moleküls Adenosintriphosphat (ATP) zu Adenosindiphosphat (ADP) und Phosphat gekoppelt:
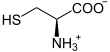 +
+ 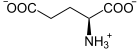 + ATP +
+ ATP + 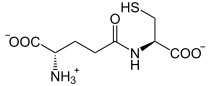 + ADP + Pi
+ ADP + Pi
Glutathion wird anschließend über die Glutathionsynthase durch Bindung eines Glycinmoleküls an die Cystein-Carboxygruppe des γ-Glutamylcysteins gebildet. Die Glutamatcysteinligase wird durch ihre Produkte sowie durch Glutathion inhibiert (negative Rückkopplung). Als spezifische Inhibitoren gelten Buthioninsulfoximin (BSO) und Methionin-Sulfoximin (MSO).
Als ATP-verbrauchendes Enzym benötigt die Glutamatcysteinligase zweifach positiv geladene Ionen als Kofaktor. Hierbei handelt es sich meist um Magnesium-Ionen, wobei diese je nach Spezies auch durch Mangan-Ionen ersetzt werden können. Während Glutamatcysteinligasen aus Tieren eine höhere Aktivität bei Anwesenheit von Natriumionen zeigen und von Kaliumionen inhibiert werden,[15] zeigen pflanzliche Enzyme genau umgekehrte Präferenzen.[16]
| Organismus | Km (Cystein) | Km (Glutamat) | Km (ATP) |
|---|---|---|---|
| Escherichia coli | 0,1 mM | 0,5 mM | 0,01 mM |
| Mensch (Homo sapiens) – Holoenzym | 0,8 mM | 0,7 mM | 0,4 mM |
| Mensch – GCLC allein | 0,5 mM | 3,5 mM | n. d. |
| Brauner Senf (Brassica juncea)[14] | 0,12 | 8,5 | 1,3 |
Regulation
Die Aktivität der Glutamatcysteinligase wird auf verschiedenen Ebenen reguliert. So reagiert die Genexpression bei vielen Organismen entwicklungs- und stressabhängig. Zum Beispiel lässt sich in Pflanzen eine erhöhte RNA- und/oder Proteinmenge nach Behandlung mit Schwermetallen und manchen Pflanzenhormonen nachweisen, welche mit einer erhöhten Glutathionsynthese einhergeht.
Des Weiteren liegen die Km-Werte des Enzyms für die Substrate Glutamat und Cystein so wie der Ki-Wert für Glutathion bei vielen Organismen nahe der physiologischen Konzentration dieser Metabolite, so dass Veränderungen ihrer Konzentration einen starken Einfluss auf die Aktivität der Glutamatcysteinligase ausüben können. Eine solche Regulation kann über Synthese und Abbau dieser Substanzen geschehen oder über deren Transport in und aus den verschiedenen Organellen. Dabei liegt die Glutamatcysteinligase bei Tieren, Pilzen und Bakterien im Cytosol vor, findet sich aber in höheren Pflanzen ausschließlich in den Plastiden.[18]
In Eukaryoten wird die Glutamatcysteinligase über die genannten Regulationsmechanismen hinaus durch Reduktionsmittel inhibiert. Bei tierischen Enzymen wird dies durch die Trennung der Disulfidbrücke zwischen katalytischer und regulatorischer Untereinheit erreicht, wodurch sich der Km-Wert für Glutamat erhöht und das Protein leichter durch Glutathion inhibierbar wird, so dass insgesamt die Aktivität sinkt.[11][12] Bei Pflanzen bricht Reduktion eine intramolekulare Disulfidbrücke auf, wodurch das Homodimer zerfällt, was wiederum zu einem Abfall der Aktivität führt.[14] Dieser Mechanismus ist dabei spezifisch für höhere Pflanzen und einige Grünalgen und findet sich nicht bei den sequenzähnlichen bakteriellen Enzymen der Gruppe 3.[13]
Medizinische Bedeutung
Die Expression von GCL findet beim Menschen prinzipiell in allen Gewebetypen und ausschließlich im Zytosol statt. Der Mensch produziert besonders viel Glutathion in der Leber, wo mengenmäßig die meisten Fremdstoffe und Stoffwechselendprodukte durch Biotransformation unschädlich gemacht werden. Das heißt, dass auch die GCL dort am stärksten exprimiert wird. Andere wichtige Gewebe, in denen GCL überdurchschnittlich stark gebildet wird, sind die Epithelzellen der Bronchien, Erythrozyten sowie dendritische und natürliche Killerzellen des Immunsystems. Ein Ausfall des Enzyms in der Leber von Mäusen führte nach kurzer Zeit zur Leberverfettung, zur Entzündung und zum Verlust des Organs.[19][20] Vergleichbare Effekte sind auch beim Menschen beschrieben.[21]
Mutationen am menschlichen GCLC-Gen können unter anderem zum sehr seltenen familiären GCL-Mangel führen; dieser geht einher mit hämolytischer Anämie. Unterschiede in der Fähigkeit der Arterien zur Gefäßerweiterung sind mit unterschiedlichen GCL-Varianten assoziiert.[22][23]
Mukoviszidose-Patienten haben durch ihr erhöhtes Lungeninfektionsrisiko einen erhöhten Bedarf an Biotransformation schwer löslicher Fremdstoffe in der Lunge. Dies ist wahrscheinlich der Grund, warum in bestimmten Patientengruppen eine unterschiedliche Schwere von Lungenproblemen mit Varianten der GCL assoziiert sind.[24]
Neben der Regulation der Enzymaktivität durch die Konzentration des Endprodukts und Glutathions ist auch die Produktion der GCL selbst Schwankungen unterworfen, die das Resultat von Signalkaskaden sind. Auf diese Weise ist der Körper in der Lage, sich auf erhöhten Bedarf nach Glutathion einzustellen, indem die Produktion von GCL erhöht wird. So sind Xenobiotika und mehrere sekundäre Pflanzenstoffe, aber auch Insulin und Zink in der Lage, über Nrf2 und den PI3K/Akt/mTOR-Signalweg die Phase-II-Antwort der Biotransformation hochzufahren und damit unter anderem die GCLC/GCLM-Expression zu erhöhen.[25][26][27] Auf der anderen Seite verringern TGF-β1 und Lipopolysaccharide die GCL-Expression und damit den Glutathiongehalt in der Leber.[28]
Geschichte
Zunächst fanden Johnston und Bloch in den Jahren 1949 und 1951 eine Möglichkeit, die aktiven Enzyme bei der Biosynthese von Glutathion aus den Aminosäuren aus Taubenleber zu isolieren. Die Zweistufigkeit der Synthese wurde 1952 von Snoke und Bloch erkannt. 1953 berichtete Webster über die Synthese von γ-Glutamylcystein bei der Gartenbohne.[29][30][31][32]
Bakterielle γ-Glutamatcysteinligase wurde bereits 1982 isoliert und 1986 sequenziert. Die Reingewinnung und erste Sequenzanalyse tierischer GCL ist eng mit dem Namen Alton Meister (1922–1995) verknüpft. Der US-amerikanische Biochemiker schuf zwischen 1971 und 1985 in seinem Labor die Voraussetzungen für die Extraktion des Enzyms aus Rattenleber und -erythrozyten. 1984 erkannte er, dass GCL aus zwei Untereinheiten besteht. 1990 konnte das Gen für die GCL der Ratten und 1992 das menschliche Gen kloniert und sequenziert werden.[33][34]
Die einzigen bekannten Proteinstrukturen sind momentan die vom braunen Senf (Brassica juncea, 2006) und von Escherichia coli K12 (2004).
Quellen
- Graham Noctor, Leonardo Gomez, Hélène Vanacker, Christine H. Foyer: Interactions between biosynthesis, compartmentation and transport in the control of glutathione homeostasis and signalling. In: Journal of Experimental Botany. Band 53, Nr. 372, 2002, S. 1283–1304 (englisch, oxfordjournals.org [PDF]).
- Dale A. Dickinson, Henry Jay Forman: Cellular glutathione and thiols metabolism. In: Biochemical Pharmacology. Band 64, Nr. 5-6, 2002, S. 1019–1026 (englisch).
- Eintrag bei BRENDA
Weblinks
- D'Eustachio/reactome.org: Glutamate and cysteine combine
Einzelnachweise
- Graham Noctor, Leonardo Gomez, Hélène Vanacker, Christine H. Foyer: Interactions between biosynthesis, compartmentation and transport in the control of glutathione homeostasis and signalling. In: Journal of Experimental Botany. Band 53, Nr. 372, 2002, S. 1283–1304 (englisch, oxfordjournals.org [PDF]).
- Dale A. Dickinson, Henry Jay Forman: Cellular glutathione and thiols metabolism. In: Biochemical Pharmacology. Band 64, Nr. 5-6, 2002, S. 1019–1026 (englisch).
- Thomas Rausch, Roland Gromes, Verena Liedschulte, Ina Müller, Jochen Bogs, Vladislava Galovic, Andreas Wachter: Novel Insight into the Regulation of GSH Biosynthesis in Higher Plants. In: Plant Biology. Band 9, Nr. 5, 2007, S. 565–572 (englisch).
- Lisa A. McConnachie, Isaac Mohar, Francesca N. Hudson, Carol B. Ware, Warren C. Ladiges, Carolina Fernandez, Sam Chatterton-Kirchmeier, Collin C. White, Robert H. Pierce, Terrance J. Kavanagh: Glutamate Cysteine Ligase Modifier Subunit Deficiency and Gender as Determinants of Acetaminophen-Induced Hepatotoxicity in Mice. In: Toxicological Sciences. Band 99, Nr. 2, 2007, S. 628–636 (englisch).
- Jean T. Greenberg, Bruce Demple: Glutathione in Escherichia coli is dispensable for resistance to H2O2 and gamma radiation. In: Journal of Bacteriology. Band 168, Nr. 2, 1986, S. 1026–1029, PMID 213589 (englisch).
- Shelley D. Copley, Jasvinder K. Dhillon: Lateral gene transfer and parallel evolution in the history of glutathione biosynthesis genes. In: Genome Biology. Band 3, Nr. 5, 2002, S. research0025.1–research0025.16, PMID 115227 (englisch).
- R.C. Fahey, A.R. Sundquist: Evolution of Glutathione Metabolism. In: Advances in Enzymology and Related Areas of Molecular Biology. Band 64, 1991, S. 1–53 (englisch).
- T. Hibi, H. Nii u. a.: Crystal structure of gamma-glutamylcysteine synthetase: insights into the mechanism of catalysis by a key enzyme for glutathione homeostasis. In: Proceedings of the National Academy of Sciences. Band 101, Nummer 42, Oktober 2004, S. 15052–15057, doi:10.1073/pnas.0403277101. PMID 15477603. PMC 523444 (freier Volltext).
- B. E. Janowiak, O. W. Griffith: Glutathione synthesis in Streptococcus agalactiae. One protein accounts for gamma-glutamylcysteine synthetase and glutathione synthetase activities. In: The Journal of biological chemistry. Band 280, Nummer 12, März 2005, S. 11829–11839, doi:10.1074/jbc.M414326200. PMID 15642737.
- Roland Gromes: Post-translational regulation and evolution of plant γ-glutamate cysteine ligase. Heidelberg 2007, S. 88–92 (englisch, Volltextzugang).
- Huang, C.-S., Anderson, M.E., and Meister, A.: Amino acid sequence and function of the light subunit of rat kidney gamma-glutamylcysteine synthetase.. In: J Biol Chem. Nr. 268, 1993, S. 20578–20583.
- Huang, C.-S., Chang, L.S., Anderson, M.E., and Meister, A.: Catalytic and regulatory properties of the heavy subunit of rat kidney gamma-glutamylcysteine synthetase.. In: J Biol Chem. Nr. 268, 1993, S. 19675–19680.
- Roland Gromes, Michael Hothorn, Esther D. Lenherr, Vladimir Rybin, Klaus Scheffzek, Thomas Rausch: Redox-switch of gamma-glutamylcysteine ligase via reversible monomer-dimer transition is a mechanism unique to plants. In: Plant Journal. Band 54, Nr. 6, 2008, S. 1063–1075.
- Michael Hothorn, Andreas Wachter, Roland Gromes, Tobias Stuwe, Thomas Rausch, Klaus Scheffzek: Structural Basis for the Redox Control of Plant Glutamate Cysteine Ligase. In: Journal of Biological Chemistry. Band 281, Nr. 37, 2006, S. 27557–27565 (englisch, jbc.org [PDF]).
- J.S. Davis, J.B. Balinsky, J.S. Harington, J.B. Shepherd: Assay, purification, properties and mechanism of action of gamma-glutamylcysteine synthetase from the liver of the rat and Xenopus laevis. In: Biochemical Journal. Band 133, 1973, S. 667–678, PMID 1177756 (englisch).
- George C. Webster, J.E. Varner: Peptide-bond synthesis in higher plants. II. Studies on the mechanism of synthesis of gamma-glutamylcysteine. In: Archives of Biochemistry and Biophysics. Band 52, 1954, S. 22–32 (englisch).
- Glutamatcysteinligase bei BRENDA
- Andreas Wachter, Sebastian Wolf, Heike Steininger, Jochen Bogs, Thomas Rausch: Differential targeting of GSH1 and GSH2 is achieved by multiple transcription initiation: implications for the compartmentation of glutathione biosynthesis in the Brassicaceae. In: Plant Journal. Band 41, Nr. 1, 2005, S. 15–30, PMID 15610346 (englisch).
- (Seite nicht mehr abrufbar, Suche in Webarchiven: BioGPS Expressionsdaten menschliche GCLC)
- Chen Y, Yang Y, Miller ML, et al.: Hepatocyte-specific Gclc deletion leads to rapid onset of steatosis with mitochondrial injury and liver failure. In: Hepatology. 45, Nr. 5, Mai 2007, S. 1118–28. doi:10.1002/hep.21635. PMID 17464988.
- Oliveira, CP et al.: Association of polymorphisms of glutamate-cystein ligase and microsomal triglyceride transfer protein genes in non-alcoholic fatty liver disease. In: J Gastroenterol Hepatol. 2010 Feb;25(2):357-61
- Orphanet: Gamma-glutamylcysteine synthetase deficiency.
- Koide S, Kugiyama K, Sugiyama S, et al.: Association of polymorphism in glutamate-cysteine ligase catalytic subunit gene with coronary vasomotor dysfunction and myocardial infarction. In: J. Am. Coll. Cardiol.. 41, Nr. 4, Februar 2003, S. 539–45. PMID 12598062.
- McKone EF, Shao J, Frangolias DD, et al.: Variants in the glutamate-cysteine-ligase gene are associated with cystic fibrosis lung disease. In: Am. J. Respir. Crit. Care Med.. 174, Nr. 4, August 2006, S. 415–9. doi:10.1164/rccm.200508-1281OC. PMID 16690975.
- W. Langston, M.L. Circu, T.Y. Aw: Insulin stimulation of gamma-glutamylcysteine ligase catalytic subunit expression increases endothelial GSH during oxidative stress: influence of low glucose. In: Free Radic Biol Med. 45, Nr. 11, Dezember 2008, S. 1591–9. doi:10.1016/j.freeradbiomed.2008.09.013. PMID 18926903.
- M.M. Cortese, C.V. Suschek, W. Wetzel, K.D. Kröncke, V. Kolb-Bachofen: Zinc protects endothelial cells from hydrogen peroxide via Nrf2-dependent stimulation of glutathione biosynthesis. In: Free Radic. Biol. Med.. 44, Nr. 12, Juni 2008, S. 2002–12. doi:10.1016/j.freeradbiomed.2008.02.013. PMID 18355458.
- L.G. Higgins, C. Cavin, K. Itoh, M. Yamamoto, J.D. Hayes: Induction of cancer chemopreventive enzymes by coffee is mediated by transcription factor Nrf2. Evidence that the coffee-specific diterpenes cafestol and kahweol confer protection against acrolein. In: Toxicol. Appl. Pharmacol.. 226, Nr. 3, Februar 2008, S. 328–37. doi:10.1016/j.taap.2007.09.018. PMID 18028974.
- K. Ko, H. Yang, M. Noureddin, et al.: Changes in S-adenosylmethionine and GSH homeostasis during endotoxemia in mice. In: Lab. Invest.. 88, Nr. 10, Oktober 2008, S. 1121–9. doi:10.1038/labinvest.2008.69. PMID 18695670.
- BLOCH K: The synthesis of glutathione in isolated liver. (PDF) In: J. Biol. Chem.. 179, Nr. 3, Juli 1949, S. 1245–54. PMID 18134587.
- Johnston RB, Bloch K: Enzymatic synthesis of glutathione. In: J. Biol. Chem.. 188, Nr. 1, Januar 1951, S. 221–40. PMID 14814132.
- SNOKE JE, BLOCH K: Formation and utilization of gamma-glutamylcysteine in glutathione synthesis. In: J. Biol. Chem.. 199, Nr. 1, November 1952, S. 407–14. PMID 12999854.
- Webster GC: Enzymatic Synthesis of Gamma-Glutamyl-Cysteine in Higher Plants. In: Plant Physiol.. 28, Nr. 4, Oktober 1953, S. 728–30. PMID 16654590. PMC 540436 (freier Volltext).
- Yan N, Meister A: Amino acid sequence of rat kidney gamma-glutamylcysteine synthetase. In: J. Biol. Chem.. 265, Nr. 3, Januar 1990, S. 1588–93. PMID 1967255.
- Gipp JJ, Chang C, Mulcahy RT: Cloning and nucleotide sequence of a full-length cDNA for human liver gamma-glutamylcysteine synthetase. In: Biochem. Biophys. Res. Commun.. 185, Nr. 1, Mai 1992, S. 29–35. PMID 1350904.