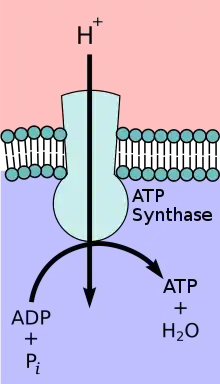
ATP-Synthase
Das Enzym ATP-Synthase oder FoF1-ATPase ist ein Transmembranprotein. Die ATP-Synthase tritt abhängig vom Verhältnis der Substrate und Produkte entweder als ATP-verbrauchende Protonenpumpe oder als protonen-getriebene ATP-Synthase auf. Unter physiologischen Bedingungen besteht die Hauptaufgabe des Enzyms allerdings darin, die Synthese von ATP zu katalysieren. ATP ist eine energiereiche Verbindung, deren Bildung der Zufuhr von Energie bedarf:
- ADP + Phosphat → ATP + H2O mit ΔH ≈ 30,5 kJ/mol unter Standardbedingungen und ca. 50 kJ/mol unter physiologischen Bedingungen.
| ATP-Synthase | ||
|---|---|---|
| Bezeichner | ||
| Gen-Name(n) | Fo, F1 | |
| Transporter-Klassifikation | ||
| TCDB | 3.A.2 | |
| Bezeichnung | F-ATPase Superfamilie | |
| Enzymklassifikation | ||
| EC, Kategorie | 7.1.2.2, Translokase | |
| Substrat | ADP + Phosphat + H+außen | |
| Produkte | ATP + H2O + H+innen | |
Um diese Energie aufzubringen, koppelt die ATP-Synthase die ATP-Bildung mit dem energetisch begünstigten Transport von Protonen (oder anderen Ionen) entlang eines Protonengefälles über eine Membran. Die ATP-Synthase ist also ein Energiewandler, der eine Energieform in eine andere umformt. Das Enzym spielt im Stoffwechsel fast aller bekannten Organismen eine zentrale Rolle, da ATP ununterbrochen als Energieüberträger benötigt wird.
Die ATP-Synthase setzt sich aus 8 bis 20 verschiedenen Untereinheiten zusammen. Sie gruppieren sich zu zwei Komplexen:
- Der wasserlösliche Komplex F1 katalysiert die Bildung von ATP.
- Der wasserunlösliche, in eine Membran eingebaute Komplex Fo transportiert Protonen.
Das Enzym wird daher auch nach seinen beiden Untereinheiten als FoF1-ATPase bezeichnet.
Bedeutung und Vorkommen
Praktisch alle Vorgänge in Organismen erfordern Adenosintriphosphat (ATP). Es stellt allen möglichen Stoffwechselvorgängen Energie zur Verfügung. Der größte Teil des verbrauchten ATP wird bei Tieren, Pflanzen und den meisten Bakterien durch die ATP-Synthase regeneriert. Der Tagesumsatz an ATP beträgt beim Menschen teilweise weit über 80 Kilogramm.
Die ATP-Synthase alias F-Typ-ATPase kommt vor
- in der Plasmamembran von Prokaryoten („Bakterien“).
- in der inneren Mitochondrienmembran von Eukaryoten (Zellen von Pflanzen und Tieren).
- in der Thylakoid-Membran der Chloroplasten von Pflanzenzellen.
Urtümliche Organismen aus dem Reich der Archaeen haben eine A-Typ-ATPase, die im Aufbau etwas von der „normalen“ ATP-Synthase abweicht. Der Grund könnte mit dem abweichenden Aufbau von Zellmembran und Zellwand dieser Organismen zusammenhängen.
ATP-Synthasen nutzen die Energie eines Ionengradienten, der zwischen den beiden Seiten der Membran existiert. In der Regel handelt es sich dabei um einen Protonengradienten. Bei alkaliphilen Bakterien existiert auch eine ATP-Synthase, die statt eines Protonen- einen Natrium-Gradienten zur ATP-Synthese verwendet. (EC 7.2.2.1)
Geschichte
Die Aufklärung von Funktion und Mechanismus der ATP-Synthase wurde im Wesentlichen von Forschern geleistet, die sich mit Mitochondrien beschäftigten. Obwohl dieses Enzym auch bei der Photosynthese der Pflanzen und bei aeroben Bakterien eine wichtige Rolle spielt, geriet die ATP-Synthase als Bestandteil der Zellatmung und der Atmungskette des Menschen in den Fokus der Biochemie.
Der Komplex V
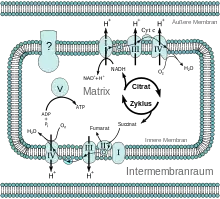
Anfang der 1960er Jahre konnte die Biochemie auf enorme Fortschritte zurückblicken. Der Energiestoffwechsel war in ein paar Jahrzehnten fast aufgeklärt worden. Der Zitronensäurezyklus war bekannt, ebenso die physiologische Rolle von NADH als Wasserstoff-Überträger und die Rolle von ATP als Energielieferant.
Man wusste, dass der NADH-Wasserstoff mit Sauerstoff zu Wasser oxidiert wird. Und es war klar, dass bei diesem Prozess die Hauptmasse des in der Zelle benötigten ATP entsteht. Darüber hinaus wusste man, dass die Oxidation des NADH in Schritten verläuft. Man hatte in den Mitochondrien-Membranen Enzyme und Coenzyme gefunden, die eine Elektronentransport-Kette vom NADH zum Sauerstoff bilden.
Die Aufklärung dieser sogenannten Atmungskette gestaltete sich indes zunehmend schwieriger. Die Enzyme der Atmungskette ließen sich nur schwer isolieren und untersuchen, da es sich um Membranproteine handelt. Zusätzlich bilden sie große Enzym-Komplexe. Vier dieser Komplexe wurden (und werden) immer weiter aufgeklärt. Komplex V, der ATP bildet, blieb auch nach Isolierung seiner wasserlöslichen Komponente im Jahr 1961 im Dunkeln. Die Aufklärung der Atmungskette, und damit die Biochemie überhaupt, hatte also noch einen „Schönheitsfehler“.
Aus den Forschungen über den Zuckerabbau (Glycolyse) kannte man immerhin bereits einen Mechanismus (Substratkettenphosphorylierung), bei dem aus ADP und Phosphat ATP entsteht. Man zog den Schluss, dass es sich in der Atmungskette irgendwie ähnlich verhalten müsste und glaubte, dicht vor der endgültigen Aufklärung zu sein. Es fehlte „nur noch“
- die genaue Verbindung der Komplexe I–IV zur ATP-Synthese, also die Kopplung von O2-Verbrauch und ATP-Bildung.
- die Erklärung, warum der gefundene Komplex V zwar ATP spaltete, aber keines herstellte, sobald man ihn isoliert hatte.
- ein „energiereiches Zwischenprodukt“ der Atmungskette als Substrat der ATP-Synthase.
Es fehlte also „nur noch“ der gesamte Zusammenhang zwischen NADH-Oxidation und ATP-Produktion. Aber man hatte schon einen bis heute gültigen Namen für den Stoffwechsel-Weg: oxidative Phosphorylierung.
In dieser Situation stellte Peter D. Mitchell 1960 eine Hypothese vor, die lange heftig angefeindet wurde. Denn Mitchell postulierte einen für die damalige Biochemie „unvorstellbaren“ Mechanismus.
Die Mitchell-Hypothese
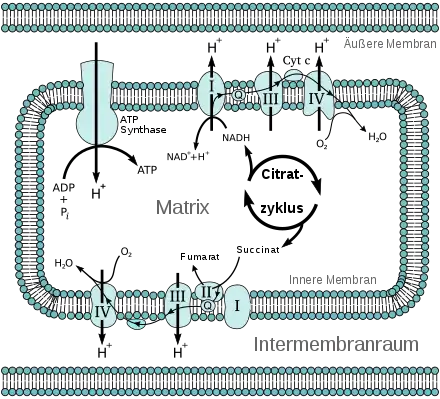
Mitchell selbst hatte zwar seit Beginn der 1940er Jahre schwerpunktmäßig die Atmungskette untersucht, kannte aber auch die Forschungsergebnisse der Transportvorgänge an Membranen. Seine Arbeitsgruppe konzentrierte sich bei der Untersuchung der Atmungskette auf den Elektronenüberträger Ubichinon (Q-Zyklus). Die Ergebnisse waren mit der Idee eines „energiereichen Zwischenprodukts“ der Atmungskette als Motor der ATP-Synthase nicht vereinbar. Mitchell postulierte stattdessen, dass das Enzym seine Energie von einem pH-Gradienten bekommt.
Die Ablehnung der Fachwelt war überwältigend. Selbst als Mitchell 1978 für seine chemiosmotische Theorie den Nobelpreis (Chemie) bekam, sprachen namhafte Biochemiker noch abwertend von der „Mitchell-Hypothese“.
Die Aufklärung des Mechanismus
Auch Wissenschaftler, die der „Mitchell-Hypothese“ offiziell sehr skeptisch gegenüberstanden, zogen bei ihrer Forschung im Labor die chemiosmotische Theorie ernsthaft in Betracht. Während sich die Beweise für Mitchells Theorie häuften, kam man auch im experimentellen Umgang mit der ATP-Synthase weiter. In Membranvesikeln konnte man sie „in Aktion“ studieren.
Paul D. Boyer, ursprünglich ein „Mitchell“-Skeptiker, klärte den molekularen Mechanismus der ATP-Synthase auf. John E. Walker und Mitarbeitern gelang es, die ATP-Synthase zu kristallisieren und ihre räumliche Struktur aufzuklären. Beide bekamen dafür den Nobelpreis 1997 im Fach Chemie. Sie teilten sich den Preis mit Jens C. Skou, der bereits 1957 die erste Protonenpumpe entdeckt und damit die Grundlage für die „Mitchell-Hypothese“ gelegt hatte.
Stellung zu anderen Enzymen
Eine Vielzahl von Enzymen verbraucht das ATP, das von der ATP-Synthase geliefert wird, als Kosubstrat. Unter den Enzymen nimmt die ATP-Synthase also eine Schlüsselstellung ein.
Von den übrigen ATPasen unterscheidet sich die ATP-Synthase in verschiedener Hinsicht:
- Sie ist die Hauptquelle des ATP. Mit ihrer Funktion als ATP-Produzent unterscheidet sich die ATP-Synthase von den übrigen (durchweg) ATP-verbrauchenden ATPasen etwa wie ein Stromgenerator von einem Elektromotor.
- Sie besteht aus den beiden Untereinheiten Fo und F1. Ihre Funktion erfordert beide Einheiten in einer spezifischen Anordnung. Man bezeichnet die ATP-Synthase daher oft auch als F-Typ-ATPase. Im Unterschied dazu sind die ATP-spaltenden ATPasen V-Typ-ATPasen.
- Wie alle ATPasen kann die ATP-Synthase prinzipiell auch als Protonenpumpe fungieren und dabei ATP verbrauchen. Dass diese „Rück“-Reaktion in vivo in Mitochondrien eine große Rolle bei der ATP-Synthase spielt, ist allerdings fragwürdig. Die ATP-Synthase hat ein anderes „Übersetzungsverhältnis“ als die Protonenpumpen-ATPasen. Letztere pumpen pro verbrauchtem ATP ca. zwei Protonen nach außen. Bei der ATP-Synthase würde sich die Energie eines ATP-Moleküls dagegen auf drei bis vier Protonen verteilen. Als Protonenpumpe könnte die ATP-Synthase also keinen so großen pH-Gradienten aufbauen.
Die ATP-Synthase hat entsprechend der IUBMB Enzym-Nomenklatur die EC-Nummer EC 7.2.2.2 und gehört zur Kategorie der Translokasen.
Bauplan der ATP-Synthase
Entsprechend ihrer Stellung zur Membran (Abb. unten, heller Bereich) unterscheidet man eine membrangebundene Fo- und eine wasserlösliche F1-Untereinheit.
Mechanisch gesehen lässt sich das Enzym in einen sich drehenden Rotor (Abb. „Vereinfachtes Modell“, rötlich bis violett) und einen Stator (Abb., grün) gliedern. Wegen des flüssigen Charakters der Membran führt der Stator eine Drehbewegung gegenläufig zum Rotor aus.
Aufgrund dieses Aufbaus ist das Enzym wie auch die anderen membrangebundenen ATPasen eine molekulare Maschine.
Nachfolgend wird der Bauplan der ATP-Synthase des Bakteriums E. coli beschrieben, weil sie intensiv untersucht und relativ einfach gebaut ist.

Aufbau von Fo
Fo besteht aus hydrophoben Peptiden (wasserunlöslichen Eiweißketten) und befindet sich in der Membran. Dieser Teil des Enzyms setzt sich zusammen aus drei verschiedenen Untereinheiten:
- Foa dient zur Kraftübertragung auf Fob und ist gleichzeitig Teil des Mechanismus, der die Protonenbewegung in eine Drehbewegung umsetzt.
- Fob verbindet Membran und die F1 Komponente. Fob dient der Kraftübertragung und besteht wie Foa aus zwei Peptidketten.
- Foc besteht bei E. coli aus zwölf Spiralen, die zu einem Ring angeordnet sind. Im Inneren des Rings befinden sich vermutlich die Phospholipide, aus denen auch die Zellmembran zusammengesetzt ist. Sie bilden eine isolierende Schicht, so dass dort kein Protonenfluss möglich ist.
Jede Foc-Peptidkette verfügt über ein aktives Zentrum. Wenn von diesem Zentrum H+ abgespalten wird, dann ändert sich die Struktur der Peptidkette in einen mechanisch angespannten Zustand. Nimmt das aktive Zentrum dann wieder ein H+ auf, dreht sich die Peptidkette zurück. Diese Drehung übt eine Kraft auf Foa aus.
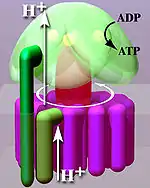
Aufbau von F1
F1 ist die wasserlösliche Komponente der ATP-Synthase. Sie befindet sich auf der Innenseite der Membran. Fünf verschiedene Peptide (α bis ε) bilden die Untereinheiten dieser Komponente:
- Je drei F1α- und F1β -Peptide bilden den F1(αβ)3-Komplex. Seine gelegentlich noch verwendete Bezeichnung als globuläre (αβ)3-ATPase weist darauf hin, dass hier die Umsetzung von ADP zu ATP stattfindet. Zwischen den Peptiden existieren insgesamt drei Poren, durch die Substrat und Produkt ein- und austreten können. In drei katalytischen Zentren wird im Inneren des F1(αβ)3-Komplexes das ATP produziert.
- F1γ ist die drehbare Achse des Systems. Sie überträgt die Drehbewegung des in der Membran platzierten Rings zu den drei katalytischen (αβ)3-Zentren.
- F1δ und F1ε sind im Bild rechts nicht dargestellt. Ersteres Peptid ist Bindeglied zwischen Fob und dem (αβ)3-Komplex. F1ε verbindet vermutlich den Foc-Ring mit der F1γ-Achse.
Übersicht über menschliche ATPase
Die menschliche ATPase besteht aus mindestens 16 Untereinheiten, von denen zwei durch mitochondriale Gene codiert sind. Sowohl von F1γ als von Foc existieren mehrere Genloci, die verschiedene Precursor, aber identische Untereinheiten codieren.
| Gen (HGNC) | Protein (UniProt) | Länge (AA) | OMIM-Eintrag | Genlocus | Bemerkung |
|---|---|---|---|---|---|
| ATP5A1 | F1α1 | 553 | 164360 | 18q12-q21 | |
| ATP5B | F1β | 529 | 102910 | 12p13-qter | |
| ATP5C1 | F1γ1 | 298 | 108729 | 10q22-23 | |
| ATP5C2 | F1γ2 | ? | 14 | Isoform | |
| ATP5D | F1δ | 168 | 603150 | 19p13.3 | |
| ATP5E | F1ε | 51 | 606153 | 20q13.3 | |
| ATP5O | F1O | 213 | 600828 | 21q22.1-22.2 | Oligomycin sensitivity conferral protein |
| ATP5F1 | Fob | 256 | 603270 | 1p13.2 | |
| ATP5G1 | Foc Isoform 1 | 136 | 603192 | 17q21.32 | Proteolipid P1; Untereinheit 9; Morbus Batten |
| ATP5G2 | Foc Isoform 2 | 141 | 603193 | 12q13.3 | Proteolipid P2; Morbus Batten |
| ATP5G3 | Foc Isoform 3 | 142 | 602736 | 2q31.1 | Proteolipid P3; Morbus Batten |
| ATP5H | Fod | 160 | 17q25 | Stator | |
| ATP5I | Foe | 68 | 601519 | 4p16.3 | |
| ATP5J | FoF6 | 108 | 603152 | 21q21.1 | Kopplungsfaktor 6 |
| ATP5J2 | Fof | 93 | 7q22.1 | ||
| ATP5L | Fog | 103 | 11q23.3 | ||
| ATP5S | Fos | 215 | 14q21.3 | Kopplungsfaktor B |
Bewegungs- und Reaktionsabläufe
Die Funktion der „molekularen Maschine“ wird hier am Beispiel der E. coli-ATP-Synthase beschrieben.
Hypothetischer Mechanismus der Drehbewegung
Der Ort des für die Bewegung zuständigen Zentrums geht aus der Abbildung „Motor“ hervor. Der Mechanismus der Drehbewegung ist in der Abbildung rechts als Animation dargestellt.

|
|
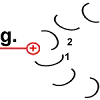
Jedes Foc-Peptid aus dem Rotor-Ring hat auf Position 61 einen Asparaginsäure(ASP)-Rest. Im Ruhezustand des Motors sind, mit einer Ausnahme, die ASP-Carboxyl-Gruppen protoniert (Peptid-COOH). Beim Rotor-Peptid 1, das neben dem Stator Fob liegt, befindet sich die ASP-Gruppe neben einer Arginin-Gruppe des Stators. Deren positive Ladung kann durch ionische Wechselwirkung eine negative Ladung stabilisieren, sodass der ASP-Rest deprotoniert vorliegt (Peptid-COO−). Das negativ geladene Rotor-Peptid 1 weicht in seiner räumlichen Struktur von den übrigen Foc-Peptiden ab. In einer Haarnadelschleife hat es wie in einer Spiralfeder eine starke mechanische Spannung aufgebaut. |
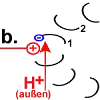 |
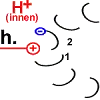
Rotor-Peptid 1 nimmt ein Proton von außen auf:
(Welchen Weg das Proton nimmt ist noch nicht genau geklärt. Die Foc-Peptide schaffen vielleicht den Raum dafür, dass es bis zur entscheidenden Position 61 vordringen kann. Nach der „Single Channel Theory“ schafft das Stator-Peptid den Zugang.) |
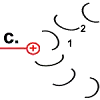 |
Sobald Rotor-Peptid 1 protoniert ist, geht seine spannungsreiche Struktur wieder in den entspannten Normalzustand über. Die „Spiralfeder“ gibt ihre Energie wieder ab. Auf den Stator wird eine Kraft ausgeübt. Das Peptid dreht sich.
Chemische Energie wird hier in Bewegungsenergie verwandelt. Die Drehung des Peptids 1 ist der Antrieb des Motors. |
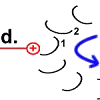 |
Die Drehbewegung bringt das Peptid 1 auf die andere Seite des Stator-Arginins und dreht den Rotor-Ring. |
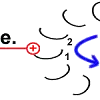 |
Nun hat Rotor-Peptid 1 dieselbe Form wie die anderen Rotor-Peptide, die keinen Stator als Nachbarn haben: protoniert und mit entspannter Spiralstruktur. |
 |
Der Ring ist nun um 30° gedreht.
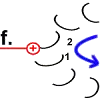
Die Bewegung des Rotor-Peptids hat allerdings gleichzeitig die nächste Foc-Peptidkette 2 in den „Bann“ der positiv geladenen Arginin-Gruppe gezogen. |
 |
Die Struktur des Peptid 2 wird in den spannungsreichen Zustand geändert. Die „Spiralfeder“ wird gespannt. |
 |
Peptid 2 trennt sich von dem H+, das es an der Aspartat-Gruppe an Position 61 hatte. Das Proton verlässt den Ort der Handlung in Richtung innen (im Bild: nach oben).
|
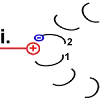 |
Der Ausgangszustand ist wiederhergestellt. Allerdings wurde
|
Als weitere Rotationsmaschine auf zellulärer Ebene, die komplette Drehungen ausführt, ist nur noch die Geißel bei vielen Einzellern bekannt.
ATP-Synthese im F1(αβ)3-Komplex
Die eigentliche Umsetzung von ADP zu ATP ist, biochemisch gesehen, im Vergleich zur Protonen getriebenen Drehung des Foc-Rings nichts Ungewöhnliches. Es finden Struktur-Änderungen der beteiligten Peptidketten statt, die die Substrate (hier ADP und Phosphat) zur Reaktion bringen.
Im F1(αβ)3-Komplex gibt es drei katalytische Zentren. Sie nehmen nacheinander drei Formen an:
- Hohe Affinität gegenüber ADP und Phosphat. Die Ausgangsprodukte ADP und Phosphat werden an das jeweilige katalytische Zentrum gebunden.
- Hohe Affinität gegenüber ATP. Das Zentrum bekommt einen hydrophoben Charakter, der die Kondensationsreaktion zum ATP energetisch begünstigt.
- Geöffnete Form, in der ATP ausgestoßen wird.
Der energieverbrauchende Schritt besteht darin, die geöffnete Form zu bilden, also das Reaktionsprodukt ATP aus dem Enzym zu entfernen. Genau das ermöglicht die Drehbewegung.
Die Drehung des Rotors um 360° liefert in drei Schritten drei Moleküle ATP. Da sich die „Maschine“ bei jedem Protonendurchgang um 30° dreht, werden nach diesem Modell für jedes ATP vier H+ verbraucht.
ATP-Synthasen bei verschiedenen Organismen
Die ATP-Synthase des Bakteriums E. coli besteht aus acht verschiedenen Proteinen. Die ATP-Synthase in den Mitochondrien der Hefe Saccharomyces cerevisiae besteht bei prinzipiell gleicher Bauweise aus 20 verschiedenen Proteinen.
Eine ganze Bandbreite (9–14) gibt es auch bei der Anzahl der Foc-Rotor-Peptide. Aus ihnen resultieren verschiedene Mengenverhältnisse zwischen verbrauchtem H+ und hergestelltem ATP.
| Typ/Herkunft | Anzahl der Foc-Peptide | H+ pro ATP |
|---|---|---|
| Bakterien (E. coli) | 10 | 4[1] |
| Mitochondrien (Hefe) | 10 | 3,3 |
| Chloroplasten (Spinat) | 14 | 4[1] |
| Na+-ATP-Synthase | 11 | 3,7 Na+/ATP |
Andere Bezeichnungen für die ATP-Synthase
- ATP-Synthetase
- Streng genommen eine fehlerhafte Bezeichnung, da Synthetasen eine chemische Bindung unter ATP-Verbrauch herstellen.
- ATPase
- Die ATP-Synthase ist die am häufigsten vorkommende ATPase. Daher werden beide Namen oft synonym gebraucht.
- F-ATPase, FoF1-ATPase (oder F1Fo-ATPase), H+-ATP-Synthase, H+-transporting two-sector ATPase
- ATP-Synthase bei Bakterien, Mitochondrien und Plastiden: Formal korrekte, teilweise aber weniger gebräuchliche Bezeichnungen
- A-ATPase, AoA1-ATPase
- ATPase bei Archaeen, ähnelt im Aufbau der V-ATPase bei Eukaryoten-Vesikeln. Gelegentliche Ausnahmen (A-Typ bei bestimmten Bakterien-, F-Typ bei einzelnen Archaeen-Spezies) werden auf horizontalen Gentransfer zurückgeführt.[2][3][4]
- FNullF1- alias F0F1-ATPase, A0A1-ATPase, V0V1-ATPase, FoFI ATP-
- Nicht korrekt. Der Name der Fo-Untereinheit leitet sich von Oligomycin ab. Das ist ein Gift (Inhibitor), das diese Untereinheit blockiert.
- mtATP-Synthase
- ATP-Synthase in Mitochondrien[5]
- CFoF1-ATP-Synthase
- ATP-Synthase in Chloroplasten
Die Richtung, in der eine ATP-Synthase wirkt, kann sich entsprechend dem chemisch-osmotischen Gleichgewicht auch umkehren – daran ändert auch die interne motorische Aktivität des Enzym nichts (siehe Gleichstrommaschine: in Abhängigkeit von der Richtung des Leistungsflusses: Dynamo versus Elektromotor). Die ATPasen arbeiten dann als echte membrangebundene Synthetasen. Beispiele sind V-ATPase, N-ATPase, P-ATPase (letztere mit anderem Aufbau, ohne rotierende Teile) und E-ATPase. Näheres über die verschiedenen Typen findet sich unter Membranständige ATPasen, Abschnitt: Typen.
Literatur
- P. D. Boyer, R. L. Cross, W. Momsen: A new concept for energy coupling in oxidative phosphorylation based on a molecular explanation of the oxygen exchange reactions. In: Proceedings of the National Academy of Sciences. Band 70, Nummer 10, Oktober 1973, S. 2837–2839, PMID 4517936, PMC 427120 (freier Volltext).
- Armen Y Mulkidjanian, Kira S Makarova, Michael Y Galperin, Eugene V Koonin: Inventing the dynamo machine: the evolution of the F-type and V-type ATPases. In: Nature Reviews Microbiology. 5, Nr. 11, 2007, S. 892–899. doi:10.1038/nrmicro1767. Dieser Artikel (PDF) bei Uni Osnabrück: Perspectives
- Armen Y Mulkidjanian, Michael Y Galperin, Eugene V Koonin: Co-evolution of primordial membranes and membrane proteins. In: Trends Biochem Sci. 4, Nr. 34, 2009, S. 206–215. doi:10.1016/j.tibs.2009.01.005. PMC 2752816 (freier Volltext).
- Nick Lane: Der Funke des Lebens – Energie und Evolution. Konrad Theiss Verlag, 2017 by WBG, ISBN 978-3-8062-3484-8. Englischer Originaltitel: Nick Lane: The Vital Question – Energy, Evolution, and the Origins of Complex Life. Ww Norton, 2015, ISBN 978-0-393-08881-6, armscoop.com (PDF). Textpassagen nahe Abbildung/Figure 10 (Struktur der ATP-Synthase).
- E. Hilario, J. P. Gogarten: Horizontal transfer of ATPase genes--the tree of life becomes a net of life. In: Biosystems, 1993, 31(2-3), S. 111–119. PMID 8155843
Weblinks
- Paul D. Boyer: ATP Synthase: eine prächtige molekulare Maschine.
- B. A. Feniouk: ATP Synthase FAQ. (englisch)
- Olaf Fritsche: Biophysiker entdeckten das älteste Rad der Welt.
- P. Gräber, A. Labahn: Die H+-ATPsynthase. (deutsch)
- Nobelpreis Chemie 1997 Präsentation. Nobel Media AB
- Oxidative phosphorylation – Reference pathway. Kanehisa Laboratories (KEGG); mit grafischer Darstellung im Zusammenhang zur Atmungskette und Links zu den EC-Nummern
- View Proteins belonging to: The H+- or NaH+-translocating F-type, V-type and A-type ATPase (F-ATPase) Superfamily. TCDB (englisch)
- Jennifer McDowall: Protein of the Month: ATP Synthase. ebi.ac.uk/interpro (englisch)
- Jassal/Reactome: Formation of ATP by chemiosmotic coupling.
- Gergely Pinke, Long Zhou, Leonid A. Sazanov: “Cryo-EM structure of the entire mammalian F-type ATP synthase”, in: Nature Structural & Molecular Biology, 14. September 2020, doi:10.1038/s41594-020-0503-8
- Complete Structure of ATPase, the World’s Smallest Turbine, Solved, auf: ScitechDaily vom 26. September 2020, Quelle: Institute of Science and Technology Austria
- Rätsel um riesige Protonenpumpe gelöst und Mystery of giant proton pump solved. EurekAlert!, 24. September 2020
Einzelnachweise
- S. Steigmiller, P. Turina, P. Gräber: The thermodynamic H+/ATP ratios of the H+-ATPsynthases from chloroplasts and Escherichia coli. In: Proceedings of the National Academy of Sciences. Band 105, Nummer 10, März 2008, S. 3745–3750, doi:10.1073/pnas.0708356105, PMID 18316723, PMC 2268821 (freier Volltext).
- Elena Hilario, Johann Peter Gogarten: Horizontal transfer of ATPase genes – the tree of life becomes a net of life. In: BioSystems. 31, Nr. 2–3, 1993, S. 111–119. doi:10.1016/0303-2647(93)90038-E. PMID 8155843 ScienceDirect University of Connecticut (PDF)
- G. Grüber et al.: ATP synthases from archaea: The beauty of a Molecular motor. In: Biochimica et Biophysica Acta (BBA) – Bioenergetics. Jahrgang 1837, Heft 6, 2014, S. 940–952, doi:10.1016/j.bbabio.2014.03.004 (englisch).
- Jennifer McDowall: ATP Synthase: The ATPase-Family (englisch)
- A. K. Srivastava, N. K. Ramaswamy, R. Mukopadhyaya, M. G. Jincy, S. F. D'Souza: Thiourea modulates the expression and activity profile of mtATPase under salinity stress in seeds of Brassica juncea. In: Annals of botany. Band 103, Nummer 3, Februar 2009, S. 403–410, doi:10.1093/aob/mcn229, PMID 19033283, PMC 2707324 (freier Volltext).