Cytochrom-c-Oxidase
Das Enzym Cytochrom-c-Oxidase (COX), genauer Cytochrom c: Sauerstoff-Oxidoreduktase (systematischer Name), Cytochrom-aa3-Komplex oder auch Komplex IV der mitochondrialen Atmungskette genannt, ist eine Oxidoreduktase. Der bei Bakterien aus drei, bei Eukaryoten aus dreizehn Untereinheiten bestehende Enzymkomplex katalysiert in einer gekoppelten Reaktion die Oxidation von Cytochrom c mit der Reduktion von Sauerstoff zu Wasser und dem Transport von Protonen über eine biologische Membran.
| Cytochrom-c-Oxidase | ||
|---|---|---|
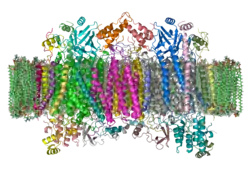
| ||
| Bändermodell eines Cytochrom-c-Oxidase-Dimers vom Rind in der Membran nach PDB 1OCC | ||
| Transporter-Klassifikation | ||
| TCDB | 3.D.4.7.1 | |
| Bezeichnung | protonenübertragende COX | |
| Enzymklassifikation | ||
| EC, Kategorie | 1.9.3.1, Oxidoreduktase | |
| Reaktionsart | Redoxreaktion | |
| Substrat | 4 Cytochrom c (reduziert) + O2 + 8 H+(in) | |
| Produkte | 4 Cytochrom c (oxidiert) + 2 H2O + 4 H+(out) | |
| Vorkommen | ||
| Übergeordnetes Taxon | Lebewesen | |
| Übergeordnet |
| Atmungskette-Komplex |
| Gene Ontology |
|---|
| QuickGO |
Mutationen in den Genen, die für die einzelnen Untereinheiten codieren (MT-CO1, MT-CO2, MT-CO3) können seltene Erbkrankheiten verursachen, die unter Cytochrom-c-Oxidasemangel (MT-C4D) zusammengefasst werden, nämlich Optikus-Neuropathie Typ Leber (LHON), rekurrente Myoglobinurie, sowie mitochondriale nichtsyndromale sensorineurale Schwerhörigkeit.[1][2][3]
Die Cytochrom-c-Oxidase gehört zur Superfamilie der Häm-Kupfer-Oxidasen, die bei allen aerob atmenden Organismen den terminalen Elektronenakzeptor der Atmungskette darstellen. Sie sind für nahezu sämtlichen Sauerstoffverbrauch der atmenden Organismen verantwortlich. Die Oxidasen sind bei Eukaryoten in der inneren Mitochondrienmembran, bei Prokaryoten in der inneren Zellmembran eingelagert. Varianten der Cytochrom-c-Oxidase kommen in der Zellmembran aerober Bakterien vor. Diese enthalten zum Teil modifizierte Kofaktoren (Häm-Varianten), oder verwenden andere Elektronendonoren als Cytochrom c (Chinol-Oxidasen z. B. in Escherichia coli). Sie besitzen sämtlich große strukturelle und funktionelle Homologie und enthalten im aktiven Zentrum eine Häm-Gruppe und ein Kupfer-Ion.
Katalysierter Transport
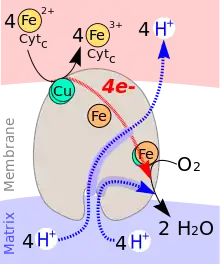
Die Transportgleichung lautet:[4]
4 Cytc(Fe2+) + O2 + 8 H+innen → 4 Cytc(Fe3+) + 2 H2O + 4 H+außen
Die Funktion der Cytochrom-c-Oxidase besteht aus der
- Reduktion von Sauerstoff zu Wasser (biologische Knallgasreaktion) mittels Elektronen vom Cytochrom c und dem
- Transport von Protonen (Protonenpumpe) über die biologische Membran.
Während des katalytischen Zyklus der Cytochrom-c-Oxidase wird ein Molekül Sauerstoff (O2) zu zwei Molekülen Wasser (H2O) reduziert. Als Reduktionsmittel werden vier Elektronen (e−) von vier Molekülen Cytochrom c sowie Protonen (H+) für die Wasserbildung aus dem Innenraum des Mitochondriums (Matrix) gebraucht. Die bei der Reduktion von Sauerstoff zu Wasser freigesetzte Energie wird zum Aufbau eines Protonengradienten über die innere Mitochondrienmembran genutzt. Pro Reaktionszyklus werden vier Protonen aus dem Innenraum des Mitochondriums in den Intermembranraum transportiert.
Über das komplexe Zusammenspiel der ablaufenden Sauerstoffchemie, der Elektronentransferreaktionen, sowie den Protonenaufnahme- und Pumpschritten und deren genauen zeitlichen Abfolgen konnten in letzter Zeit einige Vorstellungen entwickelt werden. In Abb. 3 und 4 finden sich Modelle über die Abfolge der Redoxreaktionen und der Mechanik des Pumpmechanismus.[5][6]
Gesamtzyklus bei der Reduktion von O2 zu 2 H2O.
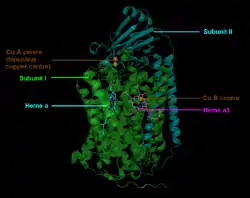
Die Cytochrom c Oxidase ist ein Membranprotein. Es enthält verschiedene metallische prosthetische Gruppen:[7]
- 2 Häm-Gruppen
- Die Cytochrome a und a3
- Zwei Kupfer-Zentren (CuA und CuB).
Das CuA befindet sich an der Bindungsstelle für Cytochrom c. Nach dessen Oxidation wird das Elektron über Cytochrom a zu Cytochrom a3 weitergeleitet, wo es zusammen mit CuB die O2-Reduktion katalysiert. Der Ablauf ist im Einzelnen in Abb. 3 dargestellt.
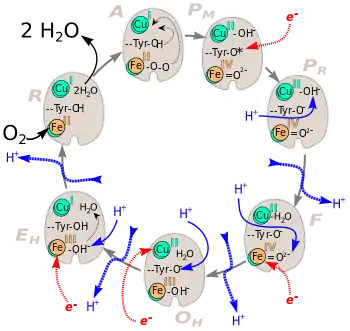
- A: Am Fe(II) des Hämins a3 ist koordinativ ein O2 Molekül gebunden.
- → Es beginnen zwei Umlagerungen (dünne schwarze gestrichelte Pfeile).
- PM: Fe(II) ist zu Fe(IV)=O2− und Cu(I) zu Cu(II)-OH− oxidiert. Das Wasserstoffatom stammt von einem Tyrosin-Rest, der dabei zum Radikal (*) wird.
- → Ein e− lagert sich an das Radikal an.
- PR: Das Radikal ist zum Tyrosin-Anion reduziert.
- → Das erste H+ wird von innen nach außen transloziert (blauer gestrichelter Pfeil). Simultan wird Cu(II)-OH− protoniert.
- F: Bei der Protonierung ist Cu(II) mit koordinativ gebundenem Wasser entstanden.
- → Das zweite H+ wird transloziert. Fe(IV)=O2− wird reduziert und protoniert. Durch Abspaltung von Cu(II) wird das erste Wassermolekül frei.
- OH: Durch Protonierung und Reduktion von Fe(IV)=O2− ist Fe(III)-OH− entstanden.
- → Dritte Protonenstransloktion. Reduktion von Cu(II) und Protonierung des Tyrosin-Anions.
- EH: Cu(II) ist zu Cu(I) reduziert und das Tyrosin-Anion zum Tyr-OH protoniert.
- → Vierte H+-Translokation. Fe(III)-OH− wird reduziert und die OH−-Gruppe protoniert.
- R: Das Zentralatom des Hämins a3 ist zu Fe(II) reduziert. Das zweite H2O ist entstanden.
- → Die beiden entstandenen Wassermoleküle werden ausgeschieden. Ein Sauerstoffatom lagert sich koordinativ am Fe(II) an.
Mechanismus der Protonentranslokation
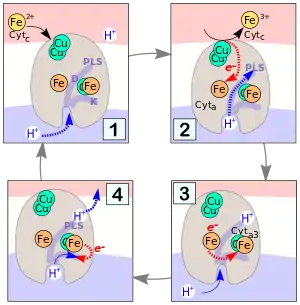
Das Enzym leistet nicht nur in Mitochondrien, sondern auch in einer Vielzahl von aeroben Prokaryoten einen wichtigen Beitrag zum Energiestoffwechsel, indem es als Protonenpumpe zum Aufbau eines chemiosmotischen Potentials beiträgt.
Bei der Oxidation von vier Cytochromc-Molekülen werden 4 Protonen transloziert. Jede dieser Reaktionen läuft (vereinfacht gesagt) nach demselben Mechanismus ab. Er ist rechts in Abb. 4 in vier Phasen (1–4) dargestellt.
- 1: Die Cytochrom c Oxidase ist in die Zellmembran (weiß) eingebettet. An der Außenseite (rötlich) lagert sich reduziertes Cytc an einem Kupferzentrum an. An der Zellplasma-Seite (unten) der Membran befinden sich die Eingänge zu zwei Protonen-Kanälen D und K.
- 2: Nach Abgabe eines Elektrons vom Cytc an das Cu-Zentrum wird das Elektron an das Fe-Zentrum von Cyta weiter geleitet. Der D-Kanal wird geöffnet und durch ihn dringt ein Proton bis zur „Protonenladestelle“ (PLS) ein.
- 3: Das Elektron gelangt vom Cyta zum Cyta3-Zentrum, während ein Proton in den K-Kanal eindringt. Das PLS-Proton oben im Enzym erfährt dabei eine deutliche Erhöhung seiner Acidität (von pK=11 zu pK=5!).
- 4: Das PLS-Proton wird an der Außenseite der Membran abgegeben. Synchron dazu geht das H+ im K-Kanal eine der links dargestellten Protonierungsreaktionen ein, während das e− vom Cyta3 in einer der vier in Abb. 3 dargestellten Reduktionen verbraucht wird.
Struktur
Der mitochondriale Enzymkomplex IV in Säugetieren besteht aus 13 Untereinheiten,[8] von denen die Untereinheiten I–III mitochondrial und die weiteren Untereinheiten IV–XIII vom Nukleus kodiert sind. Die Untereinheit I besitzt die drei redoxaktiven Metallzentren Häm a, Häm a3 und CuB. Häm a3 und CuB bilden zusammen das katalytisch aktive Zentrum, an dem Sauerstoff gebunden und zu Wasser reduziert wird. Die Untereinheit II besitzt das redoxaktive Metallzentrum CuA, das Elektronen vom Cytochrom c aufnimmt, die dann zum Häm a und weiter zum Häm a3 transferiert werden.
Der Komplex IV beim Menschen im Detail:
| Anzahl | Gen-Name | UniProt | Größe (aa) |
OMIM | Kommentar |
|---|---|---|---|---|---|
| 1 | MT-CO1 | P00395 | 513 | 516030 | Katalytische Untereinheit; Häm, Cu2+; Membrandomänen; pathologische Mutationen |
| 2 | MT-CO2 | P00403 | 227 | 516040 | Cu2+; Membrandomänen; pathologische Mutationen |
| 1 | MT-CO3 | P00414 | 261 | 516050 | Membrandomänen; pathologische Mutationen |
| 1 | COX4I1 | P13073 | 169 | 123864 | |
| 1 | COX5A | P20674 | 150 | 603773 | Häm A |
| 1 | COX5B | P10606 | 98 | 123866 | Zn2+ |
| 1 | COX6A1 | P12074 | 85 | 602072 | |
| 1 | COX6B1 | P14854 | 85 | 124089 | Pathologische Mutationen |
| 1 | COX6C1 | P09669 | 74 | 124090 | Membrandomäne |
| 1 | COX7A2L | O14548 | 59 | 605771 | |
| 1 | COX7B | P24311 | 56 | 603792 | Membrandomäne |
| 1 | COX7C | P15954 | 47 | 603774 | Membrandomäne |
| 1 | COX8A | P10176 | 44 | 123870 | Membrandomäne |
Inhibitoren
Cyanide, Kohlenmonoxid, Schwefelwasserstoff und Azide sind Inhibitoren der Cytochrom-c-Oxidase. Sie blockieren die Bindungsstelle für Sauerstoff im aktiven Zentrum.
Nachweis
Zum Nachweis des Enzyms Cytochrom-c-Oxidase in Zellen wird der Oxidase-Test verwendet.
Alternative Bezeichnungen
- Cytochrom c: O2 Oxidoreduktase
- Cytochrom-Oxidase (Zytochromoxidase)
- Komplex IV der Atmungskette
- Cytochrom-aa3-Komplex nach David Keilin
- Atmungsferment nach Otto Warburg (Nobelpreis für die Entdeckung)
- Indophenol-Oxidase nach Paul Ehrlich
Weitere Enzymkomplexe der Atmungskette:
- Komplex I auch NADH-Dehydrogenase
- Komplex II auch Succinat-Dehydrogenase
- Komplex III auch Cytochrom-c-Reduktase
- Komplex V auch ATP-Synthase
Literatur
- O. Warburg: Über Eisen, den sauerstoffübertragenden Bestandteil des Atmungsfermentes. In: Biochemische Zeitschrift. Band 152, 1924, S. 479–494, doi:10.1002/cber.19250580603.
- David Keilin: On cytochrome, a respiratory pigment, common to animals, yeast, and higher plants. In: Proc. Royal Soc. London. Series B. Band 98, 1925, S. 312–339, JSTOR 81121.
- T. Tsukihara, H. Aoyama, E. Yamashita u. a.: The whole structure of the 13-subunit oxidized cytochrome c oxidase at 2.8 A. In: Science. Band 272, Nr. 5265, Mai 1996, S. 1136–1144, doi:10.1126/science.272.5265.1136, PMID 8638158.
- S. Iwata, C. Ostermeier, B. Ludwig u. a.: Structure at 2.8 A resolution of cytochrome c oxidase from Paracoccus denitrificans. In: Nature. Band 376, Nr. 6542, August 1995, S. 660–669, doi:10.1038/376660a0, PMID 7651515.
- F. Diaz: Cytochrome c oxidase deficiency: patients and animal models. In: Biochim Biophys Acta. Band 1802, Nr. 1, Januar 2010, S. 100–110, doi:10.1016/j.bbadis.2009.07.013, PMID 19682572.
Weblinks
- B Jassal: Electron transfer from reduced cytochrome c to molecular oxygen. In: reactome.org. EBI, 28. Juni 2005, abgerufen am 4. Oktober 2010.
Einzelnachweise
- C. Orssaud: Optikus-Neuropathie Typ Leber. In: orpha.net. Orphanet, November 2003, abgerufen am 4. Oktober 2010.
- Genetic recurrent myoglobinuria. In: orpha.net. Orphanet, März 2010, abgerufen am 4. Oktober 2010.
- Mitochondrially inherited nonsyndromic sensorineural deafness. In: Online Mendelian Inheritance in Man. (englisch)
- 3.D.4 The Proton-translocating Cytochrome Oxidase (COX) Superfamily. In: TCDB. Saier Lab Bioinformatics, abgerufen am 4. Oktober 2010 (englisch).
- Y. C. Kim, M. Wikström, G. Hummer: Kinetic gating of the proton pump in cytochrome c oxidase. In: PNAS. 106/33, 2009, S. 13707–13712, doi:10.1073/pnas.0903938106.
- V. Sharma, G. Enkavi, I. Vattulainen, T. Róg, M. Wikström: Proton-coupled electron transfer and the role of water molecules in proton pumping by cytochrome c Oxidase. In: PNAS. 112/07, 2015, S. 2040–2045, doi:10.1073/pnas.1409543112.
- T. Tsukihara, H. Aoyama, E. Yamashita, T. Tomizaki, H. Yamaguchi, K. Shinzawa-Itoh, R. Nakashima, R. Yaono, S. Yoshikawa: Structures of metal sites of oxidized bovine heart cytochrome c oxidase at 2.8 A. In: Science. 269, 5227, 1995, S. 1069–1074. PMID 7652554.
- B. Kadenbach, J. Jarausch, R. Hartmann, P. Merle: Separation of mammalian cytochrome c oxidase into 13 poly-peptides by a sodium dodecyl sulfate-gel electrophoretic procedure. In: Anal. Biochem. 129, 1983, S. 517–521. PMID 6303162.