RNA-abhängige RNA-Polymerase
RNA-abhängige RNA-Polymerasen (englisch RNA-dependent RNA polymerase; RdRP oder RDR) sind Enzyme, die als Polymerasen die Synthese von Ribonukleinsäuren (RNA) aus Ribonukleotiden katalysieren anhand einer RNA-Vorlage (RNA-dependent). Sie werden auch als RNA-Replikase bezeichnet, soweit sie anhand eines RNA-Strangs eine hierzu komplementäre RNA aufbauen und diese wiederum als Matrize zur Vervielfältigung nutzen können.
| RNA-abhängige RNA-Polymerase | ||
|---|---|---|
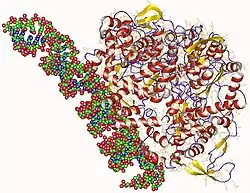 | ||
| Bänderdarstellung der RNA-abhängigen RNA-Polymerase, nach PDB 5H0R | ||
| Bezeichner | ||
| Externe IDs |
| |
| Enzymklassifikation | ||
| EC, Kategorie | 2.7.7.48, Transferase | |
| Substrat | Nukleosidtriphosphat + RNAn | |
| Produkte | Diphosphat + RNAn+1 | |
Bei verschiedenen RNA-Viren ist eine RdRP zur Vermehrung ihrer Erbinformation erforderlich, etwa beim Poliovirus. In eukaryotischen Zellen (von Tieren, Pflanzen und Pilzen) dienen RdRP dagegen im Zuge der RNA-Interferenz dem Aufbau doppelsträngiger RNA, die anschließend abgebaut wird; derart können sie auch der Abwehr einer Infektion mit RNA-Viren nützlich sein.
RNA-abhängige RNA-Polymerasen sind eine Gruppe der RNA-Polymerasen, eine andere sind DNA-abhängige RNA-Polymerasen, wie sie zur Transkription von DNA-Abschnitten benötigt werden. Daneben gibt es unabhängige RNA-Polymerasen, so bei der Polyadenylierung.
Eigenschaften
Die RNA-dependente RNA-Polymerase (RdRP) ist in RNA-Viren ein essenzielles Protein, das in Genomen von Viren codiert ist, die RNA und keine DNA beinhalten.[1][2] Es katalysiert die Synthese eines RNA-Stranges komplementär zu einer anderen RNA-Matrize. Die Replikation der RNA ist ein zweistufiger Prozess. Die Initiation der RNA-Synthese beginnt am oder in der Nähe des 3′-Endes durch einen Primer-unabhängigen (de novo) oder Primer-abhängigen Mechanismus, der das Protein VPg (viral protein genome-linked) als Primer benutzt. Die de-novo-Initiation besteht aus dem Anhängen eines Nukleosidtriphosphats am 3′-OH-Ende des ersten initiierenden NTP. Während der Elongation wird der Nukleotidyltransfer von NTPs wiederholt, um einen komplementären RNA-Strang zu generieren.[3][4] Im Gegensatz zu viralen DNA-Polymerasen besitzen RdRP keine Korrekturfunktion und replizieren daher RNA mit einer deutlich höherer Mutationsrate.[5] Dies äußert sich sowohl in einem höheren Anteil defekter Kopien als auch in der Bildung von Quasispezies, was eine Immunantwort erschwert (Immunevasion).[6]
Die RdRP vieler Eukaryoten sind in der RNA-Interferenz involviert; diese amplifizieren microRNA und small temporal RNA und produzieren mithilfe von siRNA als Primer doppelsträngige RNA.[7]
Geschichte
Virale RdRP wurden in den frühen 1960er Jahren anhand der Studien über das Mengovirus und Poliovirus entdeckt, als beobachtet wurde, dass die RdRP-beinhaltenden Viren keine Reaktion gegenüber Actinomycin D aufzeigten, ein Zytostatikum, das die zelluläre DNA-abhängige RNA-Synthese hemmt. Anhand dieser fehlenden Reaktion wurde vermutet, dass ein virusspezifisches Enzym vorhanden sein muss, das die RNA anhand einer RNA-Vorlage und nicht mittels einer DNA-Vorlage synthetisiert.
Die bekannteste RdRP ist die des Poliovirus. Das virale Genom besteht aus RNA, die mithilfe der rezeptorvermittelten Endozytose in die Zelle gelangt. Von dort aus ist die RNA unmittelbar in der Lage, als Vorlage für die komplementäre RNA-Synthese zu dienen. Der komplementäre Strang ist dann selbst in der Lage, als Vorlage für die Produktion neuer viraler Genome zu wirken, die weiter verpackt und aus der Zelle freigesetzt werden, um mehr Wirtszellen zu infizieren. Der Vorteil dieser Methode der Replikation ist, dass keine DNA als Zwischenstufe gebildet wird und somit die Reaktion schneller und effizienter abläuft. Der Nachteil besteht darin, dass es keine „Sicherheits-DNA-Kopie“ gibt, die im Falle eines Informationsverlusts die Informationen erneut abrufbar machen kann.
Viele RdRP befinden sich eng an Membranen und sind deshalb schwer zu erforschen. Die bekanntesten RdRP sind 3Dpol (Poliovirus), VSIV L (Vesicular stomatitis virus) und NS5B (Hepatitis-C-Virus).
Struktur
Alle RNA-abhängigen RNA-Polymerasen und viele DNA-abhängige Polymerasen besitzen eine Tertiärstruktur, die der Gestalt einer rechten Hand gleicht. Die Domänen der Enzyme werden deshalb in „Finger“, „Handfläche“ und „Daumen“ unterteilt.[8] Nur die „Handfläche“ besteht aus einem viersträngigen, antiparallelen β-Faltblatt mit zwei α-Helices. Außerdem umfasst die „Handfläche“ drei gut konservierte Motive (A, B und C). Motiv A (D-x(4,5)-D) und Motiv C (GDD) befinden sich räumlich nebeneinander; an die Asparaginreste dieser Motive können sich Mg2+ und/oder Mn2+ als Cofaktoren binden. Die Asparaginreste des Motivs B beteiligen sich an der Selektion von bestimmten Ribonukleosidtriphosphaten mithilfe dNTPs und bestimmen somit, ob RNA anstelle von DNA synthetisiert wird.[9]
Die Positionen der Domänen[10] und die Tertiärstruktur des aktiven Zentrums vieler RdRP, sogar die mit einer insgesamt niedrigen Sequenzhomologie, sind konserviert. Das aktive Zentrum wird durch mehrere Motive geformt, die eine Vielzahl von konservierten Aminosäuren enthalten. Nach Bindung der RNA schließen sich „Finger“ und „Daumen“ der RdRP zu einem Ring und das aktive Zentrum liegt auf der Innenseite des Rings.[5] Alle RdRP in (+)-strängigen RNA-Viren sind mit der ER- oder Zellmembran assoziiert.[11] RdRP sind strukturell mit RNA-abhängigen DNA-Polymerasen (Reverse Transkriptasen) verwandt.[5] Ein Inhibitor vieler RdRP ist Favipiravir.
Konservierte Regionen der RdRP[5]
| Konservierte Region | Position relativ zur Poliovirus-RNA | Aminosäuresequenz[12] | Funktion |
|---|---|---|---|
| Finger (G) | 113 | STSAGYPY | Bindung der RNA-Vorlage |
| Finger (F) | 153 | PLVTYVKDELRSKTKVEQGKSRLIEA | Bindung der RNA-Vorlage und NTP |
| Finger (I) | 107 | LEALDL | Bindung der RNA-Vorlage |
| Finger (II) | 184 | SVAMRMAFGNLIAAFHK | Bindung der RNA-Vorlage |
| Handfläche (A) | 229 | LFAFDYTGYDAS | Bindung der 2'-Hydroxygruppe des NTP und des ersten Cofaktors |
| Handfläche (B) | 293 | TSIFNSMINNLIIRTLLLKT | Bindung der RNA-Vorlage und die Nukleinbase des NTP |
| Handfläche (C) | 323 | MIAYGDDVIAS | Bindung eines weiteren Cofaktors und bei manchen RdRP den Primer |
| Handfläche (D) | 338 | VDASLLAQSGKDYGLTMTPADKSAT | Bindung der Triphosphatgruppe des NTP |
| Handfläche (E) | 363 | FETVTWENVTFLKRFFRA | Bindung des 3'-Endes des entstehenden RNA-Strangs |
| Daumen (III) | 405 | KDPRNTQDHVRSLCLL | Bindung des entstehenden RNA-Doppelstrangs |
Klassifizierung
RNA-abhängige RNA-Polymerasen der Viren werden in vier Familien unterteilt:
- RdRP in Viren mit (+)-strängigen oder doppelsträngiger RNA: neben den Retroviren drei weitere RdRP-Unterfamilien:
- Retroviridae (Retroviren)
- alle (+)-strängigen eukaryotischen RNA-Viren ohne DNA als Zwischenstufe, u. a. Caliciviridae,[13] Picornaviridae[14]
- alle RNA-Phagen, das sind Fiersviridae (Phagen mit (+)ssRNA) und Cystoviridae (Phagen mit dsRNA)[15]
- die dsRNA-Virus-Familien Reoviridae, Totiviridae, Hypoviridae [en], Partitiviridae [en], Birnaviridae
- RdRP in Mononegavirales ((–)-strängige RNA-Viren mit nicht-segmentierten Genomen), z. B.
Filoviridae,[16] Paramyxoviridae[17] (u. a. mit Morbillivirus),[18] Hepatitis-A- und Hepatitis-C-Virus;[19] InterPro: IPR016269 (englisch) - RdRP in (–)-strängigen RNA-Viren mit segmentierten Genomen, z. B.
Bunyavirales, Orthomyxoviridae; InterPro: IPR007099 (englisch) - RdRP in den doppelsträngigen RNA-Viren Birnaviridae; InterPro: IPR007100 (englisch)
Einzelnachweise
- E. V. Koonin, A. E. Gorbalenya, K. M. Chumakov: Tentative identification of RNA-dependent RNA polymerases of dsRNA viruses and their relationship to positive strand RNA viral polymerases. In: FEBS letters. Band 252, Nummer 1–2, Juli 1989, S. 42–46, PMID 2759231.
- P. M. Zanotto, M. J. Gibbs, E. A. Gould, E. C. Holmes: A reevaluation of the higher taxonomy of viruses based on RNA polymerases. In: Journal of virology. Band 70, Nummer 9, September 1996, S. 6083–6096, PMID 8709232, PMC 190630 (freier Volltext).
- Z. Jin, V. Leveque, H. Ma, K. A. Johnson, K. Klumpp: Assembly, purification, and pre-steady-state kinetic analysis of active RNA-dependent RNA polymerase elongation complex. In: The Journal of biological chemistry. Band 287, Nummer 13, März 2012, S. 10674–10683, doi:10.1074/jbc.M111.325530, PMID 22303022, PMC 3323022 (freier Volltext).
- C. C. Kao, P. Singh, D. J. Ecker: De novo initiation of viral RNA-dependent RNA synthesis. In: Virology. Band 287, Nummer 2, September 2001, S. 251–260, doi:10.1006/viro.2001.1039, PMID 11531403 (Review).
- Katsuhiko S. Murakami: Nucleic Acid Polymerases. Springer Science & Business Media, 2013, ISBN 978-3-642-39796-7, S. 323.
- A. V. Bordería, K. Rozen-Gagnon, M. Vignuzzi: Fidelity Variants and RNA Quasispecies. In: Current topics in microbiology and immunology. Band 392, 2016, S. 303–322, doi:10.1007/82_2015_483, PMID 26499340.
- L. M. Iyer, E. V. Koonin, L. Aravind: Evolutionary connection between the catalytic subunits of DNA-dependent RNA polymerases and eukaryotic RNA-dependent RNA polymerases and the origin of RNA polymerases. In: BMC structural biology. Band 3, Januar 2003, S. 1, PMID 12553882, PMC 151600 (freier Volltext).
- J. L. Hansen, A. M. Long, S. C. Schultz: Structure of the RNA-dependent RNA polymerase of poliovirus. In: Structure. Band 5, Nummer 8, August 1997, S. 1109–1122, PMID 9309225.
- D. W. Gohara, S. Crotty, J. J. Arnold, J. D. Yoder, R. Andino, C. E. Cameron: Poliovirus RNA-dependent RNA polymerase (3Dpol): structural, biochemical, and biological analysis of conserved structural motifs A and B. In: The Journal of biological chemistry. Band 275, Nummer 33, August 2000, S. 25523–25532, doi:10.1074/jbc.M002671200, PMID 10827187.
- E. K. O'Reilly, C. C. Kao: Analysis of RNA-dependent RNA polymerase structure and function as guided by known polymerase structures and computer predictions of secondary structure. In: Virology. Band 252, Nummer 2, Dezember 1998, S. 287–303, doi:10.1006/viro.1998.9463, PMID 9878607 (Review).
- Chundi L. Mandahar: Multiplication of RNA Plant Viruses. Springer Science & Business Media, 2006, ISBN 978-1-402-04725-1, S. 126.
- unterstrichen = konserviert, fett = konserviert in allen RdRP
- J. Deval, Z. Jin, Y. C. Chuang, C. C. Kao: Structure(s), function(s), and inhibition of the RNA-dependent RNA polymerase of noroviruses. In: Virus research. Band 234, April 2017, S. 21–33, doi:10.1016/j.virusres.2016.12.018, PMID 28041960.
- C. Ferrer-Orta, D. Ferrero, N. Verdaguer: RNA-Dependent RNA Polymerases of Picornaviruses: From the Structure to Regulatory Mechanisms. In: Viruses. Band 7, Nummer 8, August 2015, S. 4438–4460, doi:10.3390/v7082829, PMID 26258787, PMC 4576190 (freier Volltext).
- S. Alphonse, R. Ghose: Cystoviral RNA-directed RNA polymerases: Regulation of RNA synthesis on multiple time and length scales. In: Virus research. Band 234, April 2017, S. 135–152, doi:10.1016/j.virusres.2017.01.006, PMID 28104452, PMC 5476504 (freier Volltext).
- B. Martin, B. Canard, E. Decroly: Filovirus proteins for antiviral drug discovery: Structure/function bases of the replication cycle. In: Antiviral research. Band 141, Mai 2017, S. 48–61, doi:10.1016/j.antiviral.2017.02.004, PMID 28192094.
- R. Cox, R. K. Plemper: The paramyxovirus polymerase complex as a target for next-generation anti-paramyxovirus therapeutics. In: Frontiers in microbiology. Band 6, 2015, S. 459, doi:10.3389/fmicb.2015.00459, PMID 26029193, PMC 4428208 (freier Volltext).
- J. Sourimant, R. K. Plemper: Organization, Function, and Therapeutic Targeting of the Morbillivirus RNA-Dependent RNA Polymerase Complex. In: Viruses. Band 8, Nummer 9, September 2016, S. , doi:10.3390/v8090251, PMID 27626440, PMC 5035965 (freier Volltext).
- A. A. Eltahla, F. Luciani, P. A. White, A. R. Lloyd, R. A. Bull: Inhibitors of the Hepatitis C Virus Polymerase; Mode of Action and Resistance. In: Viruses. Band 7, Nummer 10, September 2015, S. 5206–5224, doi:10.3390/v7102868, PMID 26426038, PMC 4632376 (freier Volltext).