PCR-Optimierung
Die PCR-Optimierung umfasst die gezielte Veränderung der Reaktionsbedingungen einer Polymerasekettenreaktion.
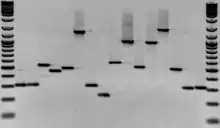
Optimierungsparameter
Eine Optimierung PCR-verwandter Methoden umfasst die gezielte Veränderung verschiedener Parameter.
- Wahl einer geeigneten thermostabilen DNA-Polymerase
- Entwurf der Sequenz des Primers (Primerdesign)
- Veränderung der Konzentrationen von Substraten (DNA-Vorlage, Primer-Vorlage, dNTP-Konzentration)
- Veränderung der Konzentrationen von Produkten (mehr kopierte DNA, korrektes Produkt, weniger Pyrophosphat),
- Vermeidung von Störsubstanzen (z. T. Substrat-ähnliche Inhibitoren)
- Steuerung der Umgebungsbedingungen (Temperatur, Thermostabilität, Ionenstärke, Osmolarität, Cofaktoren).
Polymerase
Die Wahl einer geeigneten DNA-Polymerase hat einen Einfluss auf die Synthesemenge und Fehlerrate.
Primer
Das Primerdesign umfasst die Auswahl geeigneter DNA-Sequenzen für die Primer und der darauf basierenden Primerhybridisierungs-Temperatur (synonym Annealing-Temperatur) in der PCR. Die Annealing-Temperatur der PCR liegt meistens zwei Grad Celsius unter der Schmelztemperatur der Primer, was einen Kompromiss zwischen einer möglichst vollständigen Primerhybridisierung und einer möglichst hohen Spezifität der Primerbindung darstellt.
Substratkonzentration
Die Endkonzentration der Primer während der PCR liegt zwischen 0,1 μM und 1μM (meistens 0,4 μM), die Konzentration an dNTPs beträgt für jede der vier Nukleinbasen etwa 100 – 1000 μM (meistens je 400 μM) und die Massenkonzentration der DNA von 1 bis 20 ng pro Mikroliter des PCR-Ansatzes (meistens 200 ng pro Ansatz).
Temperaturzyklus
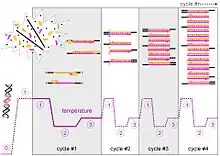
Die Dissoziation (das Schmelzen) der doppelsträngigen DNA wird meistens bei 95 °C durchgeführt. Daraufhin erfolgt die Hybridisierung (das Annealing) bei einer Primer-abhängigen Temperatur, zuletzt erfolgt die Synthese der DNA am jeweiligen Optimum der jeweiligen thermostabilen DNA-Polymerase bei 72 °C (Typ-A-Polymerasen) bzw. 68 °C (Typ-B-Polymerasen).
Fehlerrate
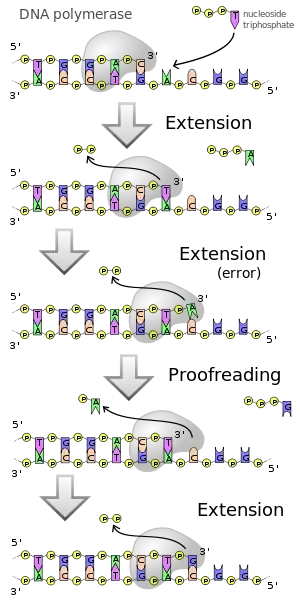
Die Fehlerraten verschiedener Polymerasen (engl. fidelity) sind bekannt und beschrieben worden. Bakterielle thermostabile DNA-Polymerasen (Typ A) besitzen meist eine höhere Fehlerrate als Archaeische (Typ B), was an der proof reading-Exonukleaseaktivität der B-Typ-Polymerasen liegt. Die Fehlerrate der Taq-Polymerase beträgt 8 · 10−6 Fehler pro Basenpaar, die der KOD-Polymerase 3,5 · 10−6 Fehler pro Basenpaar, die der Tli-Polymerase und der Herculase 2,8 · 10−6 Fehler pro Basenpaar, die der Pfu-Polymerase 1,3 · 10−6 Fehler pro Basenpaar und die der Pfu Ultra 4,3 · 10−7 Fehler pro Basenpaar.[1][2]
Syntheserate
Die Gesamt-Synthesegeschwindigkeit einer DNA-Polymerase hängt unter anderem von der Basissyntheserate und der Prozessivität der Polymerase sowie der Anwesenheit von Hemmstoffen (z. B. hemmende Produkte, PCR-Inhibitoren) ab.
Die Basissyntheseraten verschiedener Polymerasen (engl. productivity) sind verglichen worden.[2] Die Syntheserate der Taq-Polymerase liegt bei etwa 60 Basenpaaren pro Sekunde. Unter den unmodifizierten thermostabilen DNA-Polymerasen liegt nur die Syntheserate der KOD-Polymerase über 100 Basenpaaren pro Sekunde (circa 120 bp/s).[3] Unter den modifizierten thermostabilen DNA-Polymerasen sind verschiedene Mutationen beschrieben worden, die die Syntheserate steigern.[4][5] Die KOD-Polymerase und einige modifizierte thermostabile DNA-Polymerasen (iProof, Pfu Ultra, Phusion, Velocity oder Z-Taq) werden aufgrund ihrer hohen Syntheserate zu einer PCR-Variante mit kürzeren Amplifikationszyklen eingesetzt (Fast-PCR, High-speed PCR).
Die Prozessivität (engl. processivity) beschreibt die durchschnittliche Anzahl an Basenpaaren, bevor eine Polymerase von der DNA-Vorlage (engl. template) abfällt. Die Prozessivität der verwendeten Polymerase begrenzt den maximalen Abstand des Primers zur Sonde in der real time quantitative PCR. Die Prozessivität einer Taq-Polymerase liegt bei etwa 200 Basenpaaren.
Beim Erhitzen der dNTPs in wässrigen Lösungen desaminieren die Cytosin-Reste im dCTP kontinuierlich zu Uracil-Resten, welche die Syntheserate der DNA-Polymerasen herabsetzen.[6] Um dieser als dUTP-Vergiftung bezeichneten Herabsetzung entgegenzuwirken, wird bei Produkten von über 5 Kilobasen gelegentlich bei einer PCR mit archaeischen Polymerasen eine thermostabile dUTPase hinzugesetzt.[2][7][8] Analog desaminieren parallel auch die Adenosin-Reste im dATP zu Desoxyinosintriphosphat (dITP), was ebenfalls zu einer Hemmung der archaeischen DNA-Polymerasen führen kann. Daher kann auch eine thermostabile dITPase zur PCR mit archaeischen DNA-Polymerasen hinzugesetzt werden.[9]
Da die DNA-Polymerase die DNA-Kettenverlängerung unter Abspaltung von Pyrophosphat katalysiert, kann die Produktkonzentration durch Zugabe einer thermostabilen Pyrophosphatase erhöht werden. Die Pyrophosphatase katalysiert die Hydrolyse des Pyrophosphats zu Phosphaten, wodurch weniger Produkthemmung entsteht, da die Gleichgewichtskonstante der Reaktion zu Gunsten der synthetisierten DNA verändert wird.[10][11]
Das Stoffel-Fragment ist eine um die Exonukleasefunktion gekürzte Taq-Polymerase, wodurch die Produktkonzentration und die Thermostabilität des Enzyms erhöht wird.
Falsch negative Ergebnisse
Wird eine PCR durchgeführt und im Ergebnis keine vervielfältigte DNA nachgewiesen, obwohl die nachzuweisende Sequenz tatsächlich vorhanden war, spricht man von einem falsch negativen Ergebnis.
Substratverweigerung
Die vor allem bei den archaeischen Polymerasen gelegentlich vorkommende „Verweigerung“ schwieriger Substrate (engl. fussiness) kann zu falsch negativen Ergebnissen führen, z. B. bei aDNA, DNA mit hohem GC-Anteil, genomischer DNA oder DNA mit Sekundärstrukturen. Durch Zugabe schwach chaotroper Moleküle wie Dimethylsulfoxid, Formamid, Trehalose oder Betain kann die Hemmung der Reaktion gemindert werden.[12][13][14][15] Der Einsatz von chaotropen Verbindungen kann die Schmelztemperatur der Primer um bis zu 5 °C absenken, weshalb die Annealing-Temperatur der PCR entsprechend angepasst werden muss. Eine Erhöhung der Konzentration an Magnesium-Ionen, als Cofaktor der thermostabilen DNA-Polymerasen, über 1,5 mM hinaus erhöht die Produktkonzentration auf Kosten der Fehlerrate.[16] Manche alkylierte Nukleinbasen können keine Basenpaarung mit den vier natürlichen Nukleinbasen eingehen, weshalb es an diesen Stellen zu einer Unterbrechung der Amplifikation kommt. Durch Zugabe bestimmter modifizierter Nukleinbasen kann eine Basenpaarung bei der PCR erfolgen.[17]
Inhibitoren
Verschiedene Substanzen können thermostabile DNA-Polymerasen in der PCR stören, z. B. Virostatika, Hämoglobin, Heparin, Polysaccharide, Hormone, IgG, Lactoferrin, Myoglobin, Gallsäuren und ihre Salze, Harnsäure und ihre Salze, Phenol, Polyphenole, Proteasen, divalente Kationen außer Magnesium, EDTA und andere Chelatoren, Huminsäuren, Tonerde,[18] Kollagen, Hämatine, Indigo, Melanin und Tannine.[19][20] Im Allgemeinen erfordern Proben mit diesen Stoffen eine weitere DNA-Reinigung vor einer Verwendung in der PCR. Bei Vorhandensein höherer Konzentrationen an Polyphenolen kann Polyvinylpyrrolidon hinzugegeben werden, das diese bindet.[21] Ebenso wird bovines Serumalbumin zur Bindung von Inhibitoren eingesetzt.[22]
Falsch positive Ergebnisse
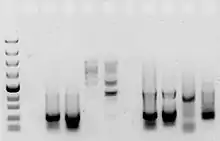
Ein falsch positives Ergebnis ist bei einer PCR eine Erzeugung von PCR-Produkten, die nicht der gewünschten und zu vervielfältigenden DNA-Sequenz entsprechen. Sie führt gegebenenfalls zu unerwünschten Banden in einem Agarosegel.
Kontaminationen
DNA-Kontaminationen und eine unspezifische Hybridisierung der Primer vor und während der PCR können zu falsch positiven Ergebnissen führen. Insbesondere Kontaminationen sind ein großes Problem. Häufig gelangt DNA von Personen, die an der Analyse beteiligt sind, oder die an der Herstellung von Komponenten mitgewirkt haben, in die Probe. Beim forensischen Einsatz (vgl. DNA-Analyse und Genetischer Fingerabdruck) machte das Heilbronner Phantom Schlagzeilen: Kriminalisten jagten lange Zeit eine vermeintliche Serientäterin, bis sich herausstellte, dass die DNA in Wirklichkeit von der Mitarbeiterin einer Zulieferfirma stammte. Problematisch können bei biologischen Proben auch bakterielle Verunreinigungen, Verunreinigungen durch Parasiten oder Nahrungsbestandteile und ähnliches sein. So wurde der wurmartige Organismus Xenoturbella längere Zeit fälschlich als Vertreter der Weichtiere angesehen, weil man DNA aus dem Darm, welche aus Nahrungsorganismen stammte, irrtümlich für die DNA des Wurms selbst hielt.[23] Besonders bei sehr alter, möglicherweise teilweise fragmentierter DNA, z. B. aus Fossilien, ist besondere Vorsicht geboten. Dies gilt besonders dann, wenn dem Menschen verwandte Sequenzen, z. B. das Erbgut des Neanderthalers, analysiert wird.
Gelegentlich wird zum enzymatischen Abbau von möglichen DNA-Kontaminationen im Reaktionsansatz eine Uracil-N-Glykosylase hinzugefügt, die im ersten Temperaturzyklus denaturiert wird.[24] Die Uracil-N-Glykosylase baut Uracil-enthaltende PCR-Produkte ab, die entstehen, wenn das Labor zuvor bei PCR zu reinen Nachweiszwecken von dTTP auf dUTP umgestellt hat und dafür Polymerasen vom Typ A verwendet hat. Dies kann jedoch bei niedrigen Konzentrationen an DNA-Ausgangsmaterial zu falsch negativen Ergebnissen führen.[25]
Hot-Start
Die Vorlagenspezifität der Polymerasen (engl. specificity) zur Vermeidung der Bindung an unerwünschte DNA-Vorlagen oder fehlgebundene Primer, und die daraus folgende Erzeugung unerwünschter Reaktionsprodukte vor Beginn des ersten Temperaturzyklus, wird durch die Verwendung von Hot-Start-Polymerasen gesteigert. Beispiele sind die mit einem Antikörper gehemmte Hot-Start Pfu Turbo, die Platinum Pfx als kommerzielle KOD-Polymerase mit hemmendem Antikörper und die Platinum Taq als Antikörper-gehemmte Taq-Polymerase.[2] Diese Polymerasen werden unter anderem durch Inaktivierung mit Formaldehyd, durch eine Komplexierung des Magnesiums mit Phosphaten[26] oder durch die Bindung eines Antikörpers an ihrem aktiven Zentrum gehemmt.[27][28] Bei Erhitzen auf 95 °C hydrolysiert das Formaldehyd,[29][30][31] alternativ werden die Magnesiumionen freigesetzt oder der Antikörper wird dabei denaturiert. Eine vierte Variante ist eine an Latex-Perlen über hydrophobe Effekte adsorbierte Polymerase, die bei zunehmender Temperatur in Lösung geht. Bei der fünften, ältesten, Variante wird der Reaktionsansatz ohne Polymerase mit Wachs überschichtet und die Polymerase auf das erkaltete Wachs gegeben. Bei Erhitzen schmilzt die Wachsschicht und die Polymerase vermischt sich mit dem Reaktionsansatz.[32]
Substratspezifität
Die Bevorzugung einzelner Nukleotide durch eine thermostabile DNA-Polymerase wird als Nukleotidspezifität (engl. bias ‚Vorliebe‘, ‚Voreingenommenheit‘) bezeichnet. Bei der PCR-basierten DNA-Sequenzierung mit Kettenabbruchsubstraten (Didesoxymethode) ist oftmals deren gleichmäßiger Einbau und somit eine gleichmäßige Erzeugung aller Kettenabbruchprodukte erwünscht, um eine höhere Sensitivität und eine leichtere Auswertung zu ermöglichen. Hierzu wurde eine KlenTaq-Polymerase durch Deletion erzeugt und durch ortsspezifische Mutagenese ein Phenylalanin an Position 667 gegen Tyrosin getauscht (kurz: F667Y) und als Thermo Sequenase bezeichnet.[33][10] Diese Polymerase kann auch für den Einbau fluoreszenzmarkierter Didesoxynukleotide verwendet werden.[34]
Eine Veränderung der Vorlagenspezifität von DNA zu RNA kann zur Erzeugung thermostabiler RNA-abhängiger DNA-Polymerasen genutzt werden, die auch als thermostabile reverse Transkriptasen bezeichnet werden. Konventionelle zur RT-PCR verwendete reverse Transkriptasen retroviralen Ursprungs, wie die AMV- und die MoMuLV-Reverse-Transkriptase, sind bei 95 °C nicht thermostabil. Bei den niedrigeren Temperaturen einer reversen Transkription mit diesen Enzymen kommen jedoch unspezifische Bindungen von Primern an die DNA-Vorlage und Sekundärstrukturen in der DNA-Vorlage vor, welche einerseits unerwünschte Produkte begünstigen und andererseits die Synthese des korrekten Produkts verhindern können. Allerdings kann die reverse Transkriptase von AMV bis zu 70 °C eingesetzt werden.[35] Zum Erreichen einer Thermostabilität bei 95 °C wurde zunächst die Vorlagenspezifität thermostabiler DNA-Polymerasen durch Austausch des Cofaktors (zweiwertige Magnesiumionen) gegen zweiwertige Manganionen herabgesetzt, so dass mit einer DNA-abhängigen thermostabilen Polymerase auch RNA in einer RT-PCR als Vorlage zur Synthese von DNA eingesetzt werden konnte.[36] Da die Syntheserate der Taq-Polymerase mit Manganionen relativ niedrig war, wurde bei dieser Variante der RT-PCR zunehmend die Tth-Polymerase eingesetzt.[37] Jedoch erhöhte die Zugabe von Manganionen auch die Anzahl fehlerhafter Produkte und erhöhte die notwendige Menge an Vorlagen-DNA, weshalb diese Enzyme heute kaum noch zur reversen Transkription eingesetzt werden. Diese Probleme konnten mit der thermostabilen 3173-Polymerase aus thermophilen Bakteriophagen vermieden werden, welche die hohen Temperaturen einer PCR für eine längere Zeit übersteht und RNA als Vorlage bevorzugt.[38]
Literatur
- J. Sambrook, T. Maniatis, D. W. Russel: Molecular cloning: a laboratory manual. Cold Spring Harbor Laboratory Press; 3rd edition (2001), ISBN 0-87969-577-3.
- Cornel Mülhardt: Der Experimentator: Molekularbiologie/Genomics, Springer 2008, ISBN 3-8274-2036-9.
Einzelnachweise
- J. Cline, J. C. Braman, H. H. Hogrefe: PCR fidelity of pfu DNA polymerase and other thermostable DNA polymerases. In: Nucleic Acids Res. (1996), Bd. 24(18), S. 3546-51. PMID 8836181; PMC 146123 (freier Volltext).
- Bahram Arezi, Weimei Xing, Joseph A. Sorge, Holly H. Hogrefe: Amplification efficiency of thermostable DNA polymerases. In: Analytical Biochemistry. Band 321, Nr. 2, 15. Oktober 2003, S. 226–235, doi:10.1016/S0003-2697(03)00465-2, PMID 14511688. PDF.
- Patentanmeldung EP1752534A1: Hochgeschwindigkeits-PCR unter Verwendung von Hochgeschwindigkeits-DNA-Polymerase. Angemeldet am 12. Mai 2005, veröffentlicht am 14. Februar 2007, Anmelder: Toyo Boseki, Erfinder: Masaya Segawa et Al.
- Patentanmeldung US2013034879A1: DNA Polymerases. Angemeldet am 2. August 2012, veröffentlicht am 14. Februar 2007, Anmelder: Fermentas UAB et Al, Erfinder: Remigijus Skirgaila et Al.
- Patentanmeldung US2009280539A1: DNA Polymerases and related methods. Angemeldet am 16. April 2009, veröffentlicht am 12. November 2009, Anmelder: Roche Molecular Systems Inc, Erfinder: Keith A. Bauer.
- Bernard A. Connolly: Recognition of deaminated bases by archaeal family-B DNA polymerases. In: Biochemical Society Transactions. Band 37, Nr. 1, Februar 2009, S. 65–68, doi:10.1042/BST0370065, PMID 19143603.
- Y. Cho, H. S. Lee, Y. J. Kim, S. G. Kang, S. J. Kim, J. H. Lee: Characterization of a dUTPase from the hyperthermophilic archaeon Thermococcus onnurineus NA1 and its application in polymerase chain reaction amplification. In: Mar Biotechnol (NY) (2007), Bd. 9(4), S. 450-8. PMID 17549447.
- Holly H. Hogrefe, Connie J. Hansen, Bradley R. Scott, Kirk B. Nielson: Archaeal dUTPase enhances PCR amplifications with archaeal DNA polymerases by preventing dUTP incorporation. In: Proceedings of the National Academy of Sciences. Band 99, Nr. 2, 22. Januar 2002, S. 596–601, doi:10.1073/pnas.012372799, PMID 11782527, PMC 117351 (freier Volltext).
- Y. J. Kim, Y. G. Ryu, H. S. Lee, Y. Cho, S. T. Kwon, J. H. Lee, S. G. Kang: Characterization of a dITPase from the hyperthermophilic archaeon Thermococcus onnurineus NA1 and its application in PCR amplification. In: Appl Microbiol Biotechnol. (2008), Band 79(4), S. 571-8. PMID 18438658.
- P. B. Vander Horn, M. C. Davis, J. J. Cunniff, C. Ruan, B. F. McArdle, S. B. Samols, J. Szasz, G. Hu, K. M. Hujer, S. T. Domke, S. R. Brummet, R. B. Moffett, C. W. Fuller: Thermo Sequenase DNA polymerase and T. acidophilum pyrophosphatase: new thermostable enzymes for DNA sequencing. In: Biotechniques (1997), Band 22(4), S. 758-62, 764-5. PMID 9105629. PDF (Memento des Originals vom 1. Februar 2014 im Internet Archive) Info: Der Archivlink wurde automatisch eingesetzt und noch nicht geprüft. Bitte prüfe Original- und Archivlink gemäß Anleitung und entferne dann diesen Hinweis..
- S. Y. Park, B. Lee, K. S. Park, Y. Chong, M. Y. Yoon, S. J. Jeon, D. E. Kim: Facilitation of polymerase chain reaction with thermostable inorganic pyrophosphatase from hyperthermophilic archaeon Pyrococcus horikoshii. In: Appl Microbiol Biotechnol. (2010), Band 85(3), S. 807–12. PMID 19882151.
- P. R. Winship: An improved method for directly sequencing PCR amplified material using dimethyl sulphoxide. In: Nucleic Acids Research. Band 17, Nr. 3, 11. Februar 1989, S. 1266, PMID 2922271, PMC 331767 (freier Volltext).
- S. A. Masoud, L. B. Johnson, F. F. White: The sequence within two primers influences the optimum concentration of dimethyl sulfoxide in the PCR. In: Genome Research. Band 2, Nr. 1, 8. Januar 1992, S. 89–90, doi:10.1101/gr.2.1.89, PMID 1490180.
- G. Sarkar, S. Kapelner, S. S. Sommer: Formamide can dramatically improve the specificity of PCR. In: Nucleic Acids Res. (1990), Band 18(24), S. 7465. PMID 2259646; PMC 332902 (freier Volltext).
- Andrej-Nikolai Spiess, Richard Ivell: A Highly Efficient Method for Long-Chain cDNA Synthesis Using Trehalose and Betaine. In: Analytical Biochemistry. Band 301, Nr. 2, 15. Februar 2002, S. 168–174, doi:10.1006/abio.2001.5474, PMID 11814287.
- K. A. Eckert, T. A. Kunkel: DNA polymerase fidelity and the polymerase chain reaction. In: Genome Research. Band 1, Nr. 1, 8. Januar 1991, S. 17–24, doi:10.1101/gr.1.1.17, PMID 1842916.
- Laura A. Wyss, Arman Nilforoushan, Fritz Eichenseher, Ursina Suter, Nina Blatter, Andreas Marx, Shana J. Sturla: Specific Incorporation of an Artificial Nucleotide Opposite a Mutagenic DNA Adduct by a DNA Polymerase. In: Journal of the American Chemical Society. 137, 2015, S. 30–33, doi:10.1021/ja5100542.
- C. Schrader, A. Schielke, L. Ellerbroek, R. Johne: PCR inhibitors - occurrence, properties and removal. In: J Appl Microbiol. 2012, doi:10.1111/j.1365-2672.2012.05384.x, PMID 22747964. PDF.
- K. L. Faber, E. C. Person, W. R. Hudlow: PCR inhibitor removal using the NucleoSpin® DNA Clean-Up XS kit. In: Forensic science international. Genetics. Band 7, Nummer 1, Januar 2013, S. 209–213, ISSN 1878-0326. doi:10.1016/j.fsigen.2012.06.013. PMID 22784879.
- Joseph Warren: A Review of PCR Inhibition and It’s Implications for Human Identity Testing. Association of Forensic DNA Analysts and Administrators. (PDF).
- Priyum K. Koonjul, Wolf F. Brandt, George G. Lindsey, Jill M. Farrant: Inclusion of polyvinylpyrrolidone in the polymerase chain reaction reverses the inhibitory effects of polyphenolic contamination of RNA. In: Nucleic Acids Research. Band 27, Nr. 3, 2. Januar 1999, S. 915–916, doi:10.1093/nar/27.3.915, PMID 9889293, PMC 148267 (freier Volltext).
- C. A. Kreader: Relief of amplification inhibition in PCR with bovine serum albumin or T4 gene 32 protein. In: Applied and environmental microbiology. Band 62, Nummer 3, März 1996, S. 1102–1106, PMID 8975603, PMC 167874 (freier Volltext).
- S. J. Bourlat, C. Nielsen, A. E. Lockyer, D. Timothy, J. Littlewood, M. J. Telford: Xenoturbella is a deuterostome that eats molluscs in Nature. Vol. 424, 2003, S. 925–928, nature.com
- M. Pruvost, T. Grange, E. M. Geigl: Minimizing DNA contamination by using UNG-coupled quantitative real-time PCR on degraded DNA samples: application to ancient DNA studies. In: Biotechniques (2005), Band 38(4), S. 569–75. PMID 15884675. PDF (Memento des Originals vom 4. März 2016 im Internet Archive) Info: Der Archivlink wurde automatisch eingesetzt und noch nicht geprüft. Bitte prüfe Original- und Archivlink gemäß Anleitung und entferne dann diesen Hinweis..
- D. J. Bacich, K. M. Sobek, J. L. Cummings, A. A. Atwood, D. S. O'Keefe: False negative results from using common PCR reagents. In: BMC Res Notes (2011), Band 4, S. 457. PMID 22032271; PMC 3219698 (freier Volltext).
- Wayne M. Barnes, Katherine R. Rowlyk: Magnesium precipitate hot start method for PCR. In: Molecular and Cellular Probes. Band 16, Nr. 3, Juni 2002, S. 167–171, doi:10.1006/mcpr.2002.0407, PMID 12219733.
- N. Paul, J. Shum, T. Le: Hot start PCR. In: Methods Mol Biol. (2010), Bd. 630, S. 301–18. PMID 20301005.
- M. F. Kramer, D. M. Coen: Enzymatic amplification of DNA by PCR: standard procedures and optimization. In: Curr Protoc Immunol. (2001), Kapitel 10, Einheit 10.20. PMID 18432685.
- H. FRAENKEL-CONRAT, B. A. BRANDON, H. S. OLCOTT: The reaction of formaldehyde with proteins; participation of indole groups; gramicidin. In: The Journal of biological chemistry. Band 168, Nummer 1, April 1947, ISSN 0021-9258, S. 99–118, PMID 20291066.
- H. FRAENKEL-CONRAT, H. S. OLCOTT: The reaction of formaldehyde with proteins; cross-linking between amino and primary amide or guanidyl groups. In: Journal of the American Chemical Society. Band 70, Nummer 8, August 1948, ISSN 0002-7863, S. 2673–2684, PMID 18876976.
- H. FRAENKEL-CONRAT, H. S. OLCOTT: Reaction of formaldehyde with proteins; cross-linking of amino groups with phenol, imidazole, or indole groups. In: The Journal of biological chemistry. Band 174, Nummer 3, Juli 1948, ISSN 0021-9258, S. 827–843, PMID 18871242.
- Q. Chou, M. Russell, D. E. Birch, J. Raymond, W. Bloch: Prevention of pre-PCR mis-priming and primer dimerization improves low-copy-number amplifications. In: Nucleic Acids Res. (1992), Band 20(7), S. 1717–23. PMID 1579465; PMC 312262 (freier Volltext).
- S. Tabor, C. C. Richardson: A single residue in DNA polymerases of the Escherichia coli DNA polymerase I family is critical for distinguishing between deoxy- and dideoxyribonucleotides. In: Proc Natl Acad Sci U S A. (1995), Band 92(14), S. 6339–43. PMID 7603992; PMC 41513 (freier Volltext).
- J. M. Prober, G. L. Trainor, R. J. Dam, F. W. Hobbs, C. W. Robertson, R. J. Zagursky, A. J. Cocuzza, M. A. Jensen, K. Baumeister: A system for rapid DNA sequencing with fluorescent chain-terminating dideoxynucleotides. In: Science (1987), Band 238(4825), S. 336–41. PMID 2443975.
- B. Fuchs, K. Zhang, M. G. Rock, M. E. Bolander, G. Sarkar: High temperature cDNA synthesis by AMV reverse transcriptase improves the specificity of PCR. In: Molecular biotechnology. Band 12, Nummer 3, Oktober 1999, S. 237–240, doi:10.1385/MB:12:3:237, PMID 10631680.
- A. M. Carothers, G. Urlaub, J. Mucha, D. Grunberger, L. A. Chasin: Point mutation analysis in a mammalian gene: rapid preparation of total RNA, PCR amplification of cDNA, and Taq sequencing by a novel method. In: Biotechniques (1989), Band 7(5), S. 494-6, 498-9. PMID 2483818.
- T. W. Myers, D. H. Gelfand: Reverse transcription and DNA amplification by a Thermus thermophilus DNA polymerase. In: Biochemistry (1991), Band 30(31), S. 7661-6. PMID 1714296.
- M. J. Moser, R. A. DiFrancesco, K. Gowda, A. J. Klingele, D. R. Sugar, S. Stocki, D. A. Mead, T. W. Schoenfeld: Thermostable DNA polymerase from a viral metagenome is a potent RT-PCR enzyme. In: PLoS One (2012), Band 7(6), S. e38371. doi:10.1371/journal.pone.0038371. PMID 22675552; PMC 3366922 (freier Volltext).