Didesoxyribonukleosid-Triphosphate
Didesoxyribonukleosid-Triphosphate (ddNTP) sind artifizielle DNA-Nukleotide, die bei der DNA-Sequenzierung nach Sanger Verwendung finden.[1] Die ddNTPs sind wie die Desoxyribonukleosid-Triphosphate (dNTPs) aufgebaut. Allerdings ist die Ribose (Zucker) an Position 2’ und 3’ desoxidiert. Dadurch fehlt am 3'-Kohlenstoff-Atom die Hydroxygruppe, an der bei der Polymerisation das nächste Nukleotid angehängt wird.
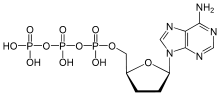
Baut die DNA-Polymerase während der Replikation an einer Stelle ein solches ddNTP ein, so kann kein weiteres Nukleotid angehängt werden und die Polymerisation bricht an dieser Stelle ab. Man kann deshalb vereinfacht von Stopp-Nukleotiden sprechen.
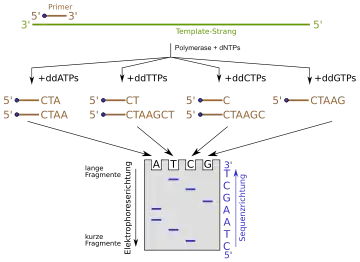
Bei der DNA-Sequenzierung nach Sanger werden nun die vier natürlichen dNTPs und eines der vier artifiziellen ddNTPs zu einem PCR-Ansatz mit der zu sequenzierenden DNA und einem entsprechenden Primer gegeben. Die Polymerase baut nun gelegentlich in statistischer Verteilung ein ddNTP ein, wodurch die Polymerisation abbricht. Es entsteht somit ein Gemisch unterschiedlich langer Stücke des zu sequenzierenden Abschnitts der DNA. Man führt dieses Experiment mit allen vier ddNTPs durch und trägt die vier Ansätze in einer Polyacrylamid-Gelelektrophorese nebeneinander auf. Aus dem Muster der entstehenden Banden kann man dann die Abfolge der Basen auf dem DNA-Abschnitt ablesen.
Einzelnachweise
- F. Sanger et al.: DNA sequencing with chain-terminating inhibitors. In: Proc. Natl. Acad. Sci. U.S.A., 1977, 74(12), S. 5463–5467; PMID 271968; PMC 431765 (freier Volltext).