Snodgrassella alvi
Snodgrassella alvi ist eine Art gramnegativer Bakterien innerhalb der Neisseriaceae und die einzige bekannte Art der Gattung Snodgrassella. Sie wurde 2012 durch Waldan K. Kwong und Nancy Moran isoliert und wissenschaftlich beschrieben, sie benannten die Bakterien nach dem amerikanischen Insektenforscher Robert Evans Snodgrass.
| Snodgrassella alvi | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
Snodgrassella alvi, elektronenmikroskopische Aufnahme | ||||||||||||
| Systematik | ||||||||||||
| ||||||||||||
| Wissenschaftlicher Name der Gattung | ||||||||||||
| Snodgrassella | ||||||||||||
| Kwong & Moran, 2012 | ||||||||||||
| Wissenschaftlicher Name der Art | ||||||||||||
| Snodgrassella alvi | ||||||||||||
| Kwong & Moran, 2012 |
Snodgrassella alvi lebt symbiotisch als Teil der Darmflora im Mitteldarm von Honigbienen (Apis mellifera) und einiger sozialer Hummelarten. In diesem Darmabschnitt stellen sie gemeinsam mit Gilliamella apicola die dominierenden Bakterien dar, wobei beide jeweils bis zu fast 40 % der dortigen Mikroflora repräsentieren. Im Darm interagieren Snodgrassella alvi und Gilliamella apicola bei der Nutzung der Stoffwechselressourcen, indem sie die Stoffwechselprodukte der jeweils anderen Art nutzen, und sie siedeln sich entsprechend in unterschiedlichen Bereichen der Darmwand an.
Einer im September 2018 veröffentlichten Studie zufolge wird Snodgrassella alvi durch die Verwendung des Pflanzenschutzmittels Glyphosat geschädigt, wodurch es zu Beeinträchtigungen der Darmmikrobiota kommt. Als Folge wurde eine Schwächung der Widerstandsfähigkeit der Bienen gegen schädliche Bakterien und darauf aufbauende Schwächungen der Tiere beobachtet.[1] Diese Wirkung wurde in der Folge in verschiedenen Medien international als eine mögliche Ursache des weltweit zu beobachtenden Bienensterbens diskutiert.
Merkmale
Erscheinungsbild
Snodgrassella alvi ist eine Bakterienart und damit ein einzelliger Organismus ohne Zellkern (Prokaryota). Die einzelnen Zellen sind kurz und stäbchenförmig mit einer Länge von etwa 1,0 µm und einem Durchmesser von 0,4 µm. Sie sind gramnegativ und besitzen entsprechend nur eine dünne Mureinhülle.[2]

Die Bakterien sind unbeweglich und bilden Kolonien mit anderen Bakterien in den Buchten der Darmwand des Bienendarms. Die Stämme können auf Blutagar, Casein-Soja-Pepton-Agar (CASO bzw. TSA), Heart Infusion Agar (HIA) und LB-Medium (LBA) wachsen und bilden nach 2 Tagen glatte, weiße und runde Kolonien mit einem Durchmesser von etwa 1 Millimeter oder weniger.[2]
Wachstum und Stoffwechsel
Die Art ist mikroaerophil, wächst also am besten bei geringem Sauerstoffgehalt. Eine fünfprozentige CO2-Atmosphäre bei einer Temperatur von 37 °C bietet optimale Wachstumsbedingungen, während die Bakterien in der Luft oder ohne Sauerstoff (Anaerobie) nur sehr schwaches oder gar kein Wachstum zeigen. Im TSA liegt der Wachstumsbereich bei etwa einem pH-Wert zwischen 6,0 und 6,5.[2] Der Katalase-Test und der Test auf Nitratreduktasen fällt positiv, der Oxidase-Test (Nachweis des Enzyms Cytochrom-c-Oxidase) fällt negativ aus. Tests der Stämme auf β-Glucosidase, β-Galactosidase, Indolproduktion, Proteolyse von Gelatine und Glukosefermentation sind negativ. Sie sind nicht hämolytisch und zeigen variable Reaktionen auf Urease und Argininindihydrolase.[2]
Das Bakterium kann Zitronen- und Äpfelsäure als wichtigste Kohlenstoffquelle nutzen.[2]
Chemotaxonomische Merkmale
Der gemeinsame Anteil von Cytosin und Guanin (GC-Gehalt) an der DNA beträgt 41 bis 43 mol%. Das wichtigste Isoprenoid-Chinon ist das Ubichinon-10. Die Hauptbestandteile der produzierten Fettsäuren sind Palmitinsäure (C 16:0), cis-Vaccensäure (C 18:1ω7 c/C 18:1ω6 c) und Laurinsäure (C 12:0),[2] worin sich Snodgrassella alvi von nahe verwandten Arten abgrenzen lässt.[3]
Lebensweise und Physiologie
Snodgrassella alvi lebt im Darm von Honigbienen und anderen Vertretern der corbiculaten Bienen. Im Genom vergleichbare Stämme wurden in allen daraufhin untersuchten Arten der Gattungen Apis (n=6) und Bombus (n=8) sowie in 9 von 13 untersuchten Arten der Stachellosen Bienen (Meliponini) gefunden,[7] in anderen Tierarten und außerhalb ihrer Wirte ist es nicht nachgewiesen. Sie ist eine Schlüsselart im Honigbienendarm und dominiert dieses Mikrobiom gemeinsam mit sieben weiteren, teilweise noch nicht final identifizierten Arten: Lactobacillus spp. Firm-4, Lactobacillus spp. Firm-5 (Phylum Firmicutes), Bifidobacterium spp. (phylum Actinobacteria), Gilliamella apicola, Frischella perrara, Bartonella apis und Alpha 2.1 (Phylum Proteobacteria). Diese Arten stellen gemeinsam 95 % der Darmbakterien. Snodgrassella alvi, Gilliamella apicola und Frischella perrara werden dabei als artspezifische Schlüsselarten betrachtet.[8]
Snodgrassella alvi und das Gammaproteobakterium Gilliamella apicola dominieren den Bereich des Ileums und besiedeln dort die Innenwand des Darms. Im vorderen Bereich des Darms existieren nur wenige Bakterien, während im kurzen Bereich des Pylorus Frischella perrara dominiert und fast ausschließlich dort vorkommt. Im Enddarm befinden sich vor allem Lactobacillus-Stämme und Bifidobacterium.[9]
Die Weitergabe des Mikrobioms und vor allem der Schlüsselarten erfolgt bei sozialen Insekten innerhalb des Stocks über die Weitergabe von Speichel und Nahrung. Bienenlarven und junge Arbeiterinnen sind in ihren ersten Lebenstagen nahezu frei von Darmbakterien und erwerben ihre normale mikrobielle Darmflora erst später oral durch soziale Interaktionen mit anderen Arbeitern und durch Übertragung zwischen den Individuen innerhalb eines Bienenstocks bei der gegenseitigen Nahrungsweitergabe (Trophallaxe) in den ersten Tagen außerhalb der Waben und zu Beginn ihres Lebens im Volk.[10][8][1] Obwohl auch die Larven von Arbeiterinnen gefüttert werden, ist ihr Darm, dessen Vorderteil und Hinterteil vor der Verpuppung nicht verbunden ist, fast frei von Bakterien. Dies wird vor allem auf eine starke Immunabwehr der Larven sowie eine bakteriozide Wirkung des Speichels der fütternden Bienen zurückgeführt.[10] Erst nach der Verpuppung und in Anwesenheit von Pflegebienen oder dem Kot dieser Bienen bilden die jungen Arbeiterinnen die für den Stock typische Darmflora aus. Bei der Exposition nur mit dem Stockmaterial wie Wabe, Honig und Bienenbrot oder nur dem Bienenspeichel und der Trophallaxe anderer Bienen bilden sie dagegen untypische Darmfloren aus.[8]
Innerhalb des Darms unterstützen die Bakterien die Verdauung von Honig und Pollen und haben darüber hinaus wahrscheinlich auch eine Funktion bei der Immunabwehr gegenüber Parasiten und pathogenen Bakterien.[11] Die Schutzwirkung der natürlichen Bakterienzusammensetzung konnte unter anderem nachgewiesen werden gegenüber Pathogenen wie die zu potenziell tödlichen Infektionen führenden Einzeller Crithidia bombi oder Nosema bombi, die verschiedene Hummelarten befallen.[12][13] Jedoch kann eine übermäßige Besiedelung des Bienendarms durch Snodgrassella alvi vermutlich die Darmflora stören und die Anfälligkeit gegenüber einer Infektion mit dem Trypanosomen Lotmaria passim erhöhen.[14]
Interaktion mit Gilliamella apicola
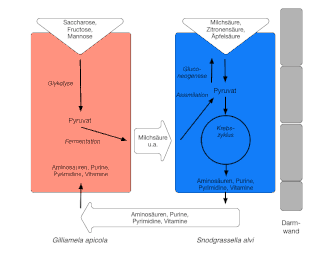
Innerhalb des Bienendarms besetzen die beiden dominanten Arten Gilliamella apicola und Snodgrassella alvi unterschiedliche Bereiche des Darms und unterschiedliche Stoffwechselnischen. Gemeinsam bilden sie einen Biofilm auf der inneren Darmwand, bei dem die Kolonien von Snodgrassella alvi direkt an der Darmwand sitzen und die Kolonien von Gilliamella apicola diese überdecken.[4][15][16]
Gilliamella apicola ist ein Bakterium, das Zucker abbaut und Carbonsäuren produziert (saccharolytischer Fermenter), während Snodgrassella alvi Carbonsäuren oxidiert. Im Darm bilden sie ein Netzwerk zur Aufteilung der Stoffwechselressourcen, bei dem beide Arten jeweils von den Eigenschaften der anderen profitieren. Gilliamella apicola wandelt entsprechend einfache Kohlenhydrate (Zucker) durch Glykolyse in Energie um und übergibt die übrig gebliebenen Moleküle an Snodgrassella alvi, die die notwendigen Gene für den Krebszyklus hat und diese nutzt, jedoch keine Glykolyse durchführen kann. Beide Arten besitzen zudem zahlreiche Gene und Proteine, die die Darmkolonisation sowie Interaktionen zwischen den Bakterienzellen ermöglichen.[4]
Variationen in diesen Genen könnten die Wirtstreue der Stämme erklären, die in früheren phylogenetischen Studien beobachtet wurde. Stämme von Snodgrassella alvi können ihren angestammten Bienenwirt kolonisieren, nicht aber Bienen einer anderen Gattung.[4] In Übereinstimmung mit der spezifischen, langfristigen Wirtsassoziation ergab eine vergleichende genomische Analyse große Unterschiede und einen geringen oder gar keinen Genfluss zwischen Darmsymbionten von Hummeln und Bienen. Innerhalb eines Wirtstyps (Apis oder Bombus) entdeckten Forscher jedoch Anzeichen eines horizontalen Gentransfers zwischen Gilliamella apicola und Snodgrassella alvi und demonstrierten damit die Bedeutung der breiteren Darmgemeinschaft bei der Ausrichtung der Evolution der einzelnen Mitglieder. Die Ergebnisse zeigten, dass die Wirtsspezifität wahrscheinlich von mehreren Faktoren beeinflusst wird, darunter direkte Interaktionen der Bakterien mit den Wirten, Mikroben-Mikroben-Interaktionen und die soziale Übertragung der Darmflora.[4]
Wirkung von Antibiotika und Glyphosat
Die Wirkungen von verschiedenen Substanzen auf die Darmflora der Bienen sind nur punktuell erforscht. Dabei liegen Untersuchungen zur Wirkung von Antibiotika und von Glyphosat auf die Bakterienflora und vor allem auf die Besiedlung durch Gilliamella apicola und Snodgrassella alvi vor.
Kasie Raymann und Kollegen untersuchten 2017 die Wirkung des Antibiotikums Tetracyclin, das in Bienenstöcken teilweise zur Prävention von bakteriellen Infektionen der Bienenlarven eingesetzt wird, auf die Darmflora der Bienen. Sie stellten fest, dass die Behandlung zu einer Reduzierung der Zellen von Snodgrassella alvi führt, während Gilliamella apicola in der Anzahl der Zellen kaum reduziert wurde.[17] In einer zweiten Untersuchung konnten sie jedoch feststellen, dass es zu einer Verschiebung der Genotypen bei Gilliamella apicola zugunsten antibiotikaresistenter Zellen und damit zu einer Reduzierung der genetischen Diversität kommt, während die genetische Diversität bei Snodgrassella alvi nicht beeinträchtigt wurde.[16] Nach ihren Untersuchungen führte die Antibiotika-Exposition zudem zu einer verminderten Überlebensrate der damit behandelten Bienen, sowohl im Bienenstock als auch in Laborexperimenten, bei denen die Bienen opportunistischen Bakterienpathogenen ausgesetzt waren.[17]
Generell wird angenommen, dass das Herbizid Glyphosat für Tiere, auch Bienen und andere Insekten, unschädlich ist, da es in seiner Wirkung auf das Enzym 5-Enolpyruvylshikimat-3-Phosphat-Synthase (EPSPS) abzielt, das nur in Pflanzen und Mikroorganismen vorkommt. Die Wirkungen von Glyphosat auf die Mikrobiome der Tiere und damit auch die Mikroorganismen, die den Bienendarm besiedeln und dort die natürliche Darmflora darstellen, wurden bei dieser Annahme allerdings bislang nicht betrachtet. Das Gen, das EPSPS kodiert, ist in fast allen sequenzierten Genomen von Bienendarm-Bakterien vorhanden, was darauf hindeutet, dass diese potenziell anfällig für Glyphosat sind. Einer im September 2018 veröffentlichten Untersuchung zufolge beeinträchtigt Glyphosat die Darmmikrobiota von jungen Honigbienen, indem der Shikimisäureweg vor allem bei Snodgrassella alvi gehemmt wird. Die Exposition der Bienen mit Glyphosat verändert somit zumindest experimentell die Darmflora junger Bienenarbeiterinnen und erhöht damit auch die Anfälligkeit der Bienen für Infektionen. Als Folge wurde eine Schwächung der Widerstandsfähigkeit gegen opportunistische Krankheitserreger und vor allem die schädliche Bakterienart Serratia marcescens beobachtet, die mit einer erhöhten Sterblichkeit der Bienen einhergeht.[1] Diese Wirkung wurde in der Presse als eine mögliche Ursache des international zu beobachtenden Bienensterbens diskutiert.u. a.[18][19][20][21] Es wurden allerdings auch Resistenzen mancher Stämme von Snodgrassella alvi gegen Glyphosat beschrieben.[1]
Nutzung für Milbenabwehr und Infektionsschutz

Im Januar 2020 wurden die Ergebnisse einer Forschungsarbeit veröffentlicht, bei der gentechnisch veränderte Snodgrassella alvi genutzt wurden, um die Immunabwehr der Bienen gegen den Befall durch die Varroamilbe (Varroa destructor) und durch diese induzierte Virusinfektionen zu stärken.[22] Das Team veränderte die Bakterien so, dass durch ein eingebautes Plasmid markierte doppelsträngige RNA (dsRNA) gebildet wird. Das dsRNA-Modul kann gezielt genutzt werden, um mit spezifischen Bienengenen sowie mit wichtigen Virus- und Milbengenen zu interferieren. Im Labor konnte die Genexpression der ausgewählten Bereich für mindestens 15 Tage blockiert werden, wenn sich die modifizierten Bakterien im Bienendarm etablierten und die dsRNA kontinuierlich exprimierten. Als Effekt wurde sowohl das Überleben der Varroamilben auf den Bienen reduziert wie auch die Übertragung und die Infektion mit dem Flügeldeformationsvirus gehemmt.[22]
Taxonomie
Externe Systematik
Snodgrassella alvi wurde von Waldan K. Kwong und Nancy A. Moran gemeinsam mit Gilliamella apicola aus dem Darm der Europäischen Honigbiene isoliert und 2012 wissenschaftlich beschrieben. Bereits vorher wurden beide Arten von Vincent G. Martinson und Kollegen ebenfalls in der Arbeitsgruppe von Nancy A. Moran, identifiziert und provisorisch als Candidatus Snodgrassella alvi und Gilliamella apicola benannt.[10] Die Benennung der Gattung Snodgrassella erfolgte nach dem amerikanischen Entomologen Robert Evans Snodgrass, der damit als Pionier bei der Erforschung der Insektenphysiologie im frühen 20. Jahrhundert geehrt wurde, während sich das Artepitheton „alvi“ auf die Lakunen des Bienendarms bezieht.[2] Der Typus-Stamm ist wkB2 T (= NCIMB 14803 T = ATCC BAA-2449 T = NRRL B-59751 T),[23] isoliert aus dem Darm der Westlichen Honigbiene (A. mellifera) in Connecticut, Vereinigte Staaten.
| |||||||||||||||||||||||||||||||||||||||||||
Phylogenetische Position von Snodgrassella alvi nach Kwong 2017[24]
Die Bakterien werden als Betaproteobacteria innerhalb der Neisseriaceae eingeordnet und sind verwandt mit ähnlichen Bakterien aus dem Darm von Termiten oder anderen Insekten.[2][4] Kwong erstellte 2017 ein Kladogramm der Neisseriaceae auf der Basis veröffentlichter Genomdaten, bei der er Snodgrassella alvi als Schwesterart der im Darm von Termiten vorkommenden Art Stenoxybacter acetivorans einordnete und gemeinsam mit der Gattung Vitreoscilla an die Basis eines Taxons aus den Gattungen Neisseria (polyphyletisch), Eikenella, Conchiformibius, Alysiella, Simonsiella, Klingella und Bergeriella platzierte.[24][25] Während die basaleren Taxa vor allem in offenen Lebensräumen wie im Boden oder in Gewässern vorkommen, sind die abgeleiteten Formen einschließlich Stenoxybacter und Snodgrassella in der Regel an andere Organismen, vor allem Tiere gebunden.[24]
Eine Arbeitsgruppe um den chinesischen Forscher Yong Li gruppierte Vitreoscilla, Stenoxybacter und Snodgrassella in ein Taxon, das zusätzlich noch die in der Arbeit neu beschriebene Art Populibacter corticis aus der Borke eines Baumkrebses der Bastard-Schwarz-Pappel (Populus ×euramericana) als Schwesterart von Snodgrassella alvi enthält.[3]
Stämme und Wirtsspezifität
Innerhalb der Art werden die Bakterien nach Stämmen klassifiziert, die verschiedenen Wirten entnommen wurden. Der für die Erstbeschreibung genutzte Stamm wkB2 T wurde entsprechend aus dem Darm der Westlichen Honigbiene (Apis mellifera) in Connecticut, Vereinigte Staaten, isoliert. Als spezialisierter Darmsymbiont entwickelte sich Snodgrassella alvi seit Millionen von Jahren mit Honigbienen und Hummeln (Gattung Bombus), in denen die Bakterien zu finden sind.
Bei Untersuchungen konnte festgestellt werden, dass sich die verschiedenen Stämme von Snodgrassella alvi aus Honigbienen weltweit in den Gensequenzen der 16S rRNA (in der V4-Region) kaum unterscheiden und nahezu identisch sind. Dagegen gibt es Unterschiede im Single-copy-Gen minD (eine ATPase, welche die Zellteilung hemmt) vor allem in Stämmen von Snodgrassella alvi aus der Honigbiene, während Hummeln nur einen Stamm von Snodgrassella alvi aufweisen.[26] Beim Vergleich mit Snodgrassella-alvi-Stämme aus Honigbienen mit denen aus verschiedenen Hummelarten konnte zum einen festgestellt werden, dass die genetische Variabilität innerhalb der einzelnen Honigbienen eines Stocks deutlich höher ist als bei den Hummeln eines Volks, wobei die Diversität auf die Gründung einer Bienenkolonie durch Schwärme von Arbeiterinnen im Gegensatz zu einer einzelnen Hummel zurückgeführt wird.[26] Dadurch wird der Darm der einzelnen Honigbienen in der Regel (zu 86 %) durch mehrere Stämme von Snodgrassella alvi besiedelt, während der der Hummeln in der Regel (zu 72 %) nur einen Stamm enthält. Zudem wurde eine Wirtsspezifität der Bakterien der Honigbiene festgestellt, bei der es keine Stämme gibt, die sowohl bei Honigbienen wie auch bei Hummelarten gefunden werden können. Innerhalb der Hummeln gibt es einige Snodgrassella-alvi-Stämme, die art- oder untergattungsspezifisch vorkommen, während andere bei mehreren Arten verschiedener Untergattungen vorkommen.[26] In weiteren Analysen zeigte sich, dass es bei den proteincodierenden Genen der Honigbienen-Stämme von Snodgrassella alvi im Gegensatz zu denen der rRNA deutlich größere Unterschiede gibt. Erklärt wird dies dadurch, dass die Variabilität der 16S-rRNA-Loci durch häufige Rekombination innerhalb der Populationen eingeschränkt worden sei, während andere Regionen des Genoms sich in Anpassung an sich verändernde ökologische Bedingungen im Darm kontinuierlich weiterentwickeln und diversifizieren.[27]
Belege
- Erick V. S. Motta, Kasie Raymann, Nancy A. Moran: Glyphosate perturbs the gut microbiota of honey bees. In: Proceedings of the National Academy of Sciences 115 (41), 2018; S. 10305–10310. doi:10.1073/pnas.1803880115.
- Waldan K. Kwong, Nancy A. Moran: Cultivation and characterization of the gut symbionts of honey bees and bumble bees: description of Snodgrassella alvi gen. nov., sp. nov., a member of the family Neisseriaceae of the Betaproteobacteria, and Gilliamella apicola gen. nov., sp. nov., a member of Orbaceae fam. nov., Orbales ord. nov., a sister taxon to the order 'Enterobacteriales' of the Gammaproteobacteria. International Journal of Systematic and Evolutionary Microbiology, Band 63, 6, 5. Oktober 2012, doi:10.1099/IJS.0.044875-0.
- Yong Li, Han Xue, Sheng-qi Sang, Cai-li Lin, Xi-zhuo Wang: Phylogenetic analysis of family Neisseriaceae based on genome sequences and description of Populibacter corticis gen. nov., sp. nov., a member of the family Neisseriaceae, isolated from symptomatic bark of Populus × euramericana canker. Plos One, 13. April 2017. doi:10.1371/journal.pone.0174506.
- Waldan K. Kwong, Philipp Engel, Hauke Koch, Nancy A. Moran: Genomics and host specialization of honey bee and bumble bee gut symbionts. Proceedings of the National Academy of Sciences 111 (31), 5. August 2014; S. 11509–11514. doi:10.1073/pnas.1405838111.
- 597812552 - Nucleotide Result. In: ncbi.nlm.nih.gov. Abgerufen am 20. Mai 2019 (englisch).
- J. E. Powell, S. P. Leonard, W. K. Kwong, P. Engel, N. A. Moran: Genome-wide screen identifies host colonization determinants in a bacterial gut symbiont. In: Proceedings of the National Academy of Sciences. Band 113, Nummer 48, 11 2016, S. 13887–13892, doi:10.1073/pnas.1610856113, PMID 27849596, PMC 5137728 (freier Volltext).
- Waldan K. Kwong, Luis A. Medina, Hauke Koch, Kong-Wah Sing, Eunice Jia Yu Soh, John S. Ascher, Rodolfo Jaffé, Nancy A. Moran: Dynamic microbiome evolution in social bees. Science Advances 3 (3), 2017: e1600513. doi:10.1126/sciadv.1600513
- Elijah Powell, Vincent G. Martinson, Katherine Urban-Mead, Nancy A. Moran: Routes of Acquisition of the Gut Microbiota of the Honey Bee Apis mellifera. In: Applied and Environmental Microbiology 80, 2014; S. 7378–7387. doi:10.1128/AEM.01861-14.
- Waldan K. Kwong, Nancy A. Moran: Gut microbial communities of social bees. Nature Reviews Microbiology 14, 2016; S. 374–384. doi:10.1038/nrmicro.2016.43.
- Vincent G. Martinson, Jamie Moy, Nancy A. Moran: Establishment of Characteristic Gut Bacteria during Development of the Honeybee Worker. Applied and Environmental Microbiology 78, 2012; S. 2830–2840. doi:10.1128/AEM.07810-11.
- P. Engel, Nancy A. Moran: The gut microbiota of insects – diversity in structure and function. In: FEMS Microbiology Reviews, 37(5), 699–735, 1. September 2013. doi:10.1111/1574-6976.12025.
- Hauke Koch, Paul Schmid-Hempel: Socially transmitted gut microbiota protect bumble bees against an intestinal parasite. In: Proceedings of the National Academy of Sciences 108 (48), 2011, S. 19288–19292. doi:10.1073/pnas.1110474108.
- Daniel P. Cariveau, J. Elijah Powell, Hauke Koch, Rachael Winfree, Nancy A. Moran: Variation in gut microbial communities and its association with pathogen infection in wild bumble bees (Bombus). The ISME Journal 8, 2014; S. 2369–2379. doi:10.1038/ismej.2014.68.
- R. S. Schwarz, N. A. Moran, J. D. Evans: Early gut colonizers shape parasite susceptibility and microbiota composition in honey bee workers. In: Proceedings of the National Academy of Sciences. Band 113, Nummer 33, 08 2016, S. 9345–9350, doi:10.1073/pnas.1606631113, PMID 27482088, PMC 4995961 (freier Volltext).
- Lucie Kešnerová, Ruben A. T. Mars, Kirsten M. Ellegaard, Michaël Troilo, Uwe Sauer, Philipp Engel: Disentangling metabolic functions of bacteria in the honey bee gut. Plos One, 12. Dezember 2017. doi:10.1371/journal.pbio.2003467
- Kasie Raymann, Louis‐Marie Bobay, Nancy A. Moran: Antibiotics reduce genetic diversity of core species in the honeybee gut microbiome. Molecular Ecology 27 (8), 22. November 2017. doi:10.1111/mec.14434.
- Kasie Raymann, Zack Shaffer, Nancy A. Moran: Antibiotic exposure perturbs the gut microbiota and elevates mortality in honeybees. Plos One, 14. März 2017. doi:10.1371/journal.pbio.2001861.
- Frank Patalong: Glyphosat soll Ursache für Bienensterben sein. Spiegel online, 24. September 2018; abgerufen am 31. März 2019.
- Ashley May: Honey bees are dying. A popular weed killer might be to blame, study says. USA today, 25. September 2018; abgerufen am 31. März 2019.
- Damian Carrington: Monsanto's global weedkiller harms honeybees, research finds. The Guardian, 24. September 2018; abgerufen am 31. März 2019.
- Tina Baier: Macht Glyphosat die Bienen krank? Süddeutsche Zeitung, 25. September 2018; abgerufen am 31. März 2019.
- Sean P. Leonard, J. Elijah Powell, Jiri Perutka, Peng Geng, Luke C. Heckmann, Richard D. Horak, Bryan W. Davies, Andrew D. Ellington, Jeffrey E. Barrick, Nancy A. Moran: Engineered symbionts activate honey bee immunity and limit pathogens. Science 367 (6477), 31. Januar 2020; S. 573–576. doi:10.1126/science.aax9039.
- W. K. Kwong, N. A. Moran: Cultivation and characterization of the gut symbionts of honey bees and bumble bees: description of Snodgrassella alvi gen. nov., sp. nov., a member of the family Neisseriaceae of the Betaproteobacteria, and Gilliamella apicola gen. nov., sp. nov., a member of Orbaceae fam. nov., Orbales ord. nov., a sister taxon to the order 'Enterobacteriales' of the Gammaproteobacteria. In: International journal of systematic and evolutionary microbiology. Band 63, Pt 6Juni 2013, S. 2008–2018, doi:10.1099/ijs.0.044875-0, PMID 23041637.
- Waldan K. Kwong: Whole genome phylogeny of Neisseriaceae species. Blogbeitrag im Blog von Waldan K. Kwong, 13. Mai 2017; abgerufen am 1. März 2019.
- Waldantgu K. Kwong, Hao Zheng, Nancy A. Moran: Convergent evolution of a modified, acetate-driven TCA cycle in bacteria. Nature Microbiology 2, 2017; Artikel 17067. doi:10.1038/nmicrobiol.2017.67.
- Elijah Powell, Nalin Ratnayeke Nancy A. Moran: Strain diversity and host specificity in a specialized gut symbiont of honeybees and bumblebees. Molecular Ecology 25 (18), September 2016; S. 4461–4471. doi:10.1111/mec.13787.
- Philipp Engel, Ramunas Stepanauskas, Nancy A. Moran: Hidden Diversity in Honey Bee Gut Symbionts Detected by Single-Cell Genomics. PLOS Genetics, 11. September 2014. doi:10.1371/journal.pgen.1004596.
Literatur
- Waldan K. Kwong, Nancy A. Moran: Cultivation and characterization of the gut symbionts of honey bees and bumble bees: description of Snodgrassella alvi gen. nov., sp. nov., a member of the family Neisseriaceae of the Betaproteobacteria, and Gilliamella apicola gen. nov., sp. nov., a member of Orbaceae fam. nov., Orbales ord. nov., a sister taxon to the order 'Enterobacteriales' of the Gammaproteobacteria. International Journal of Systematic and Evolutionary Microbiology, Band 63, 6, 5. Oktober 2012, doi:10.1099/IJS.0.044875-0.
Weblinks
- Genus Snodgrassella in der List of Prokaryotic names with Standing in Nomenclature (LPSN)
- Snodgrassella alvi, Beschreibung des Typusstamms in der DSMZ-Datenbank