D-Loop
Eine D-Loop (synonym Verdrängungsschleife[1] von engl. displacement loop) ist eine Form der Sekundärstrukturen von DNA. Sie kommt vorübergehend bei der DNA-Reparatur, bei Telomeren und bei der DNA-Replikation von mtDNA vor.
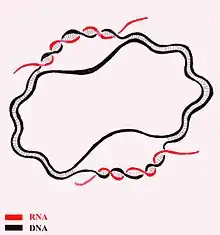
Eigenschaften
Die D-Loop besteht aus einer doppelsträngigen DNA und einer einzelsträngigen DNA mit komplementärer Sequenz zu einem der beiden DNA-Stränge. Durch die Einlagerung des dritten Stranges wird einer der beiden DNA-Stränge abschnittsweise verdrängt.
DNA-Reparatur
In einer Form der DNA-Reparatur, der Reparatur durch homologe Rekombination bildet die D-Loop einen Übergangszustand. In Bakterien wird eine D-Loop durch RecA erzeugt, die anschließend durch eine Resolvase einzelsträngig gespalten wird.[2][3] In Säugetieren sind die Proteine RPA (ein Einzelstrang-bindendes Protein) sowie Rad52, das wiederum Rad51 bindet, an der Bildung der D-Loop beteiligt.[4]
Telomere
Bei der Replikation der Telomere wird an ihrem Ende eine lassoartige Struktur ausgebildet, die als T-loop bezeichnet wird und eine D-Loop beinhaltet.[5] Die T-Loop schützt die Enden der Telomere vor einem Abbau durch Exonukleasen.[6] Bei der T-Loop stammt der dritte Strang von einem 3'-Überhang am Ende der doppelsträngigen DNA (dsDNA), die sich stromaufwärts zwischen den Doppelstrang drängt und somit die Lassoform ausbildet.[7] An die Telomere bindet Shelterin.[8]
mtDNA
In mitochondrialer DNA (mtDNA) liegt die D-Loop von etwa 660 bp (Basenpaaren) stromabwärts direkt nach dem L-Strang-Promotor (mit circa 440 bp).[9][10] Die D-Loop liegt in der mtDNA-Kontrollregion. L-Strang-Promotor und D-Loop bilden einem Bereich des Replikationsursprungs der mtDNA, der als Hauptkontrollregion bezeichnet wird und etwa 1100 Basenpaare lang ist.[11][9] Die D-Loop wird hier dauerhaft aus dsDNA mit einem DNA-verlängerten RNA-Primer (7S-DNA) gebildet.[12]
Anwendungen
Innerhalb der Hauptkontrollregion der mtDNA liegen zwei hypervariable Regionen, die im Zuge einer Erstellung eines phylogenetischen Baums durch DNA-Sequenzierung der D-Loop ermittelt werden können.[9][13] In den hypervariablen Regionen ist die Mutationsrate etwa 200- bis 400-fach höher als bei DNA aus dem Zellkern.[14] Neben der Sequenzierung von Y-Chromosomen ist die Sequenzierung der D-Loop eine der häufigst verwendeten Methoden zur Bestimmung von Verwandtschaftsgraden beim Menschen.[15]
Manche Mutationen in der hypervariablen Region sind mit verschiedenen Tumoren assoziiert, darunter Gebärmutterhalskrebs, Brustkrebs, Magenkrebs, Darmkrebs, Leberkrebs, Lungenkrebs und Nierenkrebs.[14] Die Mutationen T16126C, T16224C und T16311C in der ersten hypervariablen Region sind negative Prognosefaktoren für akute lymphatische Leukämie bei Kindern.[14] Die Mutation T16189C ist mit Koronarerkrankung bei manchen Mitteleuropäern assoziiert.[14]
Geschichte
Die D-Loop wurde erstmals im Jahr 1971 von H. Kasamatsu und Kollegen beschrieben.[16]
Einzelnachweise
- Katharina Munk: Taschenlehrbuch Biologie: Genetik. Georg Thieme Verlag, 2010, ISBN 978-3-131-68621-3 (eingeschränkte Vorschau in der Google-Buchsuche).
- D. Kidane, S. Ayora, J. B. Sweasy, P. L. Graumann, J. C. Alonso: The cell pole: the site of cross talk between the DNA uptake and genetic recombination machinery. In: Critical reviews in biochemistry and molecular biology. Band 47, Nummer 6, 2012 Nov-Dec, S. 531–555, doi:10.3109/10409238.2012.729562, PMID 23046409, PMC 3490228 (freier Volltext).
- T. Shibata, T. Nishinaka, T. Mikawa, H. Aihara, H. Kurumizaka, S. Yokoyama, Y. Ito: Homologous genetic recombination as an intrinsic dynamic property of a DNA structure induced by RecA/Rad51-family proteins: a possible advantage of DNA over RNA as genomic material. In: Proceedings of the National Academy of Sciences. Band 98, Nummer 15, Juli 2001, S. 8425–8432, doi:10.1073/pnas.111005198, PMID 11459985, PMC 37453 (freier Volltext).
- K. A. Bernstein, S. Gangloff, R. Rothstein: The RecQ DNA helicases in DNA repair. In: Annual review of genetics. Band 44, 2010, S. 393–417, doi:10.1146/annurev-genet-102209-163602, PMID 21047263, PMC 4038414 (freier Volltext).
- J. D. Griffith, L. Comeau, S. Rosenfield, R. M. Stansel, A. Bianchi, H. Moss, T. de Lange: Mammalian telomeres end in a large duplex loop. In: Cell. Band 97, Nummer 4, Mai 1999, S. 503–514, PMID 10338214.
- C. W. Greider: Telomeres do D-loop-T-loop. In: Cell. Band 97, Nummer 4, Mai 1999, S. 419–422, PMID 10338204.
- Lalit M. Srivastava: Plant Growth and Development. Academic Press, 2002, ISBN 978-0-080-51403-1, S. 87.
- Thomas D. Pollard: Cell Biology E-Book. Elsevier Health Sciences, 2016, ISBN 978-0-323-40002-2, S. 120.
- Rolf Knippers: Molekulare Genetik. Georg Thieme Verlag, 2006, ISBN 978-3-134-77009-4, S. 460 (eingeschränkte Vorschau in der Google-Buchsuche).
- D. D. Chang, D. A. Clayton: Priming of human mitochondrial DNA replication occurs at the light-strand promoter. In: Proceedings of the National Academy of Sciences. Band 82, Nummer 2, Januar 1985, S. 351–355, PMID 2982153, PMC 397036 (freier Volltext).
- Roberto Scatena: Advances in Mitochondrial Medicine. Springer Science & Business Media, 2012, ISBN 978-9-400-72869-1, S. 42 (eingeschränkte Vorschau in der Google-Buchsuche).
- T. J. Nicholls, M. Minczuk: In D-loop: 40 years of mitochondrial 7S DNA. In: Experimental Gerontology. Band 56, August 2014, S. 175–181, doi:10.1016/j.exger.2014.03.027, PMID 24709344.
- A. Larizza, G. Pesole, A. Reyes, E. Sbisà, C. Saccone: Lineage specificity of the evolutionary dynamics of the mtDNA D-loop region in rodents. In: Journal of molecular evolution. Band 54, Nummer 2, Februar 2002, S. 145–155, doi:10.1007/s00239-001-0063-4, PMID 11821908.
- H. Li, D. Liu, J. Lu, Y. Bai: Physiology and pathophysiology of mitochondrial DNA. In: Advances in Experimental Medicine and Biology. Band 942, 2012, S. 39–51, doi:10.1007/978-94-007-2869-1_2, PMID 22399417, PMC 4706180 (freier Volltext).
- S. Kundu, S. K. Ghosh: Trend of different molecular markers in the last decades for studying human migrations. In: Gene. Band 556, Nummer 2, Februar 2015, S. 81–90, doi:10.1016/j.gene.2014.12.023, PMID 25510397.
- H. Kasamatsu, D. L. Robberson, J. Vinograd: A novel closed-circular mitochondrial DNA with properties of a replicating intermediate. In: Proceedings of the National Academy of Sciences. Band 68, Nummer 9, September 1971, S. 2252–2257, PMID 5289384, PMC 389395 (freier Volltext).