DNA-Polymerasen
DNA-Polymerasen sind Enzyme, die als Polymerase die Synthese von DNA aus Desoxyribonukleotiden katalysieren. DNA-Polymerasen spielen eine Schlüsselrolle bei der DNA-Replikation.
| DNA-Polymerase | ||
|---|---|---|
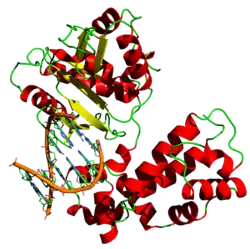
| ||
| Bänderdarstellung der DNA-bindenden Domäne der humanen DNA-Polymerase β | ||
| Bezeichner | ||
| Externe IDs | ||
| Enzymklassifikation | ||
| EC, Kategorie | 2.7.7.7, Transferase | |
| Substrat | Desoxyribonucleosidtriphosphat + DNAn | |
| Produkte | Diphosphat + DNAn+1 | |
Biochemische Aspekte
Polymerase-Aktivität
Die Polymerase ermöglicht die chemische Verknüpfung von einzelnen Molekülen (Monomere) zu einer Kette (Polymer). Im Falle der DNA-Polymerase ist das gebildete Polymer die Desoxyribonukleinsäure (DNA), als Monomere dienen Desoxyribonukleotide, genauer Desoxy-Nukleosidtriphosphate (dNTPs). Die DNA-abhängige DNA-Polymerase nutzt stets einen bereits bestehenden DNA-Einzelstrang als Matrize (Vorlage) für die Synthese eines neuen, komplementären Stranges, dessen Nukleotid-Abfolge somit von der Matrize bestimmt wird. Dieser Erhalt der DNA-Sequenz ist entscheidend für die Fähigkeit der DNA-Polymerase, die in der DNA codierte Erbinformation zu kopieren. Das korrekte Kopieren der Matrize gelingt durch komplementäre Basenpaarung der eingebauten Nukleotidbasen mit den Basen der DNA-Matrize, vermittelt durch Wasserstoffbrücken. Die Synthese des neuen DNA-Stranges erfolgt vom 5'- zum 3'-Ende. Chemisch betrachtet findet dabei ein nukleophiler Angriff der endständigen 3'-Hydroxygruppe des DNA-Stranges auf das α-Phosphat des dNTPs statt, wobei Pyrophosphat freigesetzt wird. Dieser Schritt wird von der Polymerase katalysiert.
Im Gegensatz zu RNA-Polymerasen kann die Synthese des komplementären DNA-Stranges bei DNA-Polymerasen nur erfolgen, wenn der Polymerase ein freies 3'-Hydroxyende zur Verfügung steht. An dieses wird dann das erste Nukleotid angehängt. Bei der Polymerase-Kettenreaktion (PCR) nutzt man hierzu einen ca. 15–20 Nukleotide langen DNA-Einzelstrang (Primer), der als Startpunkt der Reaktion dient. DNA-Polymerasen benötigen in der Regel Magnesium-Ionen als Kofaktor.
Die Katalyse der Bildung der Diesterbindung erfolgt funktionell analog zu der entsprechenden Reaktion der RNA-Polymerasen. Das letzte Nukleotid des bereits synthetisierten Abschnittes und das anzufügende Nukleotid sind an jeweils eines von zwei Magnesium-Ionen im katalytischen Zentrum der Polymerasendomäne koordiniert. Die erste Phosphatgruppe des anzufügenden Nukleotids ist an beide Magnesium-Ionen koordiniert. Die räumliche Lage ermöglicht einen Angriff der Hydroxygruppe des vorangegangenen Nukleotids auf die Phosphatgruppe des anzufügenden. Dabei wird ein Pyrophosphatrest abgespalten.
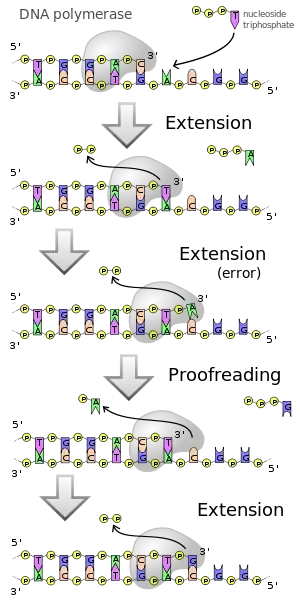
Exonuklease-Aktivität
Viele Polymerasen haben zusätzlich noch weitere Enzym-Funktionen. In Anwesenheit geringer Konzentrationen an dNTPs überwiegt die 3'→5'-Exonuklease-Aktivität zum Entfernen von Nukleotiden. Einige Polymerasen besitzen auch eine 5'→3'-Exonuklease-Aktivität. Um zu gewährleisten, dass es zu keinen Fehlern beim Ablesen der DNA-Matrize kommt, verfügen sie über diese Korrekturlese-Funktion (engl. proof reading), d. h., sie sind in der Lage, den Einbau eines unpassenden Nukleotids zu erkennen und dieses anschließend durch die Exonuclease-Aktivität wieder aus der DNA zu entfernen. Diese ermöglicht den Abbau eines bestehenden DNA- oder RNA-Stranges, der bereits mit dem Matrizenstrang gepaart ist, während ein neuer Strang gebildet wird. Es resultiert ein Austausch des alten Stranges gegen einen neuen Strang. Diese Exonuklease-Aktivität wird bei der Methode der nick translation ausgenutzt.
Verschiedene DNA-Polymerasen
In Bakterien, wie etwa Escherichia coli gibt es drei verschiedene DNA-abhängige DNA-Polymerasen. Eine davon, die DNA-Polymerase I (Pol I) wurde im Jahr 1955 von Arthur Kornberg isoliert und war die erste Polymerase überhaupt, die entdeckt wurde. Diese ist aber nicht die für die Replikation wichtigste Polymerase in E. coli, da sie nur etwa 20 Syntheseschritte katalysiert (d. h., sie besitzt nur eine niedrige Prozessivität). Sie ist jedoch durch ihre 5'→3'-Exonuklease-Aktivität bei der Replikation für den Primerabbau zuständig. DNA-Polymerase II und DNA-Polymerase III, die anderen beiden DNA-Polymerasen in E. coli, wurden erst 15 Jahre nach der Entdeckung der DNA-Polymerase I isoliert, nachdem sich E. coli-Mutanten mit Defekt im Polymerase I Gen dennoch als replikationskompetent erwiesen. Diese Mutanten waren allerdings besonders anfällig gegenüber UV-Strahlung und alkylierenden Substanzen, weshalb man annimmt, dass die DNA-Polymerase I vorwiegend Reparaturaufgaben übernimmt. Die Polymerase III, die in E. coli die eigentliche Replikation durchführt, ist aus insgesamt sieben Untereinheiten aufgebaut und kommt pro Bakterienzelle nur in sehr wenigen Kopien vor.
Eukaryotische DNA-Polymerasen werden in folgende Familien klassifiziert:
- Familie A: DNA-Polymerasen γ, θ und ν
- Familie B: DNA-Polymerasen α, δ, ε und ζ
- Familie X: DNA-Polymerasen β, λ, σ und μ
- Familie Y: DNA-Polymerasen η, ι und κ
Die Polymerase γ tritt nur in Mitochondrien auf.
In Säugern kommen nur fünf DNA-Polymerasen vor: α, β, γ, δ und ε. Es wird angenommen, dass es sich bei den für die Replikation entscheidenden um die Polymerasen δ und ε handelt, die sich durch hohe Prozessivität und Korrekturlesefunktion (proof reading) auszeichnen. Die Polymerasen α und β zeigen dagegen nur geringe Prozessivität und keine Proofreadingfunktion.
Des Weiteren existieren RNA-abhängige DNA-Polymerasen, die die RNA als Matrize nutzen und dNTPs daran anhängen. Diese nennen sich Reverse Transkriptase, wozu auch die Telomerase gehört. Als unabhängige DNA-Polymerase ist als einziges die terminale Desoxyribonucleotidyltransferase bekannt.
In Archaebakterien existieren temperaturstabile DNA-Polymerasen, die ebenfalls zur PCR genutzt werden.
Keine 3'→5'-Polymerasen
In 3'→5' prozessierende DNA-Polymerase-Komplexe sind nicht bekannt. Eine Verlängerung des DNA-Stranges in diese Richtung würde die Hydrolyse des zuvor angefügten Nukleosidtriphosphats erfordern. Dies ist zwar prinzipiell möglich, führt aber zu einem Problem im Hinblick auf eine zusätzliche Korrekturfunktion (proofreading). Denn nach der Reaktion einer Exonuklease bliebe am Strang keine Triphosphat-Gruppe am Ende, sondern nur eine einfache Phosphatgruppe zurück, womit die weitere Verlängerung des Stranges unterbunden wäre. Der folgende hypothetische Reaktionsmechanismus kann dies verdeutlichen:[1]

Biologische Bedeutung
DNA-Polymerasen sind von zentraler Bedeutung für die DNA-Replikation. Sie ermöglichen das getreue Kopieren der Erbinformation in Form von DNA, somit einen entscheidenden Schritt bei der Vermehrung und Fortpflanzung von Lebewesen. DNA-Polymerasen spielen zudem bei Vorgängen, die mit der Reparatur von DNA einhergehen, eine wichtige Rolle.
Biotechnologische Bedeutung
Im Labor werden DNA-Polymerasen oft für die Polymerase-Kettenreaktion und verwandten Methoden (z. B. RT-PCR, qPCR), bei der Nick translation, beim Random priming und bei der DNA-Sequenzierung eingesetzt. Dabei kommt eine Vielzahl von verschiedenen, teils per Protein-Engineering veränderten thermostabilen DNA-Polymerasen zum Einsatz (z. B. Taq-Polymerase). Neben einer hohen Temperaturstabilität bringen thermostabile DNA-Polymerasen archaeischen Ursprung wie die Pfu-Polymerase eine Korrekturlese-Funktion (proof-reading) mit, da es bei der PCR zu keiner Veränderung der erzeugten DNA kommen soll. Weiterhin werden in verschiedenen Methoden der isothermalen DNA-Amplifikation bei Raumtemperatur strangversetzende DNA-Polymerasen wie die φ29-DNA-Polymerase verwendet. Der Vorläufer der heute verwendeten DNA-Polymerasen war die T4-DNA-Polymerase.
Literatur
- Lehninger; David Nelson, Michael Cox: Lehninger Biochemie. 3. Auflage, Springer-Verlag, Berlin Heidelberg 2001, ISBN 3-540-41813-X
- Wilhelm Seyffert: Lehrbuch der Genetik. 2. Auflage, Spektrum Akademischer Verlag Heidelberg, Berlin 2003, ISBN 3-8274-1022-3
- Jeremy M. Berg, John L. Tymoczko, Lubert Stryer: Biochemie 6. Auflage, Springer-Verlag, Heidelberg 2007, ISBN 978-3-8274-1800-5
Weblinks
- PDB Molecule of the Month: DNA-Polymerase
Einzelnachweise
- Donald Voet: Biochemistry. 4th edition Auflage. John Wiley & Sons, Hoboken, NJ 2011, ISBN 978-0-470-57095-1, S. 1201.