DNA-Polymerase III
Die DNA-Polymerase III ist ein Enzym, welches die Synthese von DNA aus Desoxyribonukleotiden an einer DNA-Matrize katalysiert. Es handelt sich um einen Proteinkomplex. Das Holoenzym spielt die wichtigste Rolle bei der prokaryotischen DNA-Replikation. Wichtigste Merkmale sind seine vielen Untereinheiten und die sehr hohe Katalysatorwirkung, Genauigkeit und Prozessaktivität (Fähigkeit eines Enzyms viele Reaktionen hintereinander zu katalysieren ohne das Substrat zu verlieren). Neben DNA-Polymerase III sind auch noch zwei weitere DNA-Polymerasen in Prokaryoten bekannt.
| Übergeordnet |
| DNA-Polymerase |
| Gene Ontology |
|---|
| QuickGO |
Funktion
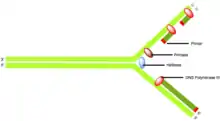
Die DNA-Polymerase III knüpft mehrere tausend Phosphodiesterbindungen mit seinem Substrat, bevor es dieses verlässt. Somit kann sie die Matrize festhalten und erst nach der vollständigen Replizierung wieder entlassen. Des Weiteren verfügt die DNA-Polymerase III über eine starke Katalysatorwirkung, die es ihr ermöglicht pro Sekunde 1000 Nukleotide anzufügen. Diese starke Katalysatorleistung ist damit zu erklären, dass sie sich nicht vom Substrat lösen muss wie zum Beispiel DNA-Polymerase I. Der neue Strang wächst in 5'→3' Richtung. Chemisch betrachtet findet ein nukleophiler Angriff der endständigen 3'-OH des DNA-Stranges auf das 5'-Phosphat des dNTPs statt, wobei Pyrophosphat abgespalten wird. Die DNA-Polymerase benötigt eine freie 3'-Hydroxygruppe, also einen Primer, um daran Nukleotide anzusetzen. Außerdem ist es der DNA-Polymerase III möglich 3'→5' Korrektur zu lesen und falsch eingebaute Nukleotide über die Exonukleaseaktivität zu ersetzen.
Aufbau
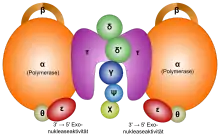
Das Holoenzym ist verglichen mit der DNA-Polymerase I um eine Zehnerpotenz schwerer und die Molekülmasse liegt bei fast 900 kDa. Das Enzym ist als asymmetrisches Dimer aufgebaut, um beide Elternstränge am selben Ort zur gleichen Zeit zu replizieren. Die Asymmetrie rührt daher, dass Leit- und Folgestrang unterschiedlich synthetisiert werden.
Die α-Untereinheit ist die Polymerase und die ε ist die 3'→5'-Exonuklease für das Korrekturlesen. Beide sind katalytisch aktiv aber nicht prozessiv, das übernehmen die Untereinheiten β und τ. Die Prozessivität lässt sich durch die Raumstruktur von β erklären. Diese Untereinheit bildet eine Klammer durch die der DNA-Doppelstrang hindurchgleitet und sich so nicht vom Substrat trennen muss.
Replikation
Die ATP-betriebene Helikase entwindet den DNA-Doppelstrang und ermöglicht die Nutzung beider Stränge als Matrize. SSB-Proteine halten die Stränge für den Zeitraum der Replikation auseinander. Es werden gleichzeitig der Leit- und der Folgestrang synthetisiert, jedoch nicht in derselben Art und Weise. Das Holoenzym beginnt mit dem Leitstrang, indem der von der Primase gesetzte Primer gebunden wird und synthetisiert diesen kontinuierlich. Der Folgestrang wird komplizierter synthetisiert, weil dieser von 5'→3' verläuft und eine Synthese nicht von 3'→5' verlaufen kann. Daher wird er in Fragmenten synthetisiert, was in vielen einzelnen 5'-3' Synthesen resultiert. Die Fragmente werden auch Okazaki-Fragment genannt. Ein Fragment ist immer ein zu einer Schleife gewundener Teil des DNA-Einzelstranges, das im aktiven Zentrum der α-Untereinheit ist und in derselben Richtung läuft wie der Leitstrang. Nach ca. 1000 Nucleotiden wird der Folgestrang entlassen und die nächste Schleife in das aktive Zentrum geführt, dazu setzt die Primase wieder einen Primer. Im Anschluss werden die RNA-Reste der Primer durch die DNA-Polymerase I entfernt, da der DNA-Polymerase III die Exonukleasefunktion 5'→3' fehlt. Die entstehenden Lücken werden durch die langsamere DNA-Polymerase I aufgefüllt und durch die Ligase verknüpft.
Quellen
- Jeremy M. Berg, John L. Tymoczko, Lubert Stryer: Biochemie. Berlin/ Heidelberg 2003, ISBN 3-8274-1303-6.
- T. Michael Madigan, M. John Martinko, Jack Parker: Biology of Microorganisms. London 2003, ISBN 0-13-066271-2.