Influenza-A-Virus H1N1
Influenza-A-Virus H1N1 (A/H1N1) bezeichnet einen Subtyp des Influenza-A-Virus (Gattung Alphainfluenzavirus) aus der Familie Orthomyxoviridae,[3] der bei Enten, Menschen und Schweinen vorkommt, aber auch zahlreiche andere Säugetierarten sowie Truthähne infizieren kann. Bei Menschen und Schweinen haben sich H1N1-Influenzaviren durch die Spanische Grippe von 1918 etabliert. Dieser Subtyp hat unter Menschen einige zehn Millionen Tote verursacht. Im Frühjahr 2009 breitete sich in Nordamerika ein zuvor unbekannter Subtyp des H1N1-Virus aus, das so genannte Schweinegrippevirus, und verursachte eine neuerliche Pandemie, die Pandemie H1N1 2009/10.
| Influenza-A-Virus H1N1 | ||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
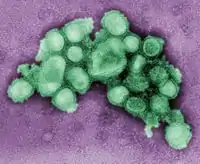
TEM-Aufnahme von H1N1-Influenza-A-Viren | ||||||||||||||||||||||
| Systematik | ||||||||||||||||||||||
| ||||||||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||||||||
| ||||||||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||||||||
| Influenza A virus A/H1N1 | ||||||||||||||||||||||
| Kurzbezeichnung | ||||||||||||||||||||||
| FLUAV/(H1N1) | ||||||||||||||||||||||
| Links | ||||||||||||||||||||||
|
Der erste Ausbruch unter Menschen: die Spanische Grippe
Die Spanische Grippe von 1918 zeigte mehrere Besonderheiten: Mit diesem H1N1 war mutmaßlich ein neuer Influenza-Subtyp aufgetaucht. Die ersten drei Wellen folgten innerhalb weniger Monate aufeinander, was sehr ungewöhnlich ist, und sie hielten sich erst mit der dritten Welle an die in den gemäßigten Breiten übliche „Influenza-Saison“ im Spätwinter. Die Sterblichkeit konzentrierte sich auf die Altersgruppe der 15- bis 40-Jährigen; als extrem gefährdet erwiesen sich Schwangere im letzten Drittel ihrer Schwangerschaft. Obwohl die Datenlage in diesem Punkt widersprüchlich ist, scheint der Erreger der Spanischen Grippe auch infektiöser als übliche Influenzaviren gewesen zu sein. Die Seuche führte in den verschiedenen Regionen der Welt zu einer sehr unterschiedlichen Sterblichkeit, wobei generell Länder in tropischen und subtropischen Klimazonen besonders schwer betroffen wurden.
Die Spanische Grippe war die größte Seuche der Neuzeit, auch wenn die genaue Zahl der Toten unbekannt bleibt. Zu den meisten Todesfällen kam es nicht direkt durch das Virus, sondern durch sekundäre bakterielle Lungenentzündungen. Jahrzehntelang ging man von 21 Millionen Todesopfern aus,[4] was zu niedrig angesetzt erscheint, denn allein in Britisch-Indien kamen 17 bis 18 Millionen Menschen um; diese Angabe ist durch Volkszählungen gut abgesichert.[5] Eine auch nur annähernd genaue Bilanz lässt sich nicht ziehen, weil in zwei bevölkerungsreichen Staaten – dem ehemaligen Zarenreich und China – Bürgerkrieg herrschte. Eine Hochrechnung für diese beiden Staaten auf der Basis der indischen Zahlen ist wegen der dort ganz anders gelagerten Verhältnisse unseriös.

Die Umstände des Ausbruchs sind ungeklärt. Erste sichere Nachrichten beziehen sich auf Soldaten, die im März 1918 in Ausbildungslagern der USA für den Einsatz im Ersten Weltkrieg vorbereitet wurden. Von den Vereinigten Staaten aus wurden Japan, die Philippinen, Kanada und Mexiko infiziert; US-amerikanische Soldaten brachten die Krankheit an die Westfront in Frankreich, von wo aus sie sich auch nach Spanien und Südamerika, dem restlichen Europa, Afrika und Asien verbreitete. Weil internationale Presseagenturen die Seuche zuerst aus Spanien meldeten, wurde ihre Herkunft fälschlich diesem Land zugeschrieben.
Wasservögel bilden das ursprüngliche Reservoir von Influenzaviren. Jeffery Taubenberger, der später den Erreger der Spanischen Grippe rekonstruiert hat, meint, dass kurz vor 1918 ein komplettes Influenzavirus von Vögeln auf Menschen übergegangen sei.[6] Andere Forscher widersprechen ihm und vermuten eine Rekombination (englisch: reassortment) mit Gensegmenten aus Schweine-Influenzaviren.[7] Zur Zeit der Spanischen Grippe wurde aus Harbin in China[8] sowie Iowa in den Vereinigten Staaten[9] auch von Erkrankungen unter Schweinen berichtet, aber zumindest in diesen Fällen wirkt die Abfolge so, als ob sie von Menschen infiziert worden wären. Auch genetische Studien am Hämagglutinin-Gen legen diesen Ablauf nahe.
Ob 1918 wirklich zum ersten Mal H1N1-Viren unter Menschen umgelaufen sind, ist umstritten. Man kann den relativen Schutz der über 45-Jährigen in der Spanischen Grippe auch so interpretieren, dass sie in der Zeit vor 1873 schon einmal H1N1-Viren ausgesetzt und dadurch immun waren.
Mit der dritten Welle im Winter 1918/19 fügte sich die Spanische Grippe wieder in die übliche Influenza-Saison in den gemäßigten Breiten ein. Aber noch die vierte Welle im Winter 1919/20 zeigte – wenn auch abgeschwächt – die charakteristische erhöhte Sterblichkeit bei jungen Erwachsenen. Infolge der Seuche etablierten sich unter Menschen und Schweinen stabile Linien von H1N1-Viren. Es ist davon auszugehen, dass Influenzaviren vom Subtyp H1N1 in der Menschheit seit 1918 zirkulierten, aufgrund von Virus-Isolaten aus diesem Jahr.[10] Schwere Influenza-Epidemien, die mindestens Europa und Nordamerika erfassten, hat es noch 1928/29[11], 1932/33,[12] 1936/37 sowie 1943/44 gegeben.
Die Entdeckung des Virus

Während der Spanischen Grippe war der Erreger noch unbekannt. Manche Forscher hatten bereits einen „filtrierbaren Virus“ in Verdacht (wobei „Virus“ zunächst die Bedeutung von „Krankheitserreger“ oder sogar „Gift“ hatte). In den Lehrbüchern galt jedoch das Bakterium, das heute Haemophilus influenzae genannt wird, als Verursacher der Influenza. Mit den üblichen Methoden der Bakteriologie ließ sich jedoch kein Erreger nachweisen: Er ließ sich weder färben, noch auf künstlichen Kulturmedien züchten. Wenn es einen Erkenntnisfortschritt durch die Spanische Grippe gegeben hat, dann insofern, als der tatsächliche Erreger dieser Grippevariante zum damaligen Zeitpunkt noch unbekannt war, Haemophilus influenzae als Erreger also ausschied.
Die klassische Schweineinfluenza blieb lange Zeit auf den Norden und Mittleren Westen der Vereinigten Staaten beschränkt, wo es nach 1918 zu jährlichen Ausbrüchen in der Winterzeit kam. 1930 isolierte Richard Shope am Rockefeller-Institut in Princeton zum ersten Mal ein Influenzavirus aus Schweinen.[13] Zwei Jahre später gelang es den britischen Forschern Wilson Smith, Christopher Andrewes und Patrick Laidlaw, auch aus Menschen Influenzaviren zu gewinnen.[14] Die einzige Methode, das Virus in Kultur zu halten, war, Frettchen mit Rachenabstrichen von Menschen zu infizieren und dann die Infektion von Frettchen zu Frettchen zu übertragen, bevor das jeweils infizierte Tier immun wurde. Später wurde auf Mäuse gewechselt.
Die Entwicklung eines Impfstoffs
1935 zeigte Wilson Smith, dass sich das Virus gut in befruchteten Hühnereiern vermehren lässt, eine Technik, die bereits seit 1932 bei anderen Viren angewandt wurde.[15] Damit war die Forschung nicht mehr ausschließlich auf Versuchstiere angewiesen. Joseph Stokes von der Universität von Pennsylvania begann dann zwischen 1936 und 1938 mit den ersten Tierversuchen mit Influenzaimpfstoffen. Die Ergebnisse waren unklar. Mit den in den 1940er-Jahren entwickelten Antibiotika ließ sich das störende Wachstum von Bakterien in den beimpften Hühnereiern behindern. Außerdem wurden verschiedene Techniken ausgearbeitet, die Viren anzureichern.
Im Zweiten Weltkrieg wurde im Auftrag des Verteidigungsministeriums der Vereinigten Staaten ein Impfstoff entwickelt, der modern anmutet. Er enthielt drei verschiedene Virusstämme, war konzentriert und mit Formalin inaktiviert. Auch der folgende Impfstofftest war innovativ: Die zur Impfung vorgesehenen Probanden wurden zufällig ausgesucht (randomisiert), die Kontrollgruppe erhielt ein Placebo, und die Studie war doppel-blind angelegt. In der Grippewelle von 1943 erbrachte der Impfstoff exzellente Resultate, und bis 1945 waren sämtliche US-Soldaten geimpft. In einer schweren Epidemie mit einem Influenza-B-Virus in diesem Jahr erwies sich der Impfstoff ebenfalls als hochwirksam.[16] 1945 erhielten auch verschiedene Hersteller in den USA die Lizenz, Influenza-Impfstoffe für Zivilisten zu produzieren.
Weitere Ausbrüche
Die Epidemien von 1947 und 1951
1947 kam es zu einer Rekombination von Gensegmenten innerhalb des Subtyps H1N1, die zu einem kompletten Versagen des Impfstoffs führte. Vor allem das Hämagglutinin wich in zahlreichen Aminosäuren von seinem Vorgänger ab (Referenzstamm: A/Fort Monmouth (FM)/1/1947, damals meistens FM-1-Virus genannt). Das Virus breitete sich zwar weltweit aus, aber die Sterblichkeit blieb relativ niedrig. Dieses Ereignis führte langfristig zu der Einsicht, dass es unmöglich sein würde, einen Influenza-Impfstoff herzustellen, der auf Dauer schützt. Die Impfung muss vor allem wegen der allmählichen Veränderung der Gene durch Punktmutationen (genetische Drift) jedes Jahr den umlaufenden Influenza-Stämmen angepasst werden. Die 1948 gegründete Weltgesundheitsorganisation baute dafür ein Überwachungssystem auf.
Eine weitere schwere Epidemie ereignete sich 1950/51, in der die Sterblichkeit im Vereinigten Königreich und in Kanada sogar die Pandemien von 1957 und 1968 übertraf. 1957 verschwanden H1N1-Viren aus der Menschheit und wurden durch die H2N2-Viren der Asiatischen Grippe ersetzt. Daraus ist geschlossen worden, dass H1- und H2-Viren in einer Population nicht koexistieren können.[17]
Die klassische Schweineinfluenza breitet sich aus
Während des Zweiten Weltkriegs oder danach muss sich auch die klassische Schweineinfluenza über die Welt verbreitet haben. Sie wurde in Kanada und Brasilien, ganz Asien, Kenia, Großbritannien, der Tschechoslowakei und 1959 auch in der Bundesrepublik Deutschland festgestellt. Danach verschwand sie aus Europa und tauchte erst 1976 bei einem Ausbruch in Norditalien wieder auf. Die dort isolierten Viren erwiesen sich als eng verwandt mit nordamerikanischen Stämmen, so dass die Krankheit wahrscheinlich aus den USA importiert worden war. Sie blieb bis 1979 auf Norditalien beschränkt und breitete sich dann über Westeuropa, Skandinavien und Großbritannien aus. Auch in den Vereinigten Staaten und Asien blieb die klassische Schweineinfluenza bis in die 1990er-Jahre die dominierende Influenzaform bei Schweinen.
Die jeweils unter Menschen kursierenden H1N1-Viren können sporadisch auch Schweine infizieren, wie bereits 1941 in Budapest dokumentiert,[18] etablieren sich dann aber nicht unter Schweinen. Ausnahme ist der Erreger der Spanischen Grippe, der zur klassischen Schweineinfluenza geführt hat.
Beim Menschen unterliegen H1N1-Viren einer ständigen Veränderung, verursacht durch Punktmutationen und befördert durch den Selektionsdruck des Immunsystems. Das Virus der klassischen Schweineinfluenza dagegen ist über Jahrzehnte hinweg genetisch stabil geblieben. Die Mutationen sind nicht seltener, aber die Selektion wirkt stets ungestört in Richtung optimaler Anpassung an den Wirt, da in der Schweinehaltung ständig junge, noch nicht immune Schweine in die Bestände eingeführt werden. Ein Forscher beobachtete, dass Influenzaviren ein ganzes Jahr lang innerhalb einer Schweineherde zirkulierten.
Die „Schweinegrippe“ von 1976

„Schweinegrippe“ bezeichnet in der Folge im Unterschied zur Schweineinfluenza eine Erkrankung beim Menschen. Im Jahre 1976 kam es zu einem lokalen Ausbruch der Variante A/New Jersey/1/1976 unter US-Soldaten in Fort Dix, New Jersey.[19]
Die Gesundheitsbehörden der Vereinigten Staaten schätzten die Gefahr einer Wiederkehr einer Seuche vom Ausmaß der Spanischen Grippe als so groß ein, dass sie ein Impfstoffproduktionsprogramm starteten, das jeden Bürger der Vereinigten Staaten schützen sollte. Die ersten Impfungen fanden am 1. Oktober 1976 statt, bis Mitte Dezember hatten sich 40 Millionen US-Amerikaner impfen lassen – bis dahin die größte Impfkampagne der Geschichte. In Europa schlossen sich einzig die Niederlande dem Impfprogramm an. Gleichzeitig wurde bekannt, dass einige Geimpfte ein Guillain-Barré-Syndrom entwickelt hatten; bis heute ist umstritten, ob es sich dabei tatsächlich um eine Nebenwirkung der Impfung handelt. Es kam zu einer Flut von Schadenersatzklagen über eine Summe von 3,5 Milliarden US-Dollar.[20]
Dass Schweine die Quelle der Influenzaviren in Fort Dix waren, ist zwar wahrscheinlich, aber nie belegt worden.
Die Russische Grippe von 1977
Im Mai 1977 kam es in Tianjin, China zu einem Ausbruch eines H1N1-Virus, nachdem dieser Subtyp zwanzig Jahre lang verschwunden gewesen war. Im November hatte sich die Epidemie bis in die Sowjetunion – wodurch sie ihren Namen erhielt – und nach Hongkong ausgebreitet. Das Virus der Russischen Grippe erwies sich als nahezu identisch mit den vor 1957 umlaufenden Viren, speziell A/Roma/1949,[21] sodass allgemein vermutet wird, dass es versehentlich aus einem Labor freigesetzt worden ist.[22] Allerdings bleiben Influenzaviren auch in Vögeln genetisch stabil, sodass das Virus der Russischen Grippe zwischen 1957 und 1977 auch in diesem Reservoir überdauert haben könnte.
Seit 1977 zirkulieren H1N1-Viren gemeinsam mit den H3N2-Viren aus der Hongkong-Grippe von 1968 in der Menschheit. H1- und H3-Viren können also miteinander koexistieren. Dabei fällt die Sterblichkeit in einer Influenza-Saison, in der H1N1 dominiert, eher gering aus. Außerdem geht in Deutschland die mit Influenza verbundene Sterblichkeit im langfristigen Trend zurück.[23]
H1N1-Viren beim Menschen verändern sich auch nur noch langsam durch genetische Drift, was sich daran zeigt, dass für sie die Zusammensetzung des Impfstoffs nur noch selten geändert werden muss. Einer der drei Subtypen im Impfstoff ist traditionell für einen H1N1-Stamm reserviert, für die Saison 2009/2010 in der nördlichen Hemisphäre ist A/Brisbane/59/2007 vorgesehen.
H1N1-Pandemie 2009
Im Frühjahr 2009 breitete sich ein neuartiger Subtyp „A/California/7/2009 (H1N1)“ des H1N1-Virus zunächst in Nordamerika aus und verursachte bald darauf eine Pandemie. Einer 2016 publizierten Studie zufolge entstand die pandemische Variante des Virus (pdmH1N1) in Schweinen in Mexiko.[24]
Das Virus selbst, aber auch die Impfung, kann in seltenen Fällen zu Narkolepsie führen.[25] Zwischen 2010 und Januar 2015 wurden laut der EudraVigilance-Datenbank der Europäischen Arzneimittelagentur mehr als 1300 Fälle bei geimpften Personen bekannt, darunter einige auch aus Deutschland.[26] Mit Stand vom 28. November 2016 waren in Deutschland 86 Menschen, die mit Pandemrix geimpft worden waren, dem Paul-Ehrlich-Institut als „Narkolepsie-Verdachtsfälle“ gemeldet worden.[27] In Schweden wurde 2016 ein Gesetz für die Entschädigung von Betroffenen verabschiedet.[28]
Eine allgemeine Nomenklatur für Influenzaviren
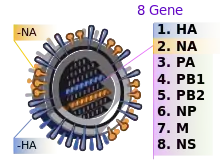
Um die Erkenntnis auszudrücken, dass Influenzaviren über Artgrenzen hinweg übertragen werden können, setzte die Weltgesundheitsorganisation ab 1980 eine Nomenklatur durch, die sich an den beiden wichtigsten Oberflächenantigenen Hämagglutinin (= H) und Neuraminidase (= N) orientiert. Auf die Wirtsart wird dabei keine Rücksicht mehr genommen.
Die Rekonstruktion des Erregers der Spanischen Grippe
Eine Arbeitsgruppe um Jeffery Taubenberger, damals am Pathologischen Institut der Streitkräfte (Armed Forces Institute of Pathology) in Washington, D.C., hat ab 1996 mit Gewebeproben aus drei verschiedenen Quellen das Genom des Erregers der Spanischen Grippe sequenziert. Damit konnten sie bestätigen, dass es sich tatsächlich um ein H1N1-Virus handelte. Aus der genetischen Sequenz konnte auch ein Virus rekonstruiert werden, mit dem seitdem experimentiert wird. Es erwies sich für Mäuse, Frettchen und Makaken als tödlich, während es bei Schweinen nur zu einer leichten Erkrankung führt.
Im Tierversuch an Makaken löste der rekonstruierte Erreger eine Überreaktion im angeborenen Zweig des Immunsystems aus, der auf Virusinfektionen reagiert. Ihr Körper produzierte dermaßen viele Zytokine und Chemokine („Zytokinsturm“), dass die Regulation der Immunabwehr zusammenbrach.[29] Derselbe Ablauf ist auch für die menschlichen Opfer der Spanischen Grippe wahrscheinlich und würde erklären, warum besonders junge Erwachsene mit einem kräftig entwickelten Immunsystem starben.
Das Influenza-A-Virus 1918 (H1N1) wurde 2006 der Risikogruppe 3 zugeordnet, während es zuvor in der Risikogruppe 2 eingestuft war. Arbeiten mit ihm dürfen folglich nur unter den strengen Sicherheitsvorkehrungen der Schutzstufe 3 erfolgen. Die Zuordnung gilt nur für das Virus der Spanischen Grippe, davon abstammende Influenza-A-Viren H1N1, wie das Klassische Schweineinfluenzavirus H1N1, das Eurasische Schweineinfluenzavirus H1N1 oder das an der Pandemie 2009/10 beteiligte Virus H1N1 verbleiben durch die Biostoffverordnung in Verbindung mit der TRBA (Technische Regeln für Biologische Arbeitsstoffe) 466 der Risikogruppe 2 zugeordnet.[30]
H1N1-Viren, die direkt von Erregern in Vögeln abstammen
Alle bisher besprochenen H1N1-Viren bilden eine Abstammungsgemeinschaft, die auf den Erreger der Spanischen Grippe zurückgeht. Seit 1979 dominieren bei europäischen Schweinen jedoch H1N1-Viren, die direkt von Vögeln übertragen worden sind (englisch: avian-like swine influenza). Sie haben sich als eng verwandt mit H1N1-Viren aus Enten erwiesen. Da sämtliche Gensegmente aus Vögeln stammen, ist anscheinend ein komplettes Virus übertragen worden. In Europa haben diese aus dem Vogelreich stammenden Viren die klassische Schweineinfluenza innerhalb weniger Jahre verdrängt. Unabhängig davon scheint 1993 dieser Prozess ein zweites Mal in Südostasien abgelaufen zu sein; dort zirkulieren die von Vögeln stammenden Viren noch gemeinsam mit der klassischen Schweineinfluenza. Die ursprünglichen Vogel-Viren aus Europa und Südostasien haben sich als miteinander verwandt erwiesen.
Übertragung von Influenzaviren von Schweinen auf Vögel
Aus Nordamerika ist über die Übertragung von H1N1-Influenzaviren von Schweinen auf Truthähne berichtet worden. Mehrfach erkrankten Truthähne unmittelbar nach dem Ausbruch einer Influenza bei Schweinen, und genetische Studien haben einen lebhaften Austausch von Influenza-A-Viren zwischen den beiden Arten belegt. In einem Fall wurde auch ein Mensch von einem Schweineinfluenza-Virus durch einen Truthahn infiziert. In Europa haben die ursprünglich von Enten stammenden H1N1-Schweineinfluenza-Viren ebenfalls Truthähne infiziert. Es besteht also die Möglichkeit, dass Schweine, Menschen, Truthähne und Enten sich immer wieder gegenseitig anstecken.
H1N1-Viren als Rekombinanten aus verschiedenen Quellen
Das Schwein als potenzielles „Mischgefäß“
Schweine sind die einzige Säugetierart, die in großer Zahl gehalten wird und in denen sich sowohl Influenzaviren aus Vögeln als auch Menschen vermehren können. Das liegt daran, dass die Luftröhre von Schweinen mit Zellen ausgekleidet ist, die zweierlei Sorten von Sialinsäure tragen, die Influenzaviren von Menschen beziehungsweise Vögeln als Andockstelle dienen können. Da das Genom der Influenza-A-Viren auf acht Segmente verteilt ist, können sich diese Segmente bei einer Doppelinfektion mit verschiedenen Viren neu kombinieren (reassortment). Schweine bilden ein mögliches „Mischgefäß“ (mixing vessel)[31] für verschiedene Varianten von Viren und so ein potenzielles Bindeglied zwischen Vögeln und Menschen.
Übertragung von Schweine-Influenzaviren von Schweinen auf Menschen
Welche Rolle Schweine bei der Entstehung der Spanischen Grippe gespielt haben, ist umstritten. Seit den 1970er-Jahren sind mehr als 50 Infektionen von Menschen mit der klassischen Schweine-Influenza in der wissenschaftlichen Literatur dokumentiert worden. Aus Studien an Serum von beruflich exponierten Menschen ist bekannt, dass noch mit einer viel größeren Zahl zu rechnen ist.
Dreifachkombinanten
Bis in die 1990er-Jahre war das klassische Schweine-Influenzavirus das häufigste Influenzavirus unter nordamerikanischen Schweinen. In den späten 1990er-Jahren wurde es durch verschiedene Stämme und Subtypen (H1N1, H3N2 und H1N2) abgelöst, die Gensegmente aus drei verschiedenen Quellen enthielten (englisch: triple-reassortant swine influenza). Die Basis bildete dabei meist das H1-Schweine-Influenzavirus nordamerikanischer Herkunft mit weiteren Gensegmenten von Influenzaviren aus Vögeln sowie Menschen.
Im Dezember 2005 wurde zum ersten Mal die Infektion eines Menschen durch eine solche Dreifachkombination in Wisconsin beobachtet. Im Juni 2007 erklärten die Vereinigten Staaten solche Infektionen für meldepflichtig. Bis zum Februar 2009 wurden insgesamt elf Fälle aus dem Mittleren Westen und Süden der USA bekannt (davon zehn H1N1, einer H1N2). Neun der Betroffenen hatten sich in der Nähe von Schweinen aufgehalten. Die Patienten waren sehr jung, das Medianalter lag bei 10 Jahren.[32]
Eine Dreifachkombination mit „Pandemiepotenzial“[33] wurde zwischen 2011 und 2018 und seit 2016 vorherrschend in Schweinebeständen der Volksrepublik China nachgewiesen.[34] Die Gene der als „G4 EA H1N1“ bezeichneten H1N1-Variante entstammen teils einer aus europäischen und asiatischen Vögeln bekannten Linie, teils dem pandemischen H1N1-Virus aus dem Jahr 2009 („Schweinegrippe“) und teils einer aus Nordamerika bekannten Linie, die ihrerseits aus mehreren Herkünften zusammengesetzt ist.[35] Laut einer im Juni 2020 im Fachblatt PNAS veröffentlichten Studie sind bereits mehr als 10 Prozent der Schweinehalter (35 von 338 untersuchten Personen) infiziert. Zudem wurde darauf hingewiesen, dass eine Infektion mit saisonaler Influenza keinen Schutz vor „G4 EA H1N1“ bietet.
Vierfachkombinanten
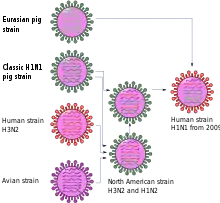
A/California/7/2009 (H1N1)
Die erste und bisher einzige Vierfachkombination wurde 2009 entdeckt. Obwohl sie bereits an den Menschen angepasst war, siehe Influenza-Pandemie 2009, wurde sie vielfach als „Schweinegrippe“ bezeichnet, denn sie war aus Schweine-Influenzaviren und vermutlich in Schweinen entstanden.
Die erste wissenschaftliche Veröffentlichung[36] beschrieb die neue Virusvariante als eine genetische Neukombination aus zwei Viruslinien der Schweineinfluenza, einer nordamerikanischen und einer eurasischen. Drei der acht Gensegmente – NP, NS und ein das H1-Hämagglutinin codierendes – gehen auf das klassische Schweineinfluenza-Virus von 1918 zurück. Zwei Gensegmente – PB2 und PA – sind etwa 1998 von Vögeln auf nordamerikanische Schweine übergegangen und PB1 etwa 1968 von Vögeln auf Menschen und von dort etwa 1998 auf Schweine. Diese Kombination von sechs Gensegmenten findet sich in Dreifachkombinanten, die in Nordamerika verbreitet sind. Dagegen stammen die beiden übrigen Gensegmente, die eine N1-Neuraminidase bzw. die Matrixproteine M1 und M2 codieren, von jener ursprünglich unter Vögeln verbreiteten Variante ab, die seit 1977 unter eurasischen Schweinen verbreitet ist, aber in Amerika nie nachgewiesen worden war. Das Gen für das Matrix-Protein enthält eine Mutation, die das Virus gegen die Influenza-Medikamente Amantadin und Rimantadin resistent macht.
Siehe auch
Literatur
- Report on the Pandemic of Influenza, 1918–1919. Ministry of Health (Hrsg.). In: Reports on Public Health and Medical Subjects. Band 4, His Majesty’s Stationery Office, London 1920 (bester zeitgenössischer Überblick zur Spanischen Grippe)
- Ian H. Brown: The epidemiology and evolution of influenza viruses in pigs. In: Veterinary Microbiology. Band 74, Nr. 1–2, 2000, S. 29–46, PMID 10799776 (Übersichtsartikel zur Schweineinfluenza)
Weblinks
- Viren Schlitzohren im Mikrokosmos. Planet Wissen – Sendung vom 14. Januar 2011
Belege
- ICTV Master Species List 2018b.v2. MSL #34, März 2019
- ICTV: ICTV Taxonomy history: Akabane orthobunyavirus, EC 51, Berlin, Germany, July 2019; Email ratification March 2020 (MSL #35)
- Negative Sense RNA Viruses: Orthomyxoviridae, in: ICTV 9th Report (2011)
- Edwin O. Jordan: Epidemic Influenza. A Survey. American Medical Association, Chicago 1927, S. 229.
- I. D. Mills: The 1918–1919 Influenza Pandemic. The Indian Experience. In: The Indian Economic and Social History Review. Band 23, Nr. 1, 1986, S. 1–40.
- Jeffery K. Taubenberger et al.: Characterization of the 1918 influenza virus polymerase genes. In: Nature, Band 437, 2005, S. 889–893.
- Mark J. Gibbs und Adrian J. Gibbs: Was the 1918 pandemic caused by a bird flu? sowie Janis Antonovics, Michael E. Hood und Christi Howell Baker: Was the 1918 flu avian in origin? beide in: Nature. Band 440, 2006, S. E8f.
- J. W. H. Chun: Influenza Including its Infection Among Pigs. In: The National Medical Journal of China. Band 5, Nr. 1, 1919, S. 34–44.
- J. S. Koen: A Practical Method for Field Diagnosis of Swine Diseases. In: American Journal of Veterinary Medicine. Band 14, 1919, S. 468–470.
- http://www.pandemicinfluenza.org/about.html (Memento vom 30. Januar 2010 im Internet Archive) Katzelab
- Selwyn D. Collins: The Influenza Epidemic of 1928-1929 with Comparative Data for 1918-1919. In: American Journal of Public Health and the Nations Health. Band 20, Nr. 2, Februar 1930, ISSN 0002-9572, S. 119–129, doi:10.2105/AJPH.20.2.119, PMID 18012936, PMC 1555806 (freier Volltext) – (aphapublications.org [abgerufen am 18. Februar 2021]).
- E. Eckardt, H. Flohn und H. J. Jusatz: Ausbreitung und Verlauf der Grippeepidemie 1933 in Abhängigkeit von meteorologischen und geographischen Faktoren. In: Zeitschrift für Hygiene und Infektionskrankheiten. Band 118, 1936, S. 64–91, doi:10.1007/BF02177495.
- Richard E. Shope: Swine Influenza. III. Filtration Experiments and Etiology. In: Journal of Experimental Medicine. Band 54, 1931, S. 373–385.
- Wilson Smith, C. H. Andrewes und P. P. Laidlaw: A Virus Obtained From Influenza Patients. In: The Lancet. Band 222, 1933, S. 66–68.
- W. I. B. Beveridge: Influenza. The Last Great Plague. Heinemann, London 1977, S. 9.
- John M. Eyler: De Kruif’s Boast: Vaccine Trials and the Construction of a Virus. In: Bulletin of the History of Medicine. Band 80, Nr. 3, 2006, S. 409–438.
- Martha I. Nelson et al.: Multiple Reassortment Events in the Evolutionary History of H1N1 Influenza A Virus Since 1918. In: PLoS Pathogens. Band 4, Nr. 2, 2008.
- Miklós Dreguss: Über die Infektion von Schweinen mit menschlichem Influenzavirus. In: Archives of Virology. Band 3, Nr. 1–4, 1944, S. 35–48.
- J. C. Gaydos et al.: Swine influenza A outbreak, Fort Dix, New Jersey, 1976. In: Emerging Infectious Diseases. Band 12, 2006, S. 23–28; Volltext (auf Englisch)
- Gina Kolata: Influenza. Die Jagd nach dem Virus. S. Fischer, Frankfurt 2001, S. 153–213
- Martha I. Nelson et al.: Multiple Reassortment Events in the Evolutionary History of H1N1 Influenza A Virus Since 1918. In: PLoS Pathogens. Bd. 4, Nr. 2, 29. Februar 2008, e1000012, doi:10.1371/journal.ppat.1000012.
- Alan P. Kendal et al.: Antigenic Similarity of Influenza A (H1N1) Viruses from Epidemics in 1977–1978 to 'Scandinavian' Strains Isolated in Epidemics of 1950–1951. In: Virology. Band 89, 1978, S. 632–636.
- Influenza-assoziierte Mortalität in Deutschland 1985–2006. In: Epidemiologisches Bulletin. Nr. 35, 2007, S. 325–327.
- Ignacio Mena et al.: Origins of the 2009 H1N1 influenza pandemic in swine in Mexico. In: eLife. Online-Veröffentlichung vom 28. Juni 2016, doi:10.7554/eLife.16777
- Syed Sohail Ahmed et al.: Antibodies to influenza nucleoprotein cross-react with human hypocretin receptor 2. In: Science Translational Medicine. Band 7, Nr. 294, 2015, S. 294ra105, doi:10.1126/scitranslmed.aab2354
Wie H1N1 die Schlafkrankheit auslösen kann. Zeit Online, 1. Juli 2015 - Grippeimpfung: Wie Pandemrix eine Narkolepsie auslöst. Auf: aerzteblatt.de vom 2. Juli 2015
- Aktuelle Informationen zu Narkolepsie im zeitlichen Zusammenhang mit A/H1N1-Influenzaimpfung. Auf: pei.de vom 28. November 2016
- Schaden nach Schweinegrippe-Impfung: Schweden entschädigt Narkolepsie-Patienten. Spiegel Online, 13. Mai 2016
- Darwyn Kobasa: Aberrant innate immune response in lethal infection of macaques with the 1918 influenza virus. In: Nature. Band 445, 2007, S. 319–323.
- TRBA (Technische Regeln für Biologische Arbeitsstoffe) 462: Einstufung von Viren in Risikogruppen. In: Website der Bundesanstalt für Arbeitsschutz und Arbeitsmedizin (BAuA). 25. April 2012, abgerufen am 13. Mai 2014.
- Hermann Müller: Die Klassische Geflügelpest („Hoch pathogene Aviäre Influenza“; „Vogelgrippe“) – Hintergrundinformationen zum aktuellen Seuchengeschehen in Südostasien und Hinweise auf Informationsmöglichkeiten im Internet. (PDF) Direktor des Instituts für Virologie, Veterinärmedizinische Fakultät, Universität Leipzig, Stand: 2004
- Vivek Shinde et al.: Triple-Reassortant Swine Influenza A (H1) in Humans in the United States, 2005–2009. In: The New England Journal of Medicine. Band 360, Nr. 25, 2009, S. 2616–2625, doi:10.1056/NEJMoa0903812.
- Neue Art der Schweinegrippe mit Pandemiepotenzial. Auf: science.orf.at vom 30. Juni 2020.
- Honglei Sun et al.: Prevalent Eurasian avian-like H1N1 swine influenza virus with 2009 pandemic viral genes facilitating human infection. In: PNAS. Online-Vorabveröffentlichung vom 29. Juni 2020, doi:10.1073/pnas.1921186117.
- Swine flu strain with human pandemic potential increasingly found in pigs in China. Auf: sciencemag.org vom 29. Juni 2020.
- R. J. Garten, C. T. Davis et al.: Antigenic and Genetic Characteristics of Swine-Origin 2009 A(H1N1) Influenza Viruses Circulating in Humans. In: Science. 10. Juli 2009, Band 325, Nr. 5937, S. 197–201, doi:10.1126/science.1176225.