Reassortment
Unter Reassortment oder Reassortierung versteht man in der Virologie die Vermischung oder Neuverteilung genetischer Information zwischen zwei ähnlichen Viren. Meist handelt es sich um Varianten oder Subtypen einer Virusspezies oder um nahe verwandte Spezies innerhalb einer Virusgattung. Ein Reassortment ist unter natürlichen Bedingungen nur möglich, wenn:
- die beiden Virustypen sich in derselben infizierten Zelle vermehren und
- ihr Genom aus mehreren Segmenten besteht.
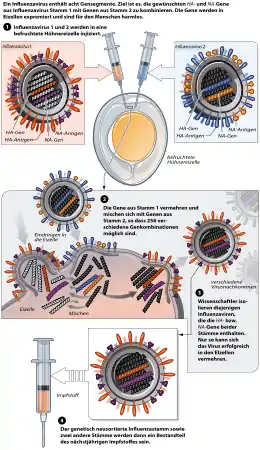
Die erste Bedingung kann nur erfüllt werden, wenn beide Viren auch den gleichen Wirt infizieren können, da sich nur dann ihre Genome in einer Zelle befinden können. Viren, die nicht auf einen einzigen Wirt festgelegt sind, also eine niedrige Wirtspezifität haben oder sich häufig durch Varianten an einen neuen Wirt anpassen können, reassortieren häufiger, weil ihnen mehr Wirte und damit mehr Viren zum Reassortment zur Verfügung stehen.
Die zweite Bedingung ist nur bei einigen Virusfamilien erfüllt, die durch ein segmentiertes Genom charakterisiert sind.
Man unterscheidet zwischen
- dem Reassortment bei Viren mit segmentierten Genom (wie bei den Influenzaviren), bei der die einzelnen Nukleinsäurestränge unverändert bleiben, und
- der homologen Rekombination innerhalb eines solchen Segments, wie es auch bei Viren mit unsegmentierten Genom möglich ist (wie bei den Coronaviren).[1]
Die erste Möglichkeit entspricht bei Eukaryoten (inklusive des Menschen) der Meiose (Reifeteilung) und späterer sexueller Verschmelzung der Zellkerne der mütterlichen und väterlichen Gameten, die zweite dem Crossing-over.
Ergebnis eines Reassortments
.jpg.webp)
Das Ergebnis eines Reassortments ist das plötzliche Auftauchen einer genetisch sehr abweichenden Variante, die Segmente von beiden vermischten Virusgenomen enthält. Wenn das Reassortment die Segmente für die Oberflächenproteine oder (bei unbehüllten Viren) die Kapsidproteine eines Virus durchmischt hat, so resultiert auch eine plötzliche Veränderung der Epitope auf der Virusoberfläche. Dieses Phänomen als Resultat des Reassortments nennt man Antigenshift (englisch antigenic shift).
Reassortments sind wegen der notwendigen gleichzeitigen Infektion eines Wirtes mit zwei Viren in natürlichen Biotopen eher selten. Auch ist das Ergebnis eines Reassortments in den meisten Fällen Viren, die nicht oder nur eingeschränkt vermehrungsfähig sind, oder Nachkommenviren produzieren, die mit ihren neuen Oberflächenproteinen die Zielzelle nicht mehr erkennen und somit nicht mehr infizieren können. Die Wahrscheinlichkeit für ein Reassortment steigt dann signifikant an, wenn ein oder zwei Populationen (z. B. Menschen und Schweine oder Hühnervögel) mit verschiedenen Virusvarianten in großer Zahl und Dichte die Möglichkeit zur gegenseitigen Infektion haben.
Bei einer Reassortierung finden schlagartig größere Änderungen der Sequenz der viralen Erbinformation und somit auch der daraus codierten viralen Proteine statt. Dadurch werden diese neuen Varianten der viralen Proteine nicht so gut von Gedächtniszellen des Immunsystems erkannt, und es tritt eine Immunevasion auf, die eine Herdenimmunität unterlaufen kann und somit eine neue Epidemie ermöglichen kann.
Im Labor kann ein Reassortment verhindert werden, indem die Verpackungssignale von mindestens zwei Segmenten vertauscht werden.[2]
Reassortment bei vertebralen Viren
Reassortment bei Viren, die Wirbeltiere (Vertebrata) infizieren können, gibt es ausschließlich bei RNA-Viren, und zwar bei den folgenden Familien:
- Reoviridae (z. B. Gattung Rotavirus) mit 10–12 Segmenten
- Bunyaviridae (z. B. die Gattung Hantavirus) mit 3 Segmenten
- Arenaviridae (z. B. Lassa-Virus) mit 2 Segmenten
- Orthomyxoviridae (Gattung Influenzavirus A als bekanntestem Beispiel) mit 6 bis 8 Segmenten.
Reassortment bei nicht-vertebralen Viren
Neben den oben genannten Virusfamilien gibt es noch weitere Familien oder Gattungen mit segmentiertem Genom, die jedoch nur Bakterien, Pflanzen, Pilze oder Insekten (anstelle von Wirbeltieren, wiss. Vertebrata) infizieren. Auch bei diesen wurde ein Reassortment beschrieben. Diese sind:
- Gattung Nanovirus (Pflanzenviren, Fam. Nanoviridae): 6–9 Segmente ssDNA
- Familie Cystoviridae (Bakteriophagen): 3 Segmente dsRNA
- Familie Chrysoviridae (Pilzviren): 4 Segmente dsRNA
- Gattung Varicosavirus (Pflanzenviren, Negarnaviricota: Haploviricotina: Monjiviricetes: Mononegavirales: Fam. Rhabdoviridae): 2 Segmente ssRNA
- Gattung Tenuivirus (Pflanzen- und Insektenviren, Negarnaviricota: Haploviricotina: Ellioviricetes: Bunyavirales: Fam. Phenuiviridae): 3–4 Segmente ssRNA
- Gattungen Sadwavirus und Cheravirus (Pflanzenviren, Picornavirales: Fam. Secoviridae): 2 Segmente ssRNA
- Unterfamilie Comovirinae (Pflanzenviren, Picornavirales: Fam. Secoviridae): 2 Segmente ssRNA
- Gattungen Tobravirus und Furovirus (Pflanzenviren, Fam. Vigaviridae): 2 Segmente ssRNA
- Gattungen Hordeivirus[3] und Pomovirus (Pflanzenviren, Fam. Vigaviridae): 3 Segmente ssRNA
- Familie Bromoviridae (Pflanzenviren): 3 Segmente ssRNA
Literatur
- David M. Knipe, Peter M. Howley et al. (Hrsg.): Fields’ Virology. 4. Auflage. Philadelphia 2001, ISBN 0-7817-1832-5.
- J. R. Gentsch, A. R. Laird, B. Bielfelt, D. D. Griffin et al.: Serotype diversity and reassortment between human and animal rotavirus strains: implications for rotavirus vaccine programs. In: The Journal of Infectious Diseases. 192 Suppl 1, 1. September 2005, S. S146–S159 (Review)
- R. N. Charrel, J. J. Lemasson, M. Garbutt, R. Khelifa et al.: New insights into the evolutionary relationships between arenaviruses provided by comparative analysis of small and large segment sequences. In: Virology. 317(2), 20. Dezember 2003, S. 191–196.
- T. Yanase, T. Kato, M. Yamakawa, K. Takayoshi et al.: Genetic characterization of Batai virus indicates a genomic reassortment between orthobunyaviruses in nature. In: Archives of Virology. 151(11), November 2006, S. 2253–2260.
- M. I. Nelson, L. Simonsen, C. Viboud, M. A. Miller et al.: Stochastic Processes Are Key Determinants of Short-Term Evolution in Influenza A Virus. In: PLoS Pathog. 2(12), 1. Dezember 2006, S. e125. PMID 17140286
- B. Schweiger, L. Bruns, K. Meixenberger: Reassortment between human A(H3N2) viruses is an important evolutionary mechanism. In: Vaccine. 24(44–46), 10. November 2006, S. 6683–6690.
Siehe auch
Einzelnachweise
- Maciej F. Boni, Philippe Lemey, Xiaowei Jiang, Tommy Tsan-Yuk Lam, Blair W. Perry, Todd A. Castoe, Andrew Rambaut, David L. Robertson: Evolutionary origins of the SARS-CoV-2 sarbecovirus lineage responsible for the COVID-19 pandemic, in: Nature Microbiology vom 28. Juli 2020, doi:10.1038/s41564-020-0771-4, §Main
- Q. Gao, P. Palese: Rewiring the RNAs of influenza virus to prevent reassortment. In: Proceedings of the National Academy of Sciences. Band 106, Nummer 37, September 2009, S. 15891–15896, doi:10.1073/pnas.0908897106. PMID 19805230, PMC 2747214 (freier Volltext).
- SIB: Hordeivirus, auf: ViralZone