ChIP-Seq
Die ChIP-Seq (von engl. Chromatin ImmunoPrecipitation DNA-Sequencing) ist eine biochemische Methode zur Bestimmung von Protein-DNA-Interaktionen. Die ChIP-Seq ist eine Kombination aus einer Chromatin-Immunpräzipitation und der DNA-Sequenzierung im Hochdurchsatz.[1][2] DNA-Protein-Interaktionen kommen bei Bindungssequenzen für Transkriptionsfaktoren an Promotoren, Enhancer, Repressoren, Silencer, Isolatoren sowie bei Bindungssequenzen für die DNA-Replikation vor.[3]
Eigenschaften
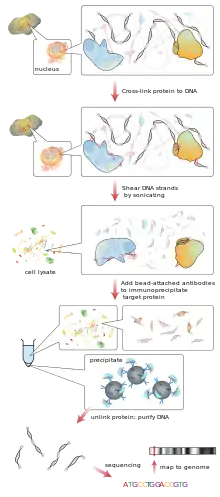
Das gereinigte rekombinante Protein wird mit der DNA vermischt oder in vivo gebunden, mit Formaldehyd reversibel quervernetzt. Anschließend erfolgt eine Fragmentierung per Ultraschall und eine Immunpräzipitation der vernetzten Protein-DNA-Komplexe mit einem Antikörper gegen das rekombinante Protein oder sein Protein-Tag. Abschließend erfolgt die thermische Freisetzung der DNA und eine DNA-Sequenzierung im Hochdurchsatz.
Eine Alternativmethode ist die ChIP-on-Chip, welche die Chromatin-Immunpräzipitation mit einer Hybridisierung mit einem DNA-Microarray (synonym DNA-Chip) verbindet. Durch eine parallele Verwendung beider Methoden können systematische Fehler der Methoden teilweise gemindert werden. Im Gegensatz zum ChIP-on-Chip erhöhen sich bei der ChIP-Seq die Kosten bei einer Erhöhung der Sensitivität, da hierbei viele Sequenziervorgänge erforderlich werden. Eine Variante der ChIP-Seq ist die Competition ChIP-Seq mit der untersucht wird, ob zwei Transkriptionsfaktoren um die Bindung an DNA-Sequenzen kompetieren.[4]
Verwandte Methoden ohne Immunpräzipitation sind die DNAse-Seq und die FAIRE-Seq (formaldehyde-assisted isolation of regulatory elements with high-throughput sequencing), welche entfaltete Regionen der DNA sequenzieren, an denen eine Regulation der Genexpression stattfindet.[5] Die ChIRP-Seq ist eine ChIP-Seq, bei der DNA-bindende RNA selektiv nachgewiesen werden kann.[6]
Die Kombination eines Nuclease Protection Assays mit einer ChIP-Seq wird als ChIP-Exo bezeichnet, bei der von Proteinen unbedeckte DNA-Bereiche durch eine Exonuklease aus dem Bakteriophagen λ verdaut werden, die übrigen dagegen sequenziert werden.[7]
Einzelnachweise
- T. S. Furey: ChIP-seq and beyond: new and improved methodologies to detect and characterize protein-DNA interactions. In: Nat Rev Genet. (2012), Band 13, Ausgabe 12, S. 840–852. doi:10.1038/nrg3306. PMID 23090257; PMC 3591838 (freier Volltext).
- C. S. Pareek, R. Smoczynski, A. Tretyn: Sequencing technologies and genome sequencing. In: J Appl Genet. (2011), Band 52, Ausgabe 4, S. 413–435. doi:10.1007/s13353-011-0057-x. PMID 21698376; PMC 3189340 (freier Volltext).
- M. J. Buck, J. D. Lieb: ChIP-chip: considerations for the design, analysis, and application of genome-wide chromatin immunoprecipitation experiments. In: Genomics (2004), Band 83, Ausgabe 3, S. 349–360. PMID 14986705.
- C. R. Lickwar, F. Mueller, J. D. Lieb: Genome-wide measurement of protein-DNA binding dynamics using competition ChIP. In: Nature protocols. Band 8, Nummer 7, 2013, S. 1337–1353, doi:10.1038/nprot.2013.077, PMID 23764940.
- L. Song, Z. Zhang, L. L. Grasfeder, A. P. Boyle, P. G. Giresi, B. K. Lee, N. C. Sheffield, S. Gräf, M. Huss, D. Keefe, Z. Liu, D. London, R. M. McDaniell, Y. Shibata, K. A. Showers, J. M. Simon, T. Vales, T. Wang, D. Winter, Z. Zhang, N. D. Clarke, E. Birney, V. R. Iyer, G. E. Crawford, J. D. Lieb, T. S. Furey: Open chromatin defined by DNaseI and FAIRE identifies regulatory elements that shape cell-type identity. In: Genome research. Band 21, Nummer 10, Oktober 2011, S. 1757–1767, doi:10.1101/gr.121541.111, PMID 21750106, PMC 3202292 (freier Volltext).
- Ci Chu, Qu, Kun, Zhong, Franklin L., Artandi, Steven E., Chang, Howard Y.: Genomic Maps of Long Noncoding RNA Occupancy Reveal Principles of RNA-Chromatin Interactions. In: Molecular Cell. 31. August 2011. doi:10.1016/j.molcel.2011.08.027.
- Shaun Mahony, B. Franklin Pugh: Protein–DNA binding in high-resolution. In: Critical Reviews in Biochemistry and Molecular Biology. 50, 2015, S. 269, doi:10.3109/10409238.2015.1051505.