Electrophoretic Mobility Shift Assay
Der Electrophoretic Mobility Shift Assay (EMSA) oder Band Shift Assay ist eine Affinitätselektrophorese und dient zum Nachweis von DNA- oder RNA-bindenden Proteinen, beispielsweise Transkriptionsfaktoren.[1][2]
Eigenschaften
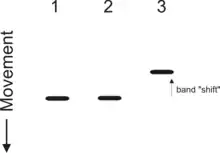
Zur Bestimmung von Protein-DNA-Interaktionen per EMSA werden Proteine mit einem DNA-Fragment bekannter Sequenz inkubiert, bei Protein-RNA-Interaktionen entsprechend mit einem RNA-Fragment. Bei der DNA-Sequenz handelt es sich meist um einen regulatorischen Bereich eines Gens (beispielsweise eines Promotors oder Enhancers). Die Probe wird auf ein Agarose- oder Polyacrylamid-Gel aufgetragen und mittels eines elektrischen Feldes werden die Komplexe aus Protein und DNA entsprechend ihrer Größe aufgetrennt.[3] Im Vergleich zur reinen DNA-Bande tritt bei der Gelelektrophorese eine Laufweitenverschiebung (engl. band shift) auf, abhängig von Ladung, Konformation und Größe des Proteinligandenkomplexes. Enthält die Affinitätselektrophorese neben der DNA und einem interagierenden Protein noch einen Antikörper gegen das Protein, wird der Assay als Supershift Assay bezeichnet. DNA-Protein-Antikörper-Komplexe wandern langsamer als DNA-Protein-Komplexe, welche langsamer als ungebundene DNA wandern. Nicht gebundene saure Proteine können in dem verwendeten Puffersystem im Gegensatz zu DNA- und RNA-bindenden Proteine, welche oftmals eine positive Nettoladung aufweisen, ebenfalls in das Gel einlaufen. Da die Proteine aber nicht markiert sind, werden sie nicht sichtbar ohne eine Proteinfärbung wie z. B. eine Silberfärbung. Durch Verdünnungsreihen lassen sich Affinitäten ermitteln.[4] Die Komplexe dissoziieren nicht in der Elektrophorese durch einen Käfig-Effekt der Poren im Gel.[5]
Durch Markierung der DNA können die Banden im Gel sichtbar gemacht werden, beispielsweise durch Molekülmarkierung mit einem Radionuklid, per Digoxygenin-Markierung, per Biotinylierung oder mittels einer Fluoreszenzmarkierung. Hierbei dient zur Spezifizierung der Banden nicht nur die DNA und der Antikörper, sondern es kann auch nicht-markierte DNA in unterschiedlicher Menge zum Blockieren aber auch mutierte DNA zum Herausfiltern eines spezifischen Signals eingesetzt werden. Der Einsatz von Punktmutanten in der markierten DNA oder in dem unmarkierten Kompetitor erlaubt Rückschlüsse auf für die Bindung wichtige Nukleotide.[6]
Der EMSA, der aufgrund seiner unkomplizierten Anwendung zu den gängigen Methoden bei der Aufklärung der Mechanismen der Genregulation gehört, basiert auf den Arbeiten von Garner und Revzin[1] und von Fried und Crothers.[2]
Eine Abwandlung ist der Methylierungs- und Uracil-Interferenzassay, bei dem der Einfluss modifizierter Basen auf die DNA-Bindung untersucht wird. Er erlaubt Rückschlüsse auf Nukleotide, die in die Protein-DNA-Interaktion involviert sind.[7]
Literatur
- Tom Moss (Hrsg.): DNA'Protein Interactions: Principles and Protocols (Methods in Molecular Biology). 2. Auflage. Humana press, 2001, ISBN 0-89603-671-5.
Weblinks
- Dr. Mirmira EMSA Protocol (englisch)
- Preparation of nuclear extract for EMSA (englisch)
- EMSA for Transcription Factor Binding (englisch)
- Chemiluminescent Gel Shift Protocol (englisch)
Einzelnachweise
- M. M. Garner, A. Revzin: A gel electrophoresis method for quantifying the binding of proteins to specific DNA regions: application to components of the Escherichia coli lactose operon regulatory system. In: Nucleic Acids Research. Band 9, Nr. 13, Juli 1981, S. 3047–3060, doi:10.1093/nar/9.13.3047, PMID 6269071, PMC 327330 (freier Volltext).
- M. Fried, D. M. Crothers: Equilibria and kinetics of lac repressor-operator interactions by polyacrylamide gel electrophoresis. In: Nucleic Acids Research. Band 9, Nr. 23, Dezember 1981, S. 6505–6525, doi:10.1093/nar/9.23.6505, PMID 6275366, PMC 327619 (freier Volltext).
- Frederick M. Ausubel: Current Protocols in molecular biology. John Wiley & Sons, Chichester 1994, ISBN 0-471-50337-1, S. 12.2.1–11.
- M. G. Fried: Measurement of protein-DNA interaction parameters by electrophoresis mobility shift assay. In: Electrophoresis. Band 10, 1989, S. 366–376. PMID 2670548.
- M. G. Fried, G. Liu: Molecular sequestration stabilizes CAP-DNA complexes during polyacrylamide gel electrophoresis. In: Nucleic Acids Research. Band 22, Nr. 23, 1994, S. 5054–5059. doi:10.1093/nar/22.23.5054. PMID 7800499.
- Jiří Kozelka: Evaluation of dissociation constants from competition binding experiments based on the relative binding ratio. In: Analytical Biochemistry. Band 409, Nr. 1, 2011, ISSN 0003-2697, S. 66–73, doi:10.1016/j.ab.2010.09.023.
- Albert S. Baldwin, Marjorie Oettinger, Kevin Struhl: Methylation and Uracil Interference Assays for Analysis of Protein-DNA Interactions. In: Current Protocols in Molecular Biology. Band 12, 2001, doi:10.1002/0471142727.mb1203s36.