Strukturaufklärung
Strukturaufklärung, auch Strukturanalyse oder Strukturanalytik, bezeichnet die Aufklärung der Zusammensetzung chemischer Verbindungen.[1] Chemische Stoffe können sich hinsichtlich der räumlichen Anordnung der einzelnen Atome in ihren Molekülen unterscheiden, auch wenn ihre atomare Zusammensetzung gleich ist (Isomerie). Die Strukturanalytik umfasst als Teilgebiet der Analytischen Chemie chemische und physikalische Methoden zur Aufklärung der chemischen Struktur von Substanzen.
Auch die Strukturchemie, Festkörperchemie, Festkörperphysik und Kristallographie, sowie Werkstoffprüfung und Metallografie beschäftigen sich mit der Aufklärung und Beschreibung von Strukturen, der räumlichen Anordnung von Atomen, Molekülen und Ionen in Feststoffen. In der Oberflächenchemie und -physik ist die Struktur einer Oberfläche von besonderer Bedeutung, da an einem Phasenübergang oft besondere Effekte entstehen.
Chemische Methoden
Einige funktionelle Gruppen in organischen Molekülen lassen sich mit einfachen chemischen Nachweisreaktionen finden:
- Alkohole: Iodoformprobe
- Aldehyde: Fehlingsche Probe, Schiff's Reagenz, Tollensprobe, Nylanders Reagenz
- Carbonsäuren: Esterbildung mit Geruchsprobe
- C–C-Mehrfachbindungen: Elektrophile Addition von Brom oder Iod (Entfärbung)
- Organische Halogenide: Beilsteinprobe
Die einzelnen Ergebnisse dieser Reaktionen sind häufig nicht als endgültiger Nachweis zu verstehen, da manche der Proben nicht vollkommen spezifisch sind oder in Gegenwart bestimmter anderer funktioneller Gruppen versagen. Meist bringt erst die Kombination mehrerer Prüfmethoden Gewissheit über die Struktur der untersuchten Verbindung.
Instrumentelle Methoden
„Kleine Moleküle“
Durch die Elementaranalyse lässt sich die Zusammensetzung, das heißt der Anteil von Atomen der einzelnen Elemente an einem Molekül, an einer chemischen Verbindung feststellen. Das reicht bei organischen Molekülen jedoch meist noch nicht aus, um eine Strukturformel des Moleküls zeichnen zu können. Um zusätzliche Informationen über die Topologie des Moleküls zu erhalten, stehen eine Reihe spektroskopischer Methoden zur Verfügung.
Dazu gehören:
- NMR-Spektroskopie: kann an verschiedenen Atomkernen (am häufigsten 1H, 13C, 19F, 31P) durchgeführt werden und liefert Information über die Umgebung dieser Kerne: a) über die Art der daran gebundenen Atome durch deren chemische Verschiebung (also die Frequenzänderung des Signals relativ zu einer Standardverbindung) und b) über die Anzahl sich in der Umgebung befindlicher anderer Atome über die Kopplung (was zu charakteristischen Aufspaltungsmustern der Signale führt)
- Massenspektrometrie: Zeigt die Gesamtmasse des Moleküls und abhängig von der eingesetzten Technik die Masse von Fragmenten, in die ein Molekül in charakteristischen Zerfallswegen während der Massenspektrometrie zerfällt.
- Infrarot-Spektroskopie: lässt Rückschlüsse über die Existenz bestimmter funktioneller Gruppen in Molekülen zu und gibt – in einfacheren Fällen – Auskunft über deren Symmetrie
- Einkristall-Röntgenstruktur-Analyse: führt zu einem dreidimensionalen Modell aller schwereren Atome (Wasserstoffatome werden nur sehr schlecht abgebildet), Voraussetzung ist das Vorliegen eines gut ausgebildeten Einkristalls der Substanz. Die Ergebnisse sind Bindungslängen und -winkel in Präzisionen typischerweise bis 0.001 Å bzw. 0.1°.
- Gasphasen-Elektronenbeugung[2] ermöglicht die Strukturbestimmung freier Moleküle, d. h. ohne die Verzerrungen, die sie durch den Einbau in einen Kristall, wie bei der Einkristall-Röntgenbeugung, erfahren. Die Methode erfordert aber die Verdampfbarkeit der Verbindung ohne Zersetzung. Die Ergebnisse sind Bindungslängen und -winkel in Präzisionen typischerweise bis 0.001 Å bzw. 0.1° für sehr kleine Systeme, weniger präzise für größere.
- Mikrowellenspektroskopie (oder Rotationsspektroskopie) ermöglicht ebenfalls die Strukturbestimmung freier Moleküle in gasförmigen Zustand, ist aber typischerweise auf recht kleine Systeme begrenzt. Es können für sehr kleine Moleküle sehr hohe Präzisionen erreicht werden.
Gelegentlich ist es nötig, die Stereochemie einer neu synthetisierten chiralen Substanz zu bestimmen. Für diese Aufgabe kommen von den oben genannten Spektroskopiemethoden nur die Röntgenstruktur-Analyse und in einigen Fällen die NMR-Spektroskopie in Frage. Bevor diese Techniken bekannt waren, konnte nur eine relative Stereochemie durch Zurückführen der noch nicht charakterisierten Substanz mittels chemischer Reaktionen auf bereits charakterisierte Substanzen bestimmt werden, dies hatte vor allem für die Konfiguration der Zucker eine große Bedeutung.
Biologische Makromoleküle
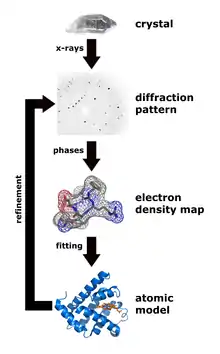
Die Strukturaufklärung von Proteinen und DNA heutzutage unterscheidet sich von der kleinerer Moleküle, da die Primärstruktur, d. h. die Verknüpfung der einzelnen Atome, bereits bekannt ist. Das Interesse gilt hier im Allgemeinen der Faltung (auch Proteinstruktur), d. h. der genauen räumlichen Anordnung der Atome im Molekül.
Von den oben genannten Techniken werden für die Aufklärung der räumlichen Struktur von Biomakromolekülen nur die Röntgen-Strukturanalyse und die NMR-Spektroskopie eingesetzt.
Für die Röntgenstrukturanalyse ist es nötig, Einkristalle der Biomakromoleküle in ausreichender Größe zu erhalten. Dies ist oft nur mittels vieler unterschiedlicher Kristallationsversuche möglich, oft werden auch gar keine Kristalle erhalten (zum Beispiel weil das Protein flexible Bereiche aufweist). Der Erhalt von Kristallen kann so Monate bis Jahre in Anspruch nehmen. Sind die Kristalle vorhanden, lassen sich jedoch die entsprechenden Strukturen anhand der aufgenommenen Beugungsmuster normalerweise innerhalb von Tagen oder Wochen erhalten.
Die Strukturaufklärung mittels NMR-Spektroskopie analysiert die Biomakromoleküle direkt in Lösung, meist ihrer natürlichen Umgebung. Dies unterscheidet diese Strukturen von den mittels Röntgenbeugung erhaltenen, denn durch die Einbettung in ein Kristallgitter und die dadurch zusätzlich auf das Molekül einwirkenden Kräfte können diese Strukturen verändert sein. Durch NMR zugänglich sind jedoch nur Atome mit einem magnetischen Moment (einer ungeraden Spinquantenzahl) des Atomkerns. Insbesondere ist dies Wasserstoff und das natürlich in Kohlenstoff zu 1 % neben 12C vorkommende 13C und Phosphor 31P (in DNA und RNA). Um mehr Informationen, auch über andere Atomarten, erhalten zu können, müssen Moleküle verwendet werden, in denen NMR-taugliche Isotope wie 13C oder 15N angereichert wurden.
Die Analyse von zwei oder dreidimensionalen NMR-Spektren kann die folgenden Informationen über die Substanz liefern:
- Abstände zwischen zwei Wasserstoff-Atomkernen (Protonen) durch den NOE (Kern-Overhauser-Effekt, NOESY-Spektren)
- Orientierung von 15N-H Bindungen durch dipolare Restkopplungen
- Diederwinkel durch Bestimmung der skalaren Kopplung in eindimensionalen oder COSY-Spektren
Proteine
Die Strukturaufklärung ist bei Proteinen von Interesse, da nur bei einer von mehreren möglichen Faltungen das Protein in der Lage ist, als ein Enzym zu wirken.
DNA und RNA
Die erste Strukturaufklärung von DNA geht auf Röntgenstrukturaufklärung durch Rosalind Franklin zurück. Ihre Röntgenbeugungsdiagramme lieferten die wesentlichen Hinweise auf die Struktur der DNA, welche im Jahre 1953 von James Watson und Francis Crick veröffentlicht wurde.
Die erste hochaufgelöste Struktur eines DNA-Duplex in B-Konformation, das sogenannte Dickerson-Dodecamer, wurde im Jahre 1981 von Drew, Dickerson et al. veröffentlicht. Die Koordinaten dieses Dodecamers sind in der Brookhaven Protein Data Bank unter dem Kürzel 1BNA zugänglich. Es gilt als ein Prototyp für die Struktur von "normaler" DNA in B-Konformation und wurde inzwischen in zahlreichen weiteren Studien verfeinert oder als Referenz verwendet.
Bei der Strukturaufklärung von DNA heute ist oft die Art der Anlagerung von DNA an ein Protein oder eines organischen Moleküls (zum Beispiel eines Arzneimittels) an die DNA von Interesse. Dies gilt insbesondere für chemisch modifizierte DNA, die in Forschung und Analytik eingesetzt wird. Zudem kann DNA Triplexe, Quatruplexe und Haarnadelstrukturen ausbilden.
Die strukturelle Vielfalt von RNA ist generell größer als die von DNA. Das bedeutet, dass RNA in größerem Umfang als DNA komplexe Strukturen ausbildet, wie zum Beispiel in t-RNA oder snRNA.
Einzelnachweise
- Rankin, David W. H.: Structural methods in molecular inorganic chemistry. John Wiley & Sons, 2013, ISBN 978-0-470-97279-3.
- Hargittai, István Hargittai, Magdolna: Stereochemical applications of gas-phase electron diffraction. VCH, 1988, ISBN 0-89573-292-0.