Sputnik-Virus
Das Sputnik-Virus (auch Sputnik-Virophage, wissenschaftlich Mimivirus-dependent virus Sputnik), ist eine Spezies von Viren der Familie Lavidaviridae, das sich in der Amöbe Acanthamoeba castellanii nur in Gegenwart eines weiteren Virus aus der Gattung Mimivirus vermehren kann. Es wurde 2008 als begleitendes Virus entdeckt und für ihn als möglichen Vertreter einer mutmaßlich neuen Gruppe von Erregern, die Bezeichnung „Virophage“ vorgeschlagen. Tatsächlich vermehrt sich das Sputnik-Virus jedoch nicht im Virion (Viruspartikel) des Mimivirus, sondern nutzt den durch das Mimivirus umgestalteten Proteinsyntheseapparat der Zelle (Viroplasma) und ist von den Replikationsenzymen des Mimivirus abhängig. Damit ist es in seinem Vermehrungsverhalten dem Hepatitis-D-Virus (Virusoid) und den Adeno-assoziierten Viren (Gattung Dependovirus) bei tierischen Viren und den Satellitenviren bei einigen Pflanzenviren sehr ähnlich. Ein verwandter Virophage namens Zamilon (wissenschaftlich Mimivirus-dependent virus Zamilon, gleiche Virusgattung) befällt einen anderen „Mamavirus“ getauften Vertreter der Mimiviridae.[2]
| Sputnik-Virus | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|

Sputnik-Virophage | ||||||||||||||||||
| Systematik | ||||||||||||||||||
| ||||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||||
| ||||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||||
| Mimivirus-dependent virus Sputnik | ||||||||||||||||||
| Links | ||||||||||||||||||
|
Genom
Das Genom des Sputnik-Virus besteht aus einer doppelsträngigen DNA, die ringförmig geschlossen ist. Sie ist 18.343 kb groß und zeichnet sich ähnlich den Mimiviren durch einen sehr geringen GC-Gehalt von 27 % aus. Das Genom enthält 21 mögliche Offene Leserahmen (ORFs), die sich zum Teil überlappen. Durch Vergleich der aus ihnen ableitbaren Proteinsequenzen mit bekannten Proteinen, konnte bei einigen Genprodukten eine wahrscheinliche, homologe Sekundärstruktur und damit eine mögliche Funktion abgeleitet werden. Im Viruspartikel (Virion) des Sputnik-Virus sind drei Proteine mittels Polyacrylamid-Gelelektrophorese als Strukturproteine identifiziert, die nach MALDI-TOF-Daten den ORFs 8, 19 und 20 zugeordnet werden konnten. Die Proteinsequenzen aus ORF 6 und 12 haben eine gewisse Ähnlichkeit mit Proteinen des Acanthamoeba-polyphaga-Mimivirus APMV (MIMI R196 und MIMI R546). Die Gensequenzen des Sputnik-Virus setzen sich somit neben bislang unbekannten viralen Sequenzen aus zwei weiteren Anteilen zusammen, die einerseits dem Mimivirus-Genom und andererseits bekannten Sequenzen aus Viren ähneln, die Archaeen oder Bakterien zum Wirt haben. Während der Replikation der Sputnik-Virus-DNA kann es zu Genaustauschen mit dem APMV kommen.
| ORF | Aminosäuren | mögliche Funktion | Protein-Homologe |
|---|---|---|---|
| ORF 1 | 144 | unbekannt | unbekannt |
| ORF 2 | 114 | unbekannt | unbekannt |
| ORF 3 | 245 | DNA-Verpackung | RecA-Superfamilie ATPasen (54 %) |
| ORF 4 | 139 | Transkriptions-Regulation? | Zinkfingerproteine |
| ORF 5 | 119 | unbekannt | unbekannt |
| ORF 6 | 310 | Protein-Protein-Interaktion im Viroplasma? | Tripel-Helix-Proteine, MIMI R196 (53 %) |
| ORF 7 | 236 | Protein-Protein-Interaktion im Viroplasma? | Tripel-Helix-Proteine, TNF-assoziiertes Protein 5 (27 %) |
| ORF 8 | 184 | Strukturprotein, Kapsid | unbekannt |
| ORF 9 | 175 | unbekannt | unbekannt |
| ORF 10 | 226 | DNA-Integrase | Integrase-Familie bei Bakteriophagen (27 %), Tyr-Rekombinase |
| ORF 11 | 162 | unbekannt | unbekannt |
| ORF 12 | 152 | unbekannt | MIMI R546 (64 %) |
| ORF 13 | 779 | DNA-Replikation (virale DNA-Polymerase) | DNA-Primase, Helikase |
| ORF 14 | 114 | Transkriptions-Regulation? | Zinkfingerproteine |
| ORF 15 | 109 | Membranprotein? | keine |
| ORF 16 | 130 | unbekannt | unbekannt |
| ORF 17 | 88 | DNA-Bindeprotein | IS3-Superfamilie Transposasen |
| ORF 18 | 167 | unbekannt | unbekannt |
| ORF 19 | 218 | Strukturproteine, Kapsid | unbekannt |
| ORF 20 | 595 | Strukturproteine, Haupt-Kapsidprotein | unbekannt |
| ORF 21 | 438 | unbekannt | unbekannt |
Interessanterweise wurde 2018 eine Gen-Homologe von Sputnik und Zamilon mit dem Orpheovirus gefunden.[4]
Morphologie
Die Virionen des Sputnik-Virus sind etwa 50 nm im Durchmesser groß und bestehen aus einem unbehüllten, ikosaedrischen Kapsid. Das Kapsid wird wahrscheinlich ausschließlich von einem 595 Aminosäuren großen Kapsidprotein gebildet (ORF 20); zwei weitere Strukturproteine (ORF 8 und 19) finden sich nur jeweils einmal im Virion. In infizierten Amöben findet man dicht gedrängte Aggregate von Sputnik-Kapsiden im Bereich des Viroplasmas. Da das Virus die Verpackung und Morphogenese des APMV beeinträchtigt, können mitverpackte Sputnik-Viren im Inneren von deformierten, defekten APMV-Virionen gefunden werden.
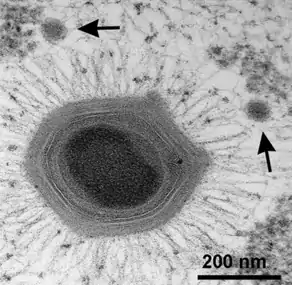
_Sputnik%2540Mimivirus.png.webp)
_Sputnik.png.webp)
_Sputnik.png.webp) TEM-Aufnahme neugebildeter reifer Sputnik-Virionen.[6]
TEM-Aufnahme neugebildeter reifer Sputnik-Virionen.[6]_Sputnik_progeny.png.webp)
Biologische Bedeutung
Das Sputnik-Virus ist ein erstes Beispiel für eine abhängige Vermehrung und einen genetischen Austausch zwischen zwei marinen Virusarten. Dies war bisher nur bei Pflanzenviren, Bakteriophagen und animalen Viren (Virusoiden) beschrieben worden. Obwohl bekannt ist, dass Viren (insbesondere Bakteriophagen) als Femto- bzw. Virioplankton die größte Biomasse und wahrscheinlich die artenreichste Gruppe in den Ozeanen und anderen Gewässern darstellen[7], sind sie in ihrem ökologischen und genetischen Zusammenhang nur wenig erforscht. Das neu entdeckte Mimivirus und ihm ähnliche genetische Elemente sind in marinen Ökosystemen regelmäßig nachweisbar.[8] Da die taxonomische und phylogenetische Stellung des Mimivirus selbst noch unklar ist, gilt dies auch für das Verhältnis von Mimivirus zu Sputnik-Virus. Große Genabschnitte des Mimivirus sind bakteriellen Genen (Plasmid-ähnlich) und eukaryotischen Genen der parasitierten Alge sehr ähnlich, was auf einem horizontalen Gentransfer zwischen Virus und Wirt sowie auf einen möglichen bakteriellen Ursprung der Evolution des Mimivirus beruhen könnte.[9] Davon ausgehend kann postuliert werden, dass das Sputnik-Virus ein ursprünglicher Bakteriophage jenes Ursprungsbakteriums darstellen würde, aus dem sich das Mimivirus durch Verlust eines eigenen Syntheseapparates entwickelte.
Systematik
Neben Sputnik 1 weitere (vorgeschlagene) Vertreter dieser Gattung gefunden. Damit:
- Gattung Sputnikvirus
- Spezies Sputnik-Virus 1 (offiziell Mimivirus-dependent virus Sputtnik) – infiziert „Mamavirus“ aus der Mimivirus-Gruppe A.
- Spezies „Sputnik-Virus 2“ – gefunden 2012, infiziert „Lentille virus“, ebenfalls aus der Mimivirus-Gruppe A.[2][10]
_Sputnik3_InCulture.tif.jpg.webp)
_Sputnik3%2540MimivirusGroupA_(ContrastEnh).tif.jpg.webp) Nachkommenschaft des „Sputnik 3“ Virophagen, produziert in einer Virusfabrik (VF) von Gruppe-A-Mimiviren. Balken 1 µm.[3]
Nachkommenschaft des „Sputnik 3“ Virophagen, produziert in einer Virusfabrik (VF) von Gruppe-A-Mimiviren. Balken 1 µm.[3]_Sputnik3%2540Moumouvirus_(ContrastEnh).tif.jpg.webp) „Sputnik 3“-Virophagen, produziert in eriner VF von Moumouvirus (Gruppe-B-Mimiviren). Balken 200 nm.[3]
„Sputnik 3“-Virophagen, produziert in eriner VF von Moumouvirus (Gruppe-B-Mimiviren). Balken 200 nm.[3]_Sputnik3%2540Courdo11_(ContrastEnh).tif.jpg.webp) „Sputnik 3“-Virophagen, produziert in eriner VF von Courdo11 (Spezies Megavirus chilensis, Gruppe C-Mimiviren). Balken 2 µm.[3]
„Sputnik 3“-Virophagen, produziert in eriner VF von Courdo11 (Spezies Megavirus chilensis, Gruppe C-Mimiviren). Balken 2 µm.[3]
- Spezies „Sputnik-Virus 3“ – gefunden 2013, kann das Mimivirus (Typusspezies APMV) infizieren, das aber nicht der natürliche Wirt ist.[3][2][11]
- Spezies Zamilon-Virus (offiziell Mimivirus-dependent virus Zamilon) mit Zamilon 1 (2013, Tunesien) und Zamilon 2 (2015, Nordamerika)
Literatur
- B. La Scola et al.: The virophage as a unique parasite of the giant mimivirus. In: Nature, 2008, 455(7209), S. 100–104; PMID 18690211
- H. Ogata H, J. M. Claverie: Microbiology. How to infect a Mimivirus. In: Science, 2008 Sep 5, 321(5894), S. 1305–1306; PMID 18772426
Weblinks
- Referenzsequenz EU606015 des Sputnik-Virus
- Pressemitteilung des CNRS vom 6. August 2008 (französisch) mit der Abbildung eines im Mimivirus-Kapsid verpackten Sputnik-Virus
- Expasy: ViralZone: Sputnikvirus
- Andreas Jahn: Das Virus-Virus. Spektrum.de, 6. August 2008
Einzelnachweise
- ICTV: ICTV Master Species List 2019.v1, New MSL including all taxa updates since the 2018b release, March 2020 (MSL #35)
- Morgan Gaia et al.: Zamilon, a Novel Virophage with Mimiviridae Host Specificity. In: PLoS One, Band 9, Nr. 4, 2014, S. e94923; Epub 18. April 2014, doi:10.1371/journal.pone.0094923, PMC 3991649 (freier Volltext)
- Morgan Gaia, Isabelle Pagnier, Angélique Campocasso, Ghislain Fournous, Didier Raoult, Bernard La Scola: Broad spectrum of mimiviridae virophage allows its isolation using a mimivirus reporter. In: PLoS One, 8, 2013, S. e61912; doi:10.1371/journal.pone.0061912 PMC 3626643 (freier Volltext)
- Julien Andreani, Jacques Y. B. Khalil, Emeline Baptiste, Issam Hasni, Caroline Michelle, Didier Raoult, Anthony Levasseur, Bernard La Scola: Orpheovirus IHUMI-LCC2: A New Virus among the Giant Viruses, in: Front. Microbiol., 22. Januar 2018, doi:10.3389/fmicb.2017.02643
- S. Duponchel, M. G. Fischer: Viva lavidaviruses! Five features of virophages that parasitize giant DNA viruses. In: PLoS pathogens, 15(3), 2019. doi:10.1371/journal.ppat.1007592.
- Said Mougari, Dehia Sahmi-Bounsiar, Anthony Levasseur, Philippe Colson, Bernard La Scola: Virophages of Giant Viruses: An Update at Eleven. In: Viruses, Band 11, Nr. 8, Reihe Viruses of Plants, Fungi and Protozoa , 8. August 2019, S. 733, doi:10.3390/v11080733.
- K. E. Wommack, R. R. Colwell: Virioplankton: viruses in aquatic ecosystems. In: Microbiology and Molecular Biology Reviews, 2000, 64,1, S. 69–114; PMID 10704475, PMC 98987 (freier Volltext)
- A. Monier, J. M. Claverie, H. Ogata: Taxonomic distribution of large DNA viruses in the sea. In: Genome Biol., 2008, 9(7), S. R106; PMID 18598358
- D. Moreira, C. Brochier-Armanet: Giant viruses, giant chimeras: the multiple evolutionary histories of Mimivirus genes. In: BMC Evol Biol., 2008, 8, S. 12; PMID 18205905
- Ed Yong: A Parasite’s Parasites, in: The Scientist, 15. Oktober 2012
- Clara Rolland, Julien Andreani, Amina Cherif Louazani, Sarah Aherfi, Rania Francis, Rodrigo Rodrigues, Ludmila Santos Silva, Dehia Sahmi, Said Mougari, Nisrine Chelkha, Meriem Bekliz, Lorena Silva, Felipe Assis, Fábio Dornas, Jacques Yaacoub Bou Khalil, Isabelle Pagnier, Christelle Desnues, Anthony Levasseur, Philippe Colson, Jônatas Abrahão, Bernard La Scola: Discovery and Further Studies on Giant Viruses at the IHU Mediterranee Infection That Modified the Perception of the Virosphere. In: Viruses, 11(4), März/April 2019, pii: E312, doi:10.3390/v11040312, PMC 6520786 (freier Volltext), PMID 30935049
_Sputnik3_Genome_Map.tif.jpg.webp)
_Sputnik3_TEM(-).tif.jpg.webp)