Zamilon-Virus
Das Zamilon-Virus (auch Zamilon-Virophage, wissenschaftlich Mimivirus-dependent virus Zamilon) ist ein kleines DNA-Virus, das Protisten infiziert und zur eigenen Replikation ein Helfervirus benötigt. Bei dem 2013 in Tunesien entdeckten Zamilon handelt es sich also um eine Art Satellitenvirus, oft auch als Virophage klassifiziert.[2] Zamilon infiziert die Amöben der Gattung Acanthamoeba polyphaga. Er ähnelt sehr dem ersten entdeckten Virophagen, dem verwandten Sputnik-Virus (gleiche Virengattung).
| Zamilon-Virus | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
.jpg.webp)
Elektronenmikroskopische Aufnahme einer Virusfabrik in einer Amöbe, | ||||||||||||||||||
| Systematik | ||||||||||||||||||
| ||||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||||
| ||||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||||
| Mimivirus-dependent virus Zamilon | ||||||||||||||||||
| Links | ||||||||||||||||||
|
Der Name Zamilon kommt aus dem Arabischen (arabisch زميل zamil, deutsch ‚Kollege, Nachbar‘, englisch colleague, coworker).[3]
Alle bekannten Virophagen sind assoziiert mit Helferviren der Riesenviren-Familie der Mimiviridae. Zamilon ist in seiner Auswahl an Hilfsviren eingeschränkt: Es ist innerhalb der Mimiviridae auf die Mimivirus-ähnlichen Linien I B und I C angewiesen, Viren der Linie I A sind für Zamilon ohne Nutzen. Dies scheint an einem rudimentären Immunsystem (genannt MIMIVIRE, englisch mimivirus virophage resistance element) der bezeichneten Helferviren der Linie I A zu liegen, das dem CRISPR-Cas-System ähnelt.[4][5] Siehe dazu auch Mougari et al. (2019), Fig. 5.[6]
Im Gegensatz zum Sputnik-Virophagen scheint Zamilon die Replikation seines Helfervirus nicht zu beeinträchtigen.
Entdeckung und verwandte Virophagen
.jpg.webp)
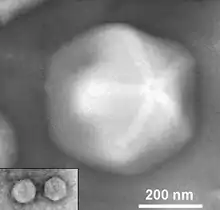
Zamilon wurde 2013 in der Amöbe Acanthamoeba polyphaga entdeckt, die mit dem aus einer tunesischen Bodenprobe isolierten Riesenvirus „Mont1“ (Gattung Mimivirus, Linie C) koinfiziert war.[2][3][7]
Mit Stand 2015 ist Zamilon einer von drei Virophagen-Spezies, die physikalisch isoliert wurden (die beiden anderen sind das Sputnik-Virus aus derselben Gattung und das Mavirus, eine Schwestergattung innerhalb der Familie Lavidaviridae).
Mehrere andere Virophagen-DNAs wurden mithilfe von Metagenomanalyse gefunden, jedoch bis dato nicht physikalisch isoliert.[2][8] Ein verwandter Stamm innerhalb der Zamilon-Spezies namens Zamilon 2 wurde 2015 durch Metagenomanalyse eines nordamerikanischen Pappelholzschnitzel-Bioreaktors entdeckt.[9] Ein anderer Virophage, Rio Negro, ist ebenfalls eng mit Sputnik verwandt.[2] Eine weitere verwandte Spezies(?) ist „Zamilon vitis“, das die Spezies „Megavirus vitis“ parasitiert.[10]
Systematik
Der Zamilon-Virophage wurde vom International Committee on Taxonomy of Viruses (ICTV) in die Spezies Mimivirus-dependent virus Zamilon der Gattung Sputnikvirus in der Familie Lavidaviridae eingeteilt.[2]
Aufbau
Die Zamilon-Viruspartikel (Virionen) sind kugelförmig (sphärisch) mit einem Durchmesser von 50–60 nm und ähneln im Aussehen denen von Sputnik und Mavirus.[3] Das zirkuläre doppelsträngige DNA-Genom ist 17.276 bp lang.[2][3]
Virophagen haben typischerweise Virionen mit einem Durchmesser im Bereich von 40–80 nm und eine Genomgröße im Bereich von 17–30 kbp.[2] Der mit Zamilon am engsten verwandte Virophage ist Sputnik, mit dem er 76 % der Genomsequenz teilt, obwohl ein Teil der Zamilon-Sequenz im Vergleich zu Sputnik umgekehrt (revers) ist.[2][3] Zamilons DNA ist reich an den Basen Adenin und Thymin, der GC-Anteil beträgt nur 29,7 %.[3]
Die Analysen ergaben, dass das Zamilon-Genom 20 offene Leserahmen (englisch openreading frames, ORFs) mit einer Länge zwischen 222 Basen und 2337 Basen enthalten sollte; es sollten also 20 Polypeptide kodiert werden. Von den 20 vorhergesagten Genprodukten ähneln 15 denen von Sputnik, drei ähneln denen des Acanthamoeba polyphaga Mimivirus (APMV alias Mimivirus sensu stricto: Gattung Mimivirus Linie A), zwei dem „Megavirus chilensis“ (Gattung Mimivirus Linie C) und eines dem Transpoviron (einem mobilen genetischen Element bei Riesenviren) von „Moumouvirus Monve“ (alias „Monve-Virus“, Gattung Mimivirus Linie B).[11]
Ein Zamilon-ORF zeigt zusätzlich eine gewisse Ähnlichkeit mit „Mavirus“ und ein anderer mit „Organic Lake-Virophagen“ und einem „Phaeocystis globosa-Virophagen“, beide sind eher mit Algen als mit Amöben assoziiert. Die verbleibenden zwei vorhergesagten Genprodukte zeigen eine begrenzte Ähnlichkeit mit anderen bekannten Proteinen. Mögliche Funktionen der Produkte umfassen Transposase, Helicase, Integrase, Cysteinprotease, Primase/DNA-Polymerase und DNA-verpackende ATPase-Enzyme, Haupt- und Nebenkapsidproteine, ein Strukturprotein und ein kollagenähnliches Protein.[3][3][12]
| ORF[13] | Aminosäuren | mögliche Funktion | Protein-Homologe | Ähnlichkeit (%) |
|---|---|---|---|---|
| 1 | 111 | ? | none | – |
| 2 | 73 | ? | none | – |
| 3 | 135 | ? | Megavirus chilensis mg3 gene product | 67 |
| 4 | 221 | Transposase | Sputnik virophage 2 putative IS3 family transposase A protein | 40 |
| 5 | 376 | Virionprotein | Sputnik virophage 2 Virionprotein | 66 |
| 6 | 609 | Kapsidprotein | Sputnik virophage Kapsidprotein V20 | 86 |
| 7 | 442 | ? | Sputnik virophage V21 | 70 |
| 8 | 81 | ? | Moumouvirus Monve vermutetes Protein tv_L8 | 72 |
| 9 | 778 | Helicase | Sputnik virophage V13 | 67 |
| 10 | 168 | ? | Sputnik virophage V11 | 53 |
| 11 | 247 | Integrase | Sputnik virophage V10 | 58 |
| 12 | 175 | ? | Sputnik virophage V9 | 77 |
| 13 | 184 | Strukturprotein | Sputnik virophage V8 | 71 |
| 14 | 241 | ? | Sputnik virophage V7 | 80 |
| 15 | 305 | kollagenähnliches Protein | Sputnik virophage V6 | 75 |
| 16 | 121 | ? | Sputnik virophage V5 | 59 |
| 17 | 133 | ? | Sputnik virophage V4 | 55 |
| 18 | 245 | DNA-verpackende ATPase | Sputnik virophage V3 | 81 |
| 19 | 147 | ? | Megavirus chilensis Genprodukt mg664 | 50 |
| 20 | 147 | ? | Sputnik virophage V1 | 60 |
Interessanterweise wurde 2018 eine Gen-Homologe von Sputnik und Zamilon mit dem „Orpheovirus“ gefunden.[14]
Vermehrungszyklus und Helferviren
Wie alle anderen Virophagen repliziert sich Zamilon im Zytoplasma innerhalb der Virusfabrik seines Helfers, der als Wirt fungiert.[3] Zamilon wurde zuerst in Verbindung mit dem Mont1-Virus isoliert, einem Mimivirus-ähnlichen Vertreter der Mimiviridae, der durch seine Polymerase B-Gensequenz in die Mimiviridae-Linie I C klassifiziert wurde. Anschließend wurde gezeigt, dass der Virophage in der Lage ist, sich auch in Verbindung mit Moumouvirus Monve (Monve-Virus) und Acanthamoeba polyphaga Moumouvirus (APMoV)[15][16] aus der Mimiviridae-Linie I B, sowie mit Terra1 und Courdo11 aus Linie I C zu replizieren. Es kann jedoch nicht in Verbindung mit Mimivirus oder Mamavirus (beide Linie I A), repliziert werden.[3][17] Hierin unterscheidet sich Zamilon von Sputnik, der sich in Verbindung mit einem Mimivirus-ähnlichen Mitglied der Mimiviridae replizieren kann.[3]
Zamilon scheint weder die Replikationsfähigkeit seines Helfervirus zu signifikant hemmen, noch die durch die Helferviren schließlich herbeigeführte Lyse der Amöbenzellen-Wirte zu beeinträchtigen. Obwohl das Helfervirus in Gegenwart von Zamilon einen hohen Anteil an abnormalen Virionen bildete, wurden diese auch in Abwesenheit des Virophagen auf einem vergleichbaren Niveau beobachtet.[3] Dies ist ebenfalls ein Unterschied zu Sputnik, der die Infektiosität seines Helfervirus merklich verringert, und dessen Lyse der Amöben hemmt und der mit der Erzeugung eines erhöhten Anteils an abnormalen Mimiviridae-Virionen verbunden ist.[2][3] Bernard La Scola und Kollegen, die sowohl Sputnik als auch Zamilon isolierten, gaben an, dass dies, wenn es sich bestätigt, „das Konzept des Virophagen in Frage stellen würde“, da diesem im Unterschied zu anderen Satellitenviren die Annahme einer schädlichen Wirkung auf das Helfervirus zugrunde liegt.[3]
Weblinks
- NCBI: Genbank accession no. NC_022990.1
- Expasy: ViralZone: Sputnikvirus
Anmerkungen
- Das Material wurde von dieser Quelle kopiert, die unter einer Creative Commons Attribution 4.0 International License verfügbar ist.
Einzelnachweise
- ICTV: ICTV Master Species List 2019.v1, New MSL including all taxa updates since the 2018b release, March 2020 (MSL #35)
- Krupovic M, Kuhn JH, Fischer MG: A classification system for virophages and satellite viruses. In: Archives of Virology. 161, 2016, S. 233–47. doi:10.1007/s00705-015-2622-9. PMID 26446887.
- Gaia M, Benamar S, Boughalmi M, Pagnier I, Croce O, Colson P, Raoult D, La Scola B: Zamilon, a Novel Virophage with Mimiviridae Host Specificity. In: PLOS ONE. 9, 2014, S. e94923. doi:10.1371/journal.pone.0094923. PMID 24747414. PMC 3991649 (freier Volltext).
- Anthony Levasseur, Meriem Bekliz, Eric Chabrière, Pierre Pontarotti, Bernard La Scola, Didier Raoult: MIMIVIRE is a defence system in mimivirus that confers resistance to virophage. In: Nature. 531, 2016, S. 249–252. doi:10.1038/nature17146.
- Ewen Callaway: CRISPR-like 'immune' system discovered in giant virus, in: Nature:News, vom 29. Juni 2016
- Said Mougari, Dehia Sahmi-Bounsiar, Anthony Levasseur, Philippe Colson, Bernard La Scola: Virophages of Giant Viruses: An Update at Eleven. In: Viruses Band 11 Nr. 8, Reihe Viruses of Plants, Fungi and Protozoa , 8. August 2019, 733, doi:10.3390/v11080733.

- Boughalmi M, Saadi H, Pagnier I, Colson P, Fournous G, Raoult D, La Scola B: High-throughput isolation of giant viruses of the Mimiviridae and Marseilleviridae families in the Tunisian environment. In: Environmental Microbiology. 15, 2013, S. 2000–2007. doi:10.1111/1462-2920.12068. PMID 23298151.
- Yutin N, Kapitonov VV, Koonin EV: A new family of hybrid virophages from an animal gut metagenome. In: Biology Direct. 10, 2015, S. 19. doi:10.1186/s13062-015-0054-9.
- Bekliz M, Verneau J, Benamar S, Raoult D, La Scola B, Colson P: A New Zamilon-like Virophage Partial Genome Assembled from a Bioreactor Metagenome. In: Frontiers in Microbiology. 6, 2015, S. 1308. doi:10.3389/fmicb.2015.01308. PMID 26640459. PMC 4661282 (freier Volltext).
- Sandra Jeudy, Lionel Bertaux, Jean-Marie Alempic, Audrey Lartigue, Matthieu Legendre, Lucid Belmudes, Sébastien Santini, Nadège Philippe, Laure Beucher, Emanuele G. Biondi, Sissel Juul, Daniel J. Turner, Yohann Couté, Jean-Michel Claverie, Chantal Abergel: Exploration of the propagation of transpovirons within Mimiviridae reveals a unique example of commensalism in the viral world, in: The ISME Journal Band 14,S. 727–739,10 Dezember 2019. doi:10.1038/s41396-019-0565-y, PDF
- NCBI: Moumouvirus Monve
- Zhang X, Sun S, Xiang Y, Wong J, Klose T, Raoult D, Rossmann MG: Structure of Sputnik, a virophage, at 3.5-Å resolution. In: Proceedings of the National Academy of Sciences of the United States of America. 109, 2012, S. 18431–36. doi:10.1073/pnas.1211702109. PMID 23091035. PMC 3494952 (freier Volltext).
- Alle Daten von Gaia et al.
- Julien Andreani, Jacques Y. B. Khalil, Emeline Baptiste, Issam Hasni, Caroline Michelle, Didier Raoult, Anthony Levasseur, Bernard La Scola: Orpheovirus IHUMI-LCC2: A New Virus among the Giant Viruses, in: Front. Microbiol., 22. Januar 2018, doi:10.3389/fmicb.2017.02643
- Virus-Host DB: Acanthamoeba polyphaga moumouvirus
- NCBI: Acanthamoeba polyphaga moumouvirus
- Aherfia S, La Scola B, Pagniera I, Raoult D, Colson P: The expanding family Marseilleviridae. In: Virology. 466–467, 2014, S. 27–37. doi:10.1016/j.virol.2014.07.014. PMID 25104553.
_Zamilon_progeny.png.webp)