Megavirus
Die „Megaviren“ (s. s.) sind eine vorgeschlagene Gruppe („Mimivirus-Linie C“) von DNA-Viren, deren erster Vertreter, die Spezies „Megavirus chilensis“ (gerne falsch als „Megavirus chiliensis“; Acronym MGVC, auch MVc) 2010 von französischen Wissenschaftlern vor der Küste von Chile entdeckt wurde.[5] MGVC ist ein entfernter Verwandter von Acanthamoeba polyphaga mimivirus (APMV)[6] und infiziert vermutlich Acanthamöben. Das National Center for Biotechnology Information (NCBI) ordnet die Spezies Megavirus chilensis (und die anderen Vertreter der Gruppe) zwar der Gattung Mimivirus (s. l.) zu,[7] wegen der nur relativ entfernten Verwandtschaft[5] käme aber auch eine eigene vorläufige Gattung „Megavirus“ („MGV“) in Frage. Innerhalb der Familie der Mimiviridae werden die Megaviren (mit der gesamten Gattung Mimivirus s. l.) der „Gruppe I“ zugerechnet, einer vorgeschlagenen Unterfamiliemit Namen „Mimivirinae“,[2] „Megavirinae“[3] oder „Megamimivirinae“.[4] Diese Gruppe wird per Vorschlag die Linien A (mit APMV), B (Moumouviren) und C aufgeteilt, wobei die Line C (auch Courdo11-Gruppe) den Megaviren entspricht.[4] Wie die anderen Mitglieder der Gattung Mimivirus s. l. können auch Viren der Spezies Megavirus chilensis von Virophagen befallen werden, z. B. von „Sputnik 3“.[8]
| „Megavirus“ („Mimivirus-Linie C“) | ||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|

„Megavirus“-Virion | ||||||||||||||||||||||
| Systematik | ||||||||||||||||||||||
| ||||||||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||||||||
| ||||||||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||||||||
| „Megavirus“ | ||||||||||||||||||||||
| Kurzbezeichnung | ||||||||||||||||||||||
| MGV | ||||||||||||||||||||||
| Links | ||||||||||||||||||||||
|
Entdeckung
„Megavirus chilensis“ wurde aus Meerwasser isoliert, das im April 2010 vor der Küste von Chile in der Nähe von Las Cruces von Jean-Michel Claverie und Chantal Abergel von der Universität Aix-Marseille gesammelt wurde. Wissenschaftler aus diesem Labor waren auch an der Charakterisierung des Mimivirus beteiligt. Das Virus wurde bei der Kokultivierung einer Reihe von Acanthamöben-Stämmen isoliert, und zwar von den Spezies (Acanthamoeba polyphaga, A. castellanii und A. griffini). Dabei wurde ein Verfahren benutzt, das von Timothy Rowbotham für die Isolierung intrazellulärer parasitischer Bakterien entwickelt worden war.[9] Der natürliche Wirt des MGVC ist vermutlich ein phagozytisches Protozoon, das im Meer- oder Brackwasser lebt.
Struktur
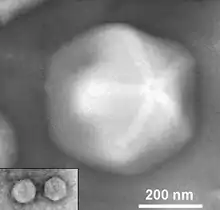
Die Virionen (Virusteilchen) des MGVC bestehen aus einem Protein-Kapsid mit einem Durchmesser von 440 nm. Sie erreichen damit fast die Ausmaße kleiner Bakterien und gehören zu den größten beschriebenen Viren. Die Virionen sind von einem mehrlagigen Tegument-Material von 75 bis 100 nm Schichtdicke umgeben (vgl. Mimivirus-Proteinfilamente). Das Kapsid erscheint hexagonal, allerdings ist seine ikosaedrische Symmetrie nicht perfekt, da es mindestens ein sogenanntes „stargate“ enthält. Als „stargate“ wird eine Struktur bezeichnet, die einem fünfzackigen Stern ähnelt und die eine Öffnung bildet, durch die das Kernmaterial des Virus in das Cytoplasma des Wirts gelangt (siehe Mimivirus §Stargate).[10] Das Kapsid ist von zwei Membranen umgeben, die unterschiedliche virale Proteine enthalten.
Genom
Das Genom von MGVC besteht aus einer linearen Doppelstrang-DNA mit einer Länge von 1.259.197 Basenpaaren. Es war zur Zeit seiner Entdeckung damit das größte bekannte Virusgenom. Es ist um 67,5 kb größer als das Genom des Mimivirus und enthält vermutlich 1120 kodierende Regionen (i. e. kodierte Proteine), was mehr ist als bei vielen Bakterien. Der GC-Gehalt liegt bei 25 %.[11]
MGVC besitzt sieben Aminoacyl-tRNA-Synthetasen, Enzyme, die üblicherweise nur in zellulären Organismen vorkommen. Vier dieser Proteine sind auch aus dem Acanthamoeba polyphaga mimivirus (ApMV) und dem Acanthamoeba castellanii mamavirus (ACMV) bekannt (diejenigen für die Aminosäuren Tyrosin, Arginin, Cystein und Methionin). MGVC enthält zusätzlich die Aminoacyl-tRNA-Synthetasen für Tryptophan, Asparagin und Isoleucin. Das Cafeteria-roenbergensis-Virus (CroV) enthält nur die Aminoacyl-tRNA-Synthetase für Isoleucin. Das MGVC enthält auch das Gen für eine besondere Variante des mismatch repair Enzyms MutS, das sonst nur in den Mitochondrien von Octocorallia vorkommt. Diese MutS-Version scheint nur bei Mitgliedern der Mimiviridae-Familie vorzukommen.[12] MGVC enthält – ähnlich wie ApMV – Gene für den Stoffwechsel von Zuckern, Fetten und Aminosäuren.[13]
MGVC und ApMV haben 594 orthologe Gene gemeinsam. Die meisten dieser übereinstimmenden Gene befinden sich in der Mitte der viralen Genome, jeweils flankiert von speziesspezifischen Abschnitten im 5′- und 3′-Bereich. Bezüglich der Aminosäurensequenzen sind die durch diese orthologen Gene codierten Proteine zu etwa 50 % identisch.
Aus der Analyse des Virusgenoms von MGVC ergaben sich Hinweise darauf, dass Mimiviren und Megaviren (d. h. die Linien A und C) von einem gemeinsamen Vorfahren abstammen könnten, der durch reduktive Evolution aus Bestandteilen eines zellulären Genoms entstanden sei.[14]
Replikation
Die Replikationsstadien von MGVC ähneln denjenigen des Mimivirus. Kurz nach der Phagozytose und der Freisetzung des inneren Kapsids in das Cytoplasma der Wirtszelle beginnt die Eklipse-Phase. Man findet dabei sog. cytoplasmatische „Seeds“, die in ihrer Größe dem Megavirus-Kern entsprechen. Diese Seeds entwickeln sich innerhalb von 14 Stunden zu Virion-Fabriken. Bis zur Lyse der Amöbenzelle und der Freisetzung der Viren dauert es beim MGVC gewöhnlich ca. 17 Stunden (im Gegensatz zu ca. 12 Stunden beim Mimivirus). Es werden dabei ca. 500 neue Viren pro infizierter Zelle produziert – im Gegensatz zu 1000 Partikeln bei Acanthamoeba polyhaga mimivirus (ApMV).
MGVC gehört damit zu einer nicht-taxonomischen Gruppe von großen cytoplasmatischen Viren, bekannt als Nucleocytoplasmic large DNA viruses (NCLDV, vorgeschlagene Virusordnung ‚Megavirales‘). Als Klassifikationsmerkmal dieser Gruppe gilt die Tatsache, dass sie sich vollständig im Cytoplasma des Wirtes mithilfe sogenannter large virion factories vermehren, ohne die doppelsträngige DNA (wie sonst bei ds-DNA-Viren üblich) mithilfe der wirtseigenen DNA-Polymerasen im Zellkern der Wirtszelle zu replizieren.
Bedeutung der Entdeckung
Zwei Eigenschaften von MGVC sind von Bedeutung: Obwohl es nur 6 % größer ist als ApMV, konnte man bereits bei seiner Entdeckung annehmen, dass es nicht das größte Virus ist. Die Tatsache, dass drei weitere Aminoacyl-tRNA-Synthetasen in Virengenomen gefunden wurden, ließ die Vermutung aufkommen, dass diese Enzyme nicht durch einen lateralen Gentransfer von anderen Viren erworben wurden. Die Analyse ihrer Sequenz zeigt Verwandtschaft mit eukaryotischen Genen an. Davon ausgehend wurde vermutet, dass das Genom dieser Viren ähnlich wie bei anderen Parasiten durch Genomreduktion aus dem Genom eines zellulären Organismus entstanden sein könnte. Weiterhin wurde vermutet, dass Megaviren evolutionär sehr alt seien, und zwar eventuell älter als die heutigen Eukaryonten. Sie könnten dann gleichzeitig mit dem Zellkern entstanden sein; eine Vermutung, die durch die Ähnlichkeit des Zellkerns mit der oben erwähnten Virusfabrik unterstützt wird. Eine weitere Vermutung ist, dass Megaviren von einer zwischenzeitlich ausgestorbenen zellulären Domäne abstammen.[15] Diesen Ansichten wurde jedoch auch widersprochen und argumentiert, dass die NCLDV ihrer Größe auf mehreren Linien unabhängig durch Acquisition von Genen per horizontalem Gentransfer erworben haben.[16] Die Entdeckung von Medusavirus hat die Frage allerdings wieder neu aufgeworfen.
Systematik
Weder „Megavirus chilensis“ noch die Gruppe der „Megaviren (Line C)“ sind mit Stand März 2019 vom International Committee on Taxonomy of Viruses (ICTV) bestätigt worden.[17]
Vermutlich werden diese Kandidaten als Mitglieder der vom ICTV bereits bestätigten Familie Mimiviridae (veraltet auch Megaviridae) beschrieben werden. Zur Familie werden vermutlich solche Viren gehören, die zum Avcanthamoeba polypahaga mimivirus (ApMV) und dem MGVC eine hinreichende Struktur- und Sequenzähnlichkeit besitzen (Mimiviridae-Gruppe I). Innerhalb dieser Gruppe wurden aufgrund besonders starker genetischer Übereinstimmung die Megaviren als Linie C (alias Courdo11-Gruppe) vorgeschlagen. Eine Reihe von Kandidaten wurden von Andreani et al. 2018 vorgeschlagen.[18] Das NCBI listet in der Spezies „Megavirus chilensis“ (dort wie auch bei S. Barik und anderen Autoren „Megavirus chiliensis“ geschrieben,[19][20][21]) die folgenden Kandidaten (innere Systematik):[7]
- Spezies: „Megavirus chilensis“ („M.C. chilensis“)[22]
_Sputnik3%2540Courdo11_(ContrastEnh).tif.jpg.webp)
Das NCBI führt u. a. als eigenständige Schwesterspezies von MGVC unter den Megaviren folgende Kandidaten (äußere Systematik):
- Spezies: „Megavirus avenue9“[32][24]
- Spezies: „Megavirus balcon“[33][24]
- Spezies: „Megavirus battle43“[34][24]
- Spezies: „Megavirus bus“[35][24] (alias Mimivirus bus[36][37])
- Spezies: „Megavirus courdo5“[38][24]
- Spezies: „Megavirus courdo7“[39][18][24][20]
- Spezies: „Megavirus feuillage“[40]
- Spezies: „Megavirus mont1“[41][23]
- Spezies: „Megavirus J3“[42][24]
- Spezies: „Megavirus montpellier“[43] (alias „Megavirus montpellier3“[24])
- Spezies: „Megavirus potager“[44]
- Spezies: „Megavirus ursino“[45][18][24]
- Spezies: „Megavirus shan“ (alias „Shan-Virus“)[46][28][24]
- Spezies: „Megavirus T1“[47][24]
- Spezies: „Megavirus T4“[48][24]
- Spezies: „Megavirus T6“[49][24]
- Spezies: „Megavirus vitis“ („M.C. vitis“)[50][19][22]
- Spezies: „Bandra megavirus“ (BMV)[51][29]
- Spezies: „Afrovirus urmite69“[52][53]
Vorgeschlagener phylogenetischer Baum der Gattung Mimivirus nach Abrahão et al. (2018), Fig. 4:[36]
| Mimivirus |
| ||||||||||||||||||||||||||||||||||||||||||
Anmerkung: Kursivschrift für vorgeschlagene Spezies.
Genomvergleich mit verwandten Viren
| Virus | Genom-Größe (bp) | Gene | Kapsid-Durchmesser (nm) | Genbank-Nr. |
|---|---|---|---|---|
| Megavirus chilensis[5] | 1.259.197 | 1120 Proteine (abgeleitet) | 440 | JN258408 |
| Mamavirus ACMV[54] | 1.191.693 | 1023 Proteine (abgeleitet) | 390 | JF801956 |
| Mimivirus ApMV[6][55] (Wildtyp M1) | 1.181.549 | 979 Proteine (39 nicht-codierend) | 390 | NC_014649 |
| Mimivirus M4[56] (bald/fiberless Variante) | 981.813 | 756 Proteine (abgeleitet) | 390 | JN036606 |
| Cafeteria-roenbergensis-Virus[57] | 617.453 (730 kbp) | 544 Proteine (abgeleitet) | 300 | NC_014637 |
Merkmale der Mimiviridae im Vergleich
| Virus | Aminoacyl-tRNA-Synthetase | Octocorallia-ähnliche MutS | Proteinfilamente (Länge) | Stargate[10] | Bekannter Virophage[58] | Cytoplasmische Virion-Fabrik |
Wirt |
|---|---|---|---|---|---|---|---|
| Megavirus chilensis | 7 (Tyr, Arg, Met, Cys, Trp, Asn, Ile) | ja | ja (75 nm) | ja | nein | ja | Acanthamöben (Unikonta, Amoebozoa) |
| Mamavirus ACMV | 4 (Tyr, Arg, Met, Cys) | ja | ja (120 nm) | ja | ja | ja | Acanthamöben (Unikonta, Amoebozoa) |
| Mimivirus ApMV (Wildtyp M1) | 4 (Tyr, Arg, Met, Cys) | ja | ja (120 nm) | ja | ja | ja | Acanthamöben (Unikonta, Amoebozoa) |
| Mimivirus M4 (bald/fiberless Variante) | 2 (Met, Cys) | nein | nein | ja | resistent | ja | Acanthamöben (Unikonta, Amoebozoa) |
| Cafeteria-roenbergensis-Virus | 1 (Ile) | ja | nein | nein | ja | ja | Phagotrophische Protozoen (Heterokonta, Stramenopiles) |
Literatur
- James L. Van Etten: Giant Viruses. In: American Scientist. Band 99, Nr. 4, 2011, S. 304, doi:10.1511/2011.91.304.
- Elodie Ghedin, Jean-Michel Claverie: Mimivirus relatives in the Sargasso sea. In: Virology Journal. Band 2, 2005, S. 62, doi:10.1186/1743-422X-2-62, PMID 16105173, PMC 1215527 (freier Volltext).
- Adam Monier, Jean-Michel Claverie, Hiroyuki Ogata: Taxonomic distribution of large DNA viruses in the sea. In: Genome Biology. Band 9, Nr. 7, 2008, S. R106, doi:10.1186/gb-2008-9-7-r106, PMID 18598358, PMC 2530865 (freier Volltext).
Weblinks
- GiantVirus.org – an information resource on the genome of giant viruses.
- Stuart Siddell: Why virus taxonomy is important, Microbiology Society, 13. Februar 2018
- Jean-Luc Goudet: Nouméavirus, un étonnant virus géant qui agit à distance. Futura Santé, 1. Mai 2017 (mit bildlichen Darstellungen von Megavirus, französisch)
Anmerkungen
- Das Material wurde von dieser Quelle kopiert, die unter einer Creative Commons Attribution 4.0 International License verfügbar ist.
Einzelnachweise
- ICTV: ICTV Taxonomy history: Acanthamoeba polyphaga mimivirus, EC 51, Berlin, Germany, July 2019; Email ratification March 2020 (MSL #35)
- Christoph M. Deeg, Cheryl-Emiliane T. Chow, Curtis A. Suttle: The kinetoplastid-infecting Bodo saltans virus (BsV), a window into the most abundant giant viruses in the sea… In: eLife Sciences, 7, März 2018, doi:10.7554/eLife.33014
- List of the main “giant” viruses known as of today (March 2019) (PDF) Centre national de la recherche scientifique, Université Aix Marseille, März 2019.
- Frederik Schulz, Lauren Alteio, Danielle Goudeau, Elizabeth M. Ryan, Feiqiao B. Yu, Rex R. Malmstrom, Jeffrey Blanchard, Tanja Woyke: Hidden diversity of soil giant viruses. In: Nature Communications, Band 9, Nr. 4881, 19. November 2018, doi:10.1038/s41467-018-07335-2
- Defne Arslan, Matthieu Legendre, Virginie Seltzer, Chantal Abergel, Jean-Michel Claverie: Distant Mimivirus relative with a larger genome highlights the fundamental features of Megaviridae. In: PNAS. 10. Oktober 2011, doi:10.1073/pnas.1110889108.
- D. Raoult u. a.: The 1.2-Megabase Genome Sequence of Mimivirus. In: Science. Band 306, Nr. 5700, 2004, S. 1344–1350, doi:10.1126/science.1101485, PMID 15486256.
- NCBI: Megavirus chiliensis (Tree), Megavirus chiliensis (Species)
- Morgan Gaia, Isabelle Pagnier, Angélique Campocasso, Ghislain Fournous, Didier Raoult, Bernard La Scola: Broad spectrum of mimiviridae virophage allows its isolation using a mimivirus reporter. PLoS One, 2013, 8, S. e61912, PMC 3626643 (freier Volltext), doi:10.1371/journal.pone.0061912
- T. J. Rowbotham: Isolation of Legionella pneumophila from clinical specimens via amoebae, and the interaction of those and other isolates with amoebae. In: Journal of Clinical Pathology. Band 36, Nr. 9, 1983, S. 978–986, doi:10.1136/jcp.36.9.978, PMID 6350372, PMC 498455 (freier Volltext).
- Nathan Zauberman, Y. Mutsafi, D. B. Halevy, E. Shimoni, E. Klein, C. Xiao, S. Sun, A. Minsky: Distinct DNA Exit and Packaging Portals in the Virus Acanthamoeba polyphaga mimivirus. In: PLoS Biology. Band 6, Nr. 5, 2008, S. e114, doi:10.1371/journal.pbio.0060114, PMID 18479185, PMC 2430901 (freier Volltext).
- David M. Needham, Alexandra Z. Worden et al.: A distinct lineage of giant viruses brings a rhodopsin photosystem to unicellular marine predators. In: PNAS, 23. September 2019, doi:10.1073/pnas.1907517116, ISSN 0027-8424, hier: Supplement 1 (xlsx)
- Hiroyuki Ogata, J. Ray, K. Toyoda, R. A. Sandaa, K. Nagasaki, G. Bratbak, J. M. Claverie: Two new subfamilies of DNA mismatch repair proteins (MutS) specifically abundant in the marine environment. In: The ISME Journal. Band 5, Nr. 7, 2011, S. 1143–1151, doi:10.1038/ismej.2010.210, PMID 21248859, PMC 3146287 (freier Volltext).
- Jean-Michel Claverie, Chantal Abergel: Mimivirus: The emerging paradox of quasi-autonomous viruses. In: Trends in Genetics. Band 26, Nr. 10, 2010, S. 431–437, doi:10.1016/j.tig.2010.07.003, PMID 20696492.
- M. Legendre, D. Arslan, C. Abergel, J. M. Claverie: Genomics of Megavirus and the elusive fourth domain of Life., in: Commun Integr Biol. 5(1), 1. Januar 2012, S. 102–106, PMID 22482024.
- Gwyneth Dickey Zakaib: The challenge of microbial diversity: Out on a limb. In: Nature. Band 476, Nr. 7358, 2011, S. 20–21, doi:10.1038/476020a, PMID 21814255.
- Natalya Yutin, Eugene V Koonin: Hidden evolutionary complexity of Nucleo-Cytoplasmic Large DNA viruses of eukaryotes. In: Virology Journal, Band 9, Nr. 161, 14. August 2012, doi:10.1186/1743-422X-9-161
- ICTV: Master Species List 2018b.v2. Abgerufen am 6. August 2019. MSL #34v
- Julien Andreani, Jacques Y. B. Khalil, Emeline Baptiste, Issam Hasni, Caroline Michelle, Didier Raoult, Anthony Levasseur, Bernard La Scola: Orpheovirus IHUMI-LCC2: A New Virus among the Giant Viruses. In: Front. Microbiol., 22. Januar 2018, doi:10.3389/fmicb.2017.02643
- Sailen Barik: A Family of Novel Cyclophilins, Conserved in the Mimivirus Genus of the Giant DNA Viruses. In: Computational and Structural Biotechnology Journal, Band 16, Juli 2018, S. 231–236, doi:10.1016/j.csbj.2018.07.001
- Julien Guglielmini, Anthony C. Woo, Mart Krupovic, Patrick Forterre, Morgan Gaia: pnas.org In: PNAS, Band 116, Nr. 39, 10./24. September 2019, S. 19585–19592, doi:10.1073/pnas.1912006116, PMID 31506349, Fig. 2
- Eugene V. Koonin, Natalya Yutin: Multiple evolutionary origins of giant viruses. In: F1000 Research, 22. November 2018, doi:10.12688/f1000research.16248.1, version 1
- Sandra Jeudy, Lionel Bertaux, Jean-Marie Alempic, Audrey Lartigue, Matthieu Legendre, Lucid Belmudes, Sébastien Santini, Nadège Philippe, Laure Beucher, Emanuele G. Biondi, Sissel Juul, Daniel J. Turner, Yohann Couté, Jean-Michel Claverie, Chantal Abergel: Exploration of the propagation of transpovirons within Mimiviridae reveals a unique example of commensalism in the viral world. In: The ISME Journal, Band 14,S. 727–739,10 Dezember 2019. doi:10.1038/s41396-019-0565-y, nature.com (PDF)
- Morgan Gaia, Samia Benamar, Mondher Boughalmi, Isabelle Pagnier, Olivier Croce, Philippe Colson, Didier Raoult, Bernard La Scola: Zamilon, a Novel Virophage with Mimiviridae Host Specificity. In: PLoS One. 9(4), 2014, S. e94923. doi:10.1371/journal.pone.0094923. PMID 24747414. PMC 3991649 (freier Volltext). Online veröffentlicht am 18. April 2014
- Didier Raoult, Anthony Levasseur, Bernard La Scola: PCR Detection of Mimivirus, in: Emerging Infectious Diseases, Juni 2017, Bd. 23, Nr. 6, S. 1044–1045, doi:https://dx.doi.org/10.3201/eid2306.161896, PDF (PDF)
- NCBI: Courdo virus CE11
- Clara Rolland, Julien Andreani, Amina Cherif Louazani, Sarah Aherfi, Rania Francis, Rodrigo Rodrigues, Ludmila Santos Silva, Dehia Sahmi, Said Mougari, Nisrine Chelkha, Meriem Bekliz, Lorena Silva, Felipe Assis, Fábio Dornas, Jacques Yaacoub Bou Khalil, Isabelle Pagnier, Christelle Desnues, Anthony Levasseur, Philippe Colson, Jônatas Abrahão, Bernard La Scola: Discovery and Further Studies on Giant Viruses at the IHU Mediterranee Infection That Modified the Perception of the Virosphere. In: Viruses, 11(4), März/April 2019, pii: E312, doi:10.3390/v11040312, PMC 6520786 (freier Volltext), PMID 30935049, siehe Tab. 1
- NCBI: LBA111 (Isolate)
- Hanene Saadi, Dorine-Gaelle Ikanga Reteno, Philippe Colson, Sarah Aherfi, Philippe Minodier, Isabelle Pagnier, Didier Raoult, Bernard La Scola: Shan Virus: A New Mimivirus Isolated from the Stool of a Tunisian Patient with Pneumonia. (PDF; 592 kB) In: Intervirology, 2013, Nr. 56, S. 424–429, doi:10.1159/000354564
- Hansika Chhabra: Giant viruses found in water samples from Mumbai. Hindu BusinessLine: Science, Bangalore, 9. Mai 2019
- Anirvan Chatterjee et al.: Complete Genome Sequence of a New Megavirus Family Member Isolated from an Inland Water Lake for the First Time in India. In: Microbiology Resource Announcements, 2016, doi:10.1128/genomeA.00402-16
- Anirvan Chatterjee, Thomas Sicheritz-Pontén, Rajesh Yadav, Kiran Kondabagil: Genomic and metagenomic signatures of giant viruses are ubiquitous in water samples from sewage, inland lake, waste water treatment plant, and municipal water supply in Mumbai, India, in: Scientific Reports, Band 9, Nr. 3690, 6. März 2019, doi:10.1038/s41598-019-40171-y, PMID 30842490, PMC 6403294 (freier Volltext)
- NCBI: Megavirus avenue9 (Species)
- NCBI: Megavirus balcon (Species)
- NCBI: Megavirus battle43 (Species)
- NCBI: Megavirus bus (Species)
- Jônatas Abrahão, Lorena Silva, Ludmila Santos Silva, Jacques Yaacoub Bou Khalil, Rodrigo Rodrigues, Thalita Arantes, Felipe Assis, Paulo Boratto, Miguel Andrade, Erna Geessien Kroon, Bergmann Ribeiro, Ivan Bergier, Herve Seligmann, Eric Ghigo, Philippe Colson, Anthony Levasseur, Guido Kroemer, Didier Raoult, Bernard La Scola: Tailed giant Tupanvirus possesses the most complete translational apparatus of the known virosphere. In: Nature Communications. 9, Nr. 1, 27. Februar 2018. doi:10.1038/s41467-018-03168-1.
- Anirvan Chatterjee, Thomas Sicheritz-Pontén, Rajesh Yadav, Kiran Kondabagil: Isolation and complete genome sequencing of Mimivirus bombay, a Giant Virus in sewage of Mumbai, India. In: Genomics Data, 9(C), Mai 2016, doi:10.1016/j.gdata.2016.05.013, Fig. 2
- NCBI: Megavirus courdo5 (Species)
- NCBI: Megavirus courdo7 (Species)
- NCBI: Megavirus feuillage (Species)
- NCBI: Megavirus mont1 (Species)
- NCBI: Megavirus J36 (Species)
- NCBI: Megavirus montpellier (Species)
- NCBI: Megavirus potager (Species)
- NCBI: Megavirus ursino (Species)
- NCBI: Megavirus shan (Species)
- NCBI: Megavirus T1 (Species)
- NCBI: Megavirus T4 (Species)
- NCBI: Megavirus T6 (Species)
- NCBI: Megavirus vitis (Species)
- NCBI: Bandra megavirus (Species)
- NCBI: Afrovirus urmite69 (Species)
- Anthony Levasseur, Meriem Bekliz, Eric Chabrière, Pierre Pontarotti, Bernard La Scola, Didier Raoult: MIMIVIRE is a defence system in mimivirus that confers resistance to virophage. In: Nature, 2016, doi:10.1038/nature17146, Supplementary Information. (PDF)
- P. Colson u. a.: Viruses with More Than 1,000 Genes: Mamavirus, a New Acanthamoeba polyphaga mimivirus Strain, and Reannotation of Mimivirus Genes. In: Genome Biology and Evolution. Band 3, 2011, S. 737–742, doi:10.1093/gbe/evr048, PMID 21705471, PMC 3163472 (freier Volltext).
- Matthieu Legendre u. a.: Breaking the 1000-gene barrier for Mimivirus using ultra-deep genome and transcriptome sequencing. In: Virology Journal. Band 8, 2011, S. 99, doi:10.1186/1743-422X-8-99, PMID 21375749, PMC 3058096 (freier Volltext).
- M. Boyer u. a.: Mimivirus shows dramatic genome reduction after intraamoebal culture. In: Proceedings of the National Academy of Sciences. Band 108, Nr. 25, 2011, S. 10296–10301, doi:10.1073/pnas.1101118108, PMC 3121840 (freier Volltext).
- M. G. Fischer u. a.: Giant virus with a remarkable complement of genes infects marine zooplankton. In: Proceedings of the National Academy of Sciences. Band 107, Nr. 45, 2010, S. 19508–19513, doi:10.1073/pnas.1007615107, PMC 2984142 (freier Volltext).
- M. G. Fischer, C. A. Suttle: A Virophage at the Origin of Large DNA Transposons. In: Science. Band 332, Nr. 6026, 2011, S. 231–234, doi:10.1126/science.1199412, PMID 21385722.