Rekombinase
Rekombinasen sind Enzyme, welche die genetische Rekombination katalysieren. Hierbei kommt es zu einer Spaltung und Neuverknüpfung von DNA-Abschnitten, was zur genetischen Diversität führt und die Reparatur mutierter DNA ermöglicht.
Homologe Rekombination
Die homologe Rekombination wurde 1964 zuerst von Robin Holliday beschrieben. Sie beruht auf der Paarung ausgedehnter homologer Sequenzen, ist in Bakterien und Hefen häufig, in Säugerzellen hingegen ineffizient. Dies hängt mit der Komplexität und Größe höherer Genome zusammen und beschränkt die Einsatzmöglichkeiten des Prozesses zur gezielten genetischen Modifikation dieses Zelltyps.
Ortsspezifische Rekombination
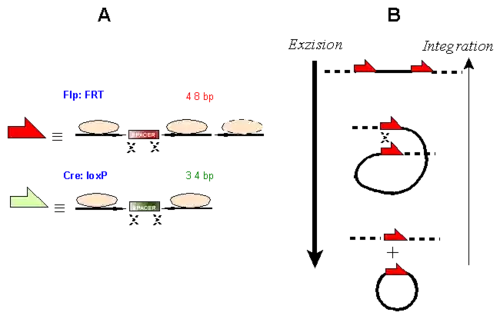 |
Abbildung: ortsspezifische Rekombinasen: Exzision und Integration am Beispiel der Rekombinasen Cre und Flp. Die Erkennungsstelle der Cre-Rekombinase (loxP-Stelle [Locus of Crossingover of P1 phage]) umfasst 34 Basenpaare, darin zwei 13 Basenpaare lange Kontaktstellen und ein 8 Basenpaare umfassender Abstandhalter („Spacer“). Die Flp Erkennungsstelle (FRT [Flp-recombinase target]) ist durch eine zusätzliche Kontaktstelle etwas länger (A).
Als „Methode der Wahl“ gilt zu diesem Zweck heute das RMCE Kassettenaustauschverfahren, welches diese Probleme umgeht.
|
Rekombinationsereignisse dieses Typs verlaufen über kurze Erkennungsstellen, wie sie in Hefen und Phagen vorkommen. Da es gelingt, den entsprechenden enzymatischen Apparat in Säugerzellen zu übertragen, steht ein äußerst effizientes System zur genetischen Modifikation auch höherer Zellen zur Verfügung. Davon wird seit einigen Jahren zunehmend Gebrauch gemacht.
Die am häufigsten verwendeten Rekombinationssysteme dieser Klasse leiten sich von den Rekombinasen Cre (cyclization recombinase oder auch causes recombination) des Phagen P1, Flp (benannt nach der Flippase-Aktivität, durch die Hefen Sequenzabschnitte invertieren) oder Xer ab.[1] Beide Enzyme gehören zur Integrase Familie der Rekombinasen, die derzeit etwa 30 Mitglieder umfasst. Die folgende Abbildung illustriert Möglichkeiten, die aus diesen Systemen resultieren. Darüber hinaus hat die systematische Mutagenese der Erkennungsstellen einen weiteren Reaktionstyp ermöglicht: das RMCE Kassettenaustauschverfahren, dem ein gesonderter Eintrag gewidmet ist.
Medizinische Bedeutung
2007 gelang es deutschen Forschern erstmals eine Rekombinase für HIV-1 infizierte Zellen zu nutzen. Hierbei wird die virale DNA mit Hilfe einer modifizierten Cre-Rekombinase (Tre) aus dem Genom einer CD4-Helferzelle herausgetrennt.[2] Ob die vollständige Eradikation von HIV-1 unter Mithilfe der Tre-Rekombinase in Zukunft als gentherapeutische Behandlungsstrategie tatsächlich möglich sein wird, muss derzeit mit größter Vorsicht beurteilt werden. Es existieren eine Reihe prinzipieller, wie auch technischer Probleme, die in den kommenden Jahren sorgfältig analysiert und gelöst werden müssen.[3]
Dennoch lassen die aktuellen Forschungsergebnisse zumindest theoretische Gedankenspiele zur vollständigen Eradikation zu. Sollte eine Eradikation nicht möglich sein, könnte zumindest ein weiterer, alternativer Ansatz zur HAART gegeben sein.
Quellen
- B. Das, E. Martínez, C. Midonet, F. X. Barre: Integrative mobile elements exploiting Xer recombination. In: Trends in microbiology. Band 21, Nummer 1, Januar 2013, S. 23–30, doi:10.1016/j.tim.2012.10.003. PMID 23127381.
- Sarkar, I. et al. (2007): HIV-1 proviral DNA excision using an evolved recombinase. In: Science. Bd. 316, S. 1912–1915. PMID 17600219
- Frank Buchholz, Joachim Hauber: Maßgeschneiderte Rekombinase – ein neuer Hoffnungsschimmer zur HIV-Eradikation. In: Retrovirus Bulletin. Jg. 2007, Nr. 3, S. 9–11.
Literatur
- Andrews, B.J. et al. (1985) The FLP recombinase of the 2 micron circle DNA of yeast: interaction with its target sequences. In: Cell. Bd. 40, S. 795–803. PMID 3879971
Weblinks
- DNA-Rekombination: Holliday Modell
- Martin E. Mulligan: Biochemistry 3107 - Fall 2001: Site-Specific Recombination (Memento vom 9. März 2011 im Internet Archive)