Glycerinaldehyd-3-phosphat-Dehydrogenase
Die Glycerinaldehyd-3-phosphat-Dehydrogenase (GAPDH) ist ein Enzym der Glycolyse und daher unentbehrlich für alle Lebewesen. Sie katalysiert die Umwandlung von Glycerinaldehyd-3-phosphat zu 1,3-Bisphosphoglycerat. Bei dieser Reaktion wird eine energiereiche Phosphat-Bindung aufgebaut, die im folgenden Schritt der Glycolyse auf ADP übertragen wird, wodurch ATP entsteht. Außerdem wird in der katalysierten Reaktion ein NAD+ zu NADH/H+ umgewandelt.
| Glyceraldehyd-3-Phosphat-Dehydrogenase | ||
|---|---|---|
| ||
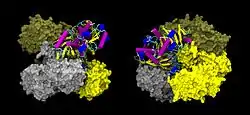
| Oberflächen-/Bändermodell des GAPDH-Tetramer, nach PDB 1U8F | ||
| Eigenschaften des menschlichen Proteins | ||
| Masse/Länge Primärstruktur | 334 aa; 35,9 kDa | |
| Sekundär- bis Quartärstruktur | Homotetramer | |
| Kofaktor | PRKCI, Sulfat | |
| Bezeichner | ||
| Gen-Namen | GAPDH ; GAPD | |
| Externe IDs | ||
| Enzymklassifikation | ||
| EC, Kategorie | 1.2.1.12, Oxidoreduktase | |
| Reaktionsart | Phosphorylierung | |
| Substrat | D-Glyceraldehyd-3-phosphat + Phosphat + NAD(+) | |
| Produkte | 3-Phospho-D-glyceroylphosphat + NADH | |
| Vorkommen | ||
| Homologie-Familie | CLU_030140_0_3 | |
| Übergeordnetes Taxon | Lebewesen | |
| Orthologe | ||
| Mensch | Huhn | |
| Entrez | 2597 | 374193 |
| Ensembl | ENSG00000111640 | ENSGALG00000014442 |
| UniProt | P04406 | P00356 |
| Refseq (mRNA) | NM_002046 | NM_204305 |
| Refseq (Protein) | NP_002037 | NP_989636 |
| Genlocus | Chr 12: 6.51 – 6.52 Mb | Chr 1: 80.09 – 80.09 Mb |
| PubMed-Suche | 2597 | 374193 |
Struktur
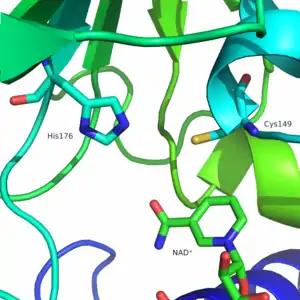
Unter normalen zellulären Bedingungen existiert das cytoplasmatische GAPDH hauptsächlich als Tetramer. Diese Form besteht aus vier identischen 37-kDa-Untereinheiten, die jeweils eine einzige katalytische Thiolgruppe enthalten und für die katalytische Funktion des Enzyms entscheidend sind.[1][2] Die GAPDH im Zellkern hat einen erhöhten isoelektrischen Punkt (pI) bei pH 8,3 bis 8,7.[2] Der Cysteinrest Cys152 im aktiven Zentrum des Enzyms ist für die Induktion der Apoptose durch oxidativen Stress erforderlich.[2] Insbesondere tragen posttranslationale Modifikationen der cytoplasmatischen GAPDH zu seinen Funktionen außerhalb der Glykolyse bei.[1]
Katalytischer Mechanismus
Zum Verständnis des Reaktionsmechanismus der GAPDH haben folgende enzymologische Experimente beigetragen:[3]
- GAPDH wurde durch Alkylierung mit stöchiometrischen Mengen an Iodacetat unwirksam gemacht. Das Vorhandensein von Carboxymethylcystein im Hydrolysat des resultierenden alkylierten Enzyms zeigt, dass GAPDH im aktiven Zentrum einen Cysteinrest besitzt, dessen Thiolgruppe im Mechanismus eine Rolle spielt.

- GAPDH überträgt quantitativ ein Tritium vom C1-Atom von GAP zu NAD+. Damit konnte man beweisen, dass die Reaktion über eine Hydridübertragung verläuft.

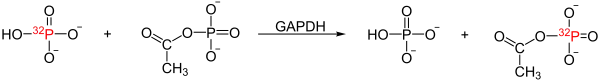
- GAPDH katalysiert den Austausch von 32P zwischen Pi und Acetylphosphat.[4] Solche Isotopenaustauschreaktionen weisen auf einen Acyl-Enzym als Zwischenprodukt hin, d. h., dass die Acylgruppe einen kovalenten Komplex mit dem Enzym bildet, ähnlich wie das Acyl-Enzym-Intermediat im Reaktionsmechanismus von Serinproteasen.
Das Hauptproblem der Reaktion stellt die Abspaltung des H−-Ion (Hydridion) von der Aldehydgruppe des Glycerinaldehyd-3-Phosphats dar. Dies ist energetisch ungünstig, da der Kohlenstoff in der Aldehydgruppe eine positive Partialladung trägt. Durch eine Sulfhydrylgruppe eines Cysteins des Enzyms wird über eine kovalente Bindung ein nukleophiler Rest eingeführt. Das Nukleophil greift den Carbonylkohlenstoff von Glycerinaldehyd-3-phosphat (GAP) an, um ein Thiohalbacetal zu bilden. Nach Abstraktion des Protons der Hydroxygruppe am C1-Atom von GAP durch eine Base löst sich das Hydridion und zwischen Enzym und Substrat entsteht ein Thioester (Oxidation). Diese energiereiche Verbindung wird in der nachfolgenden Reaktion benutzt, um ein anorganisches Phosphat zu binden und das Zwischenprodukt in 1,3-Bisphosphoglycerat umzuwandeln (Phosphorylierung). Das Hydridion bindet mittlerweile an ein nicht-kovalent an einer Rossmann-Faltung gebundenes NAD+,[5] so dass NADH/H+entsteht. Dieses löst sich aus der Bindung mit dem Enzym und wird durch ein NAD+ ersetzt,[6] weil die positive Ladung des NAD+ das Thioester-Zwischenprodukt polarisiert, um den Angriff durch das Orthophosphat zu erleichtern.[7] Die katalysierte Reaktion nimmt energetisch einen hohen Stellenwert ein. Das entstandene gemischte Anhydrid aus Phosphor- und Carbonsäure wird in der nachfolgenden Reaktion zur ATP-Bildung verwendet. Aus dem NADH/H+ kann wiederum ATP in der Atmungskette gebildet werden.[8] Weil Zellen nur eine begrenzte Menge an NAD+ besitzen, würde die Glykolyse stehenbleiben, wenn das in der Glykolyse produzierte NADH nicht kontinuierlich reoxidiert werden würde. Daher wird NAD+ aus dem anaeroben Abbau von Pyruvat regeneriert.[5][9]
Die von der Glycerinaldehyd-3-phosphat-Dehydrogenase katalysierte Reaktion ist in Wirklichkeit die Summer zweier Prozesse: der Oxidation von Glycerinaldehyd-3-phosphat zu 3-Phosphoglycerat durch NAD+ und der Phosphorylierung von 3-Phosphoglycerat zu 1,3-Bisphosphoglycerat (Dehydratisierung).
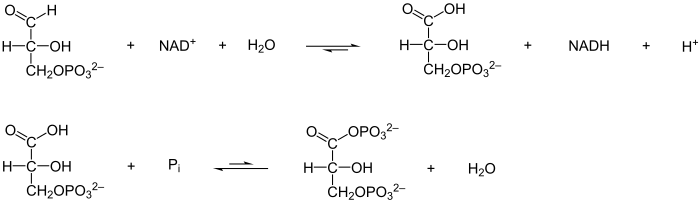
Die erste Reaktion ist mit einer Änderung der freien Standardenthalpie () von etwa −50 kJ/mol thermodynamisch recht günstig. Die zweite Reaktion verläuft mit einer freien Standardenthalpie von ungefähr +50 kJ/mol sehr ungünstig. Daher müssen die beiden Reaktion miteinander gekoppelt verlaufen, damit die Aldehydoxidation die Bildung des Acylphosphats antreiben kann. Die Kopplung erfolgt durch ein Thioester-Zwischenprodukt, welches durch eine Thioesterbindung an das Enzym gebunden ist. Thioester sind energiereiche Verbindungen, die man in vielen biochemischen Stoffwechselwegen findet. Die freie Energie des Thioester-Zwischenproduktes ist größer als die der freien Carbonsäure. Damit bleibt der größte Teil der freien Enthalpie, die bei der Oxidationsreaktion frei wurde, erhalten.[12]
Eigenschaften und Funktion
Alle Schritte der Glykolyse finden im Cytosol statt, ebenso die durch GAPDH katalysierte Reaktion. In roten Blutkörperchen bilden GAPDH und mehrere andere glykolytische Enzyme bestimmte Enzymkomplexe auf der Innenseite der Zellmembran. Der Prozess scheint durch Phosphorylierung und Oxygenierung reguliert zu werden.[13] Es wird erwartet, dass die Annäherung mehrerer glykolytischer Enzyme die Gesamtgeschwindigkeit des Glukoseabbaus stark erhöht. Jüngste Studien haben auch gezeigt, dass GAPDH auf der Außenseite der Zellmembran eisenabhängig exprimiert wird und dort eine Rolle bei der Aufrechterhaltung der zellulären Eisenhomöostase spielt,[14][15] insbesondere als Chaperonprotein für labiles Häm in Zellen.[16]
Transkription und Apoptose
GAPDH kann selbst die Transkription aktivieren. Der OCA-S-Transkriptionscoaktivator-Komplex enthält GAPDH und Lactatdehydrogenase, zwei Proteine von denen bisher angenommen wurde, dass sie nur am Stoffwechsel beteiligt sind. GAPDH bewegt sich zwischen dem Cytosol und dem Zellkern und kann so den metabolischen Zustand mit der Gentranskription verbinden.[17]
Im Jahr 2005 haben Hara et al. gezeigt, dass GAPDH die Apoptose initiiert. Die Initiation wird durch die GAPDH-Bindung an DNA vermittelt. Die Studie zeigte, dass GAPDH durch Stickstoffmonoxid als Reaktion auf Zellstress S-nitrosyliert wird, wodurch es an das Protein SIAH1, eine Ubiquitin-Ligase, bindet. Der Komplex bewegt sich in den Zellkern, in dem SIAH1 auf Kernproteine zum Abbau abzielt, wodurch ein kontrolliertes Herunterfahren der Zellen eingeleitet wird.[18] In einer anschließenden Studie zeigte die Gruppe, dass Selegilin, das klinisch zur Behandlung der Parkinson-Krankheit eingesetzt wurde, die apoptotische Wirkung von GAPDH stark reduziert, indem es dessen S-Nitrosylierung verhindert und daher möglicherweise als Medikament eingesetzt werden kann.[19]
Stoffwechselschalter
GAPDH wirkt unter oxidativem Stress als reversibler Stoffwechselschalter.[20] Wenn Zellen bestimmten Oxidationsmitteln ausgesetzt sind, benötigen sie übermäßige Mengen des antioxidativen Cofaktors NADPH. Im Cytosol wird NADPH durch mehrere Enzyme aus der Reduktion von NADP+ hergestellt, von denen drei die ersten Schritte des Pentosephosphatwegs katalysieren. Oxidationsmittel-Behandlungen verursachen eine Inaktivierung von GAPDH. Durch diese Inaktivierung wird der Stoffwechselfluss von der Glykolyse auf den Pentosephosphatweg umgeleitet, sodass die Zelle mehr NADPH erzeugen kann.[21] Unter Stressbedingungen wird NADPH von einigen Antioxidanssystemen, einschließlich Glutaredoxin und Thioredoxin, benötigt und ist für das Recycling von Glutathion essentiell.
Hemmung
Die Bildung des energiereichen Anhydrids ist durch Arsen hemmbar. AsO43− bindet dabei analog zum Phosphat an die GAPDH. NADH wird weiterhin gebildet.[22] Die Bindung zwischen dem Carboxylat, das aus der Oxidation des Aldehyds entsteht und dem Arsenat ist jedoch sehr instabil, so dass das gemischte Anhydrid zu 3-Phosphoglycerat zerfällt. Dadurch wird ein energiefixierender Schritt in der Glykolyse übersprungen, was zur Giftwirkung des Arsens beiträgt.[23]
Weitere Funktionen
GAPDH scheint auch am Vesikeltransport vom endoplasmatischen Retikulum (ER) zum Golgi-Apparat beteiligt zu sein, der Teil der Transportroute für sezernierte Proteine ist. Es wurde festgestellt, dass GAPDH von Rab2 in die vesikulär-tubulären Cluster des ER rekrutiert wird, wo es zur Bildung von COPI-Vesikeln beiträgt. GAPDH wird durch Tyrosinphosphorylierung durch Tyrosinkinase Src aktiviert.[24]
Da das GAPDH-Gen in den meisten Geweben und Zellen in hohem Maße stabil und konstitutiv exprimiert wird, wird es als Haushaltsgen angesehen. Aus diesem Grund wird GAPDH von biologischen Forschern häufig als Ladungskontrolle für Western Blot und als Kontrolle für die qPCR verwendet. Forscher berichteten jedoch über unterschiedliche GAPDH-Regulationen unter bestimmten Bedingungen.[25] Beispielsweise wurde gezeigt, dass der Transkriptionsfaktor MZF-1 das GAPDH-Gen reguliert.[26] Daher muss die Verwendung von GAPDH als Ladungskontrolle sorgfältig abgewogen werden.
Klinische Bedeutung
Krebs
GAPDH wird bei mehreren Krebsarten beim Menschen, wie dem Hautmelanom, überexprimiert und seine Expression korreliert positiv mit dem Fortschreiten des Tumors.[27][28] Seine glykolytischen und anti-apoptotischen Funktionen tragen zur Proliferation und zum Schutz von Tumorzellen bei und fördern die Karzinogenese. Insbesondere schützt GAPDH vor Telomerverkürzung, die durch chemotherapeutische Medikamente induziert wird, die das Sphingolipid Ceramid stimulieren. Ebenso beeinträchtigen Bedingungen wie oxidativer Stress die GAPDH-Funktion und führen zu Zellalterung und Tod.[2] Darüber hinaus wird durch die Entfernung von GAPDH eine Seneszenz in Tumorzellen induziert, was eine neuartige Therapiestrategie zur Kontrolle des Tumorwachstums darstellt.[29]
Neurodegenerative Erkrankung
GAPDH ist an mehreren neurodegenerativen Krankheiten und Störungen beteiligt, hauptsächlich durch Wechselwirkungen mit anderen Proteinen, die für diese Krankheit oder Störung spezifisch sind. Diese Wechselwirkungen können nicht nur den Energiestoffwechsel, sondern auch andere GAPDH-Funktionen beeinflussen.[1] Beispielsweise könnten GAPDH-Wechselwirkungen mit dem β-Amyloid-Precursor-Protein (BetaAPP) die Funktion des Cytoskeletts oder des Membrantransports beeinträchtigen, während Wechselwirkungen mit Huntingtin die Funktion der Apoptose, des nukleären tRNA-Transports, der DNA-Replikation und der DNA-Reparatur beeinträchtigen könnten. Darüber hinaus wurde über die nukleäre Translokation (Transport durch die Kernporen des Zellkerns) von GAPDH bei der Parkinson-Krankheit (PD) berichtet und mehrere anti-apoptotische PD-Medikamente wie Rasagilin wirken, indem sie die nukleäre Translokation von GAPDH verhindern. Es wird vermutet, dass der Hypometabolismus (erniedrigte Stoffwechselrate) einen Beitrag zur Parkinson-Krankheit leistet. Die genauen Mechanismen, die der Beteiligung von GAPDH an neurodegenerativen Erkrankungen zugrunde liegen, müssen jedoch noch geklärt werden.[30] Der SNP rs3741916 im 5′-UTR des GAPDH-Gens kann mit der late-onset Alzheimer-Krankheit assoziiert sein.[31]
Gene, Proteine und Metaboliten sind zu den jeweiligen Artikeln verlinkt. Der Stoffwechselweg kann bei WikiPathways bearbeitet werden:
Weblinks
Einzelnachweise
- C. Tristan, N. Shahani, T. W. Sedlak, A. Sawa: The diverse functions of GAPDH: views from different subcellular compartments. In: Cellular signalling. Band 23, Nummer 2, Februar 2011, S. 317–323, doi:10.1016/j.cellsig.2010.08.003, PMID 20727968, PMC 3084531 (freier Volltext) (Review).
- C. Nicholls, H. Li, J. P. Liu: GAPDH: a common enzyme with uncommon functions. In: Clinical and experimental pharmacology & physiology. Band 39, Nummer 8, August 2012, S. 674–679, doi:10.1111/j.1440-1681.2011.05599.x, PMID 21895736 (Review).
- Donald Voet, Judith G. Voet, Charlotte W. Pratt: Fundamentals of Biochemistry. Life at the Molecular Level. 5. Auflage. John Wiley & Sons, Hoboken, NJ 2016, ISBN 978-1-118-91840-1, S. 489 (eingeschränkte Vorschau in der Google-Buchsuche).
- Acetylphosphat, Lexikon der Biologie; Acetylphosphat, Lexikon der Chemie. Auf spektrum.de.
- David L. Nelson, Albert L. Lehninger, Michael M. Cox: Lehninger Principles of Biochemistry. 5. Auflage. W. H. Freeman, 2008, ISBN 978-1-4292-0892-5, S. 530.
- Stryer, Lubert: Biochemie, Spektrum der Wissenschaft Verlag, 4. Auflage, Heidelberg 1996, S. 526.
- Jeremy M. Berg, John L. Tymoczko, Lubert Stryer, Gregory J. Gatto jr.: Stryer Biochemie. Springer-Verlag, 2014, ISBN 978-3-8274-2988-9, S. 465–466 (eingeschränkte Vorschau in der Google-Buchsuche).
- Florian Horn et al.: Biochemie des Menschen, Stuttgart 2003, ISBN 3-13-130883-4, S. 80.
- Jeremy M. Berg, John L. Tymoczko, Lubert Stryer, Gregory J. Gatto jr.: Stryer Biochemie. Springer-Verlag, 2014, ISBN 978-3-8274-2988-9, S. 470 (eingeschränkte Vorschau in der Google-Buchsuche).
- Jeremy M. Berg, John L. Tymoczko, Lubert Stryer, Gregory J. Gatto jr.: Stryer Biochemie. Springer-Verlag, 2014, ISBN 978-3-8274-2988-9, S. 465 (eingeschränkte Vorschau in der Google-Buchsuche).
- E. Duée, L. Olivier-Deyris, E. Fanchon, C. Corbier, G. Branlant, O. Dideberg: Comparison of the structures of wild-type and a N313T mutant of Escherichia coli glyceraldehyde 3-phosphate dehydrogenases: implication for NAD binding and cooperativity. In: Journal of molecular biology. Band 257, Nummer 4, April 1996, S. 814–838, doi:10.1006/jmbi.1996.0204, PMID 8636984.
- Jeremy M. Berg, John L. Tymoczko, Lubert Stryer, Gregory J. Gatto jr.: Stryer Biochemie. Springer-Verlag, 2014, ISBN 978-3-8274-2988-9, S. 464–465 (eingeschränkte Vorschau in der Google-Buchsuche).
- M. E. Campanella, H. Chu, P. S. Low: Assembly and regulation of a glycolytic enzyme complex on the human erythrocyte membrane. In: Proceedings of the National Academy of Sciences. Band 102, Nummer 7, Februar 2005, S. 2402–2407, doi:10.1073/pnas.0409741102, PMID 15701694, PMC 549020 (freier Volltext).
- M. A. Sirover: Structural analysis of glyceraldehyde-3-phosphate dehydrogenase functional diversity. In: The international journal of biochemistry & cell biology. Band 57, Dezember 2014, S. 20–26, doi:10.1016/j.biocel.2014.09.026, PMID 25286305, PMC 4268148 (freier Volltext) (Review).
- S. Kumar, N. Sheokand, M. A. Mhadeshwar, C. I. Raje, M. Raje: Characterization of glyceraldehyde-3-phosphate dehydrogenase as a novel transferrin receptor. In: The international journal of biochemistry & cell biology. Band 44, Nummer 1, Januar 2012, S. 189–199, doi:10.1016/j.biocel.2011.10.016, PMID 22062951.
- E. A. Sweeny, A. B. Singh, R. Chakravarti, O. Martinez-Guzman, A. Saini, M. M. Haque, G. Garee, P. D. Dans, L. Hannibal, A. R. Reddi, D. J. Stuehr: Glyceraldehyde-3-phosphate dehydrogenase is a chaperone that allocates labile heme in cells. In: Journal of Biological Chemistry. Band 293, Nummer 37, 09 2018, S. 14557–14568, doi:10.1074/jbc.RA118.004169, PMID 30012884, PMC 6139559 (freier Volltext).
- L. Zheng, R. G. Roeder, Y. Luo: S phase activation of the histone H2B promoter by OCA-S, a coactivator complex that contains GAPDH as a key component. In: Cell. Band 114, Nummer 2, Juli 2003, S. 255–266, doi:10.1016/s0092-8674(03)00552-x, PMID 12887926.
- M. R. Hara, N. Agrawal, S. F. Kim, M. B. Cascio, M. Fujimuro, Y. Ozeki, M. Takahashi, J. H. Cheah, S. K. Tankou, L. D. Hester, C. D. Ferris, S. D. Hayward, S. H. Snyder, A. Sawa: S-nitrosylated GAPDH initiates apoptotic cell death by nuclear translocation following Siah1 binding. In: Nature cell biology. Band 7, Nummer 7, Juli 2005, S. 665–674, doi:10.1038/ncb1268, PMID 15951807.
- M. R. Hara, B. Thomas, M. B. Cascio, B. I. Bae, L. D. Hester, V. L. Dawson, T. M. Dawson, A. Sawa, S. H. Snyder: Neuroprotection by pharmacologic blockade of the GAPDH death cascade. In: Proceedings of the National Academy of Sciences. Band 103, Nummer 10, März 2006, S. 3887–3889, doi:10.1073/pnas.0511321103, PMID 16505364, PMC 1450161 (freier Volltext).
- A. R. Agarwal, L. Zhao, H. Sancheti, I. K. Sundar, I. Rahman, E. Cadenas: Short-term cigarette smoke exposure induces reversible changes in energy metabolism and cellular redox status independent of inflammatory responses in mouse lungs. In: American Journal of Physiology – Lung Cellular and Molecular Physiology. Band 303, Nummer 10, November 2012, S. L889–L898, doi:10.1152/ajplung.00219.2012, PMID 23064950.
- M. Ralser, M. M. Wamelink, A. Kowald, B. Gerisch, G. Heeren, E. A. Struys, E. Klipp, C. Jakobs, M. Breitenbach, H. Lehrach, S. Krobitsch: Dynamic rerouting of the carbohydrate flux is key to counteracting oxidative stress. In: Journal of biology. Band 6, Nummer 4, Dezember 2007, S. 10, doi:10.1186/jbiol61, PMID 18154684, PMC 2373902 (freier Volltext).
- Bergmeyer, Hans Ulrich: Methoden der enzymatischen Analyse Band 1, Verlag Chemie, 3. Auflage, Weinheim 1974, S. 115.
- Stryer, Lubert: Biochemie, Spektrum der Wissenschaft Verlag, 4. Auflage, Heidelberg 1996, S. 527.
- E. J. Tisdale, C. R. Artalejo: A GAPDH mutant defective in Src-dependent tyrosine phosphorylation impedes Rab2-mediated events. In: Traffic. Band 8, Nummer 6, Juni 2007, S. 733–741, doi:10.1111/j.1600-0854.2007.00569.x, PMID 17488287, PMC 3775588 (freier Volltext).
- R. D. Barber, D. W. Harmer, R. A. Coleman, B. J. Clark: GAPDH as a housekeeping gene: analysis of GAPDH mRNA expression in a panel of 72 human tissues. In: Physiological genomics. Band 21, Nummer 3, Mai 2005, S. 389–395, doi:10.1152/physiolgenomics.00025.2005, PMID 15769908.
- R. T. Piszczatowski, B. J. Rafferty, A. Rozado, S. Tobak, N. H. Lents: The glyceraldehyde 3-phosphate dehydrogenase gene (GAPDH) is regulated by myeloid zinc finger 1 (MZF-1) and is induced by calcitriol. In: Biochemical and biophysical research communications. Band 451, Nummer 1, August 2014, S. 137–141, doi:10.1016/j.bbrc.2014.07.082, PMID 25065746.
- D. Ramos, A. Pellín-Carcelén, J. Agustí, A. Murgui, E. Jordá, A. Pellín, C. Monteagudo: Deregulation of glyceraldehyde-3-phosphate dehydrogenase expression during tumor progression of human cutaneous melanoma. In: Anticancer Research. Band 35, Nummer 1, Januar 2015, S. 439–444, PMID 25550585.
- D. Wang, D. R. Moothart, D. R. Lowy, X. Qian: The expression of glyceraldehyde-3-phosphate dehydrogenase associated cell cycle (GACC) genes correlates with cancer stage and poor survival in patients with solid tumors. In: PLOS ONE. Band 8, Nummer 4, 2013, S. e61262, doi:10.1371/journal.pone.0061262, PMID 23620736, PMC 3631177 (freier Volltext).
- M. Phadke, N. Krynetskaia, A. Mishra, E. Krynetskiy: Accelerated cellular senescence phenotype of GAPDH-depleted human lung carcinoma cells. In: Biochemical and biophysical research communications. Band 411, Nummer 2, Juli 2011, S. 409–415, doi:10.1016/j.bbrc.2011.06.165, PMID 21749859, PMC 3154080 (freier Volltext).
- J. L. Mazzola, M. A. Sirover: Alteration of intracellular structure and function of glyceraldehyde-3-phosphate dehydrogenase: a common phenotype of neurodegenerative disorders? In: Neurotoxicology. Band 23, Nummer 4–5, Oktober 2002, S. 603–609, PMID 12428732 (Review).
- M. Allen, C. Cox, O. Belbin, L. Ma, G. D. Bisceglio, S. L. Wilcox, C. C. Howell, T. A. Hunter, O. Culley, L. P. Walker, M. M. Carrasquillo, D. W. Dickson, R. C. Petersen, N. R. Graff-Radford, S. G. Younkin, N. Ertekin-Taner: Association and heterogeneity at the GAPDH locus in Alzheimer's disease. In: Neurobiology of aging. Band 33, Nummer 1, Januar 2012, S. 203.e25–203.e33, doi:10.1016/j.neurobiolaging.2010.08.002, PMID 20864222, PMC 3017231 (freier Volltext).