Autographiviridae
Die Autographiviridae (veraltet auch Autographivirinae, T7-Supergruppe, englisch T7 supergroup genannt) sind eine Familie von Viren in der Ordnung Caudovirales – sie wurden dort früher als Unterfamilie Autographivirinae in der Familie Podoviridae geführt. Ihre natürlichen Wirte sind Bakterien. Es gibt derzeit 40 Arten (Spezies) in dieser Unterfamilie, die meisten davon sind einer von sieben Gattungen zugewiesen.[2][3]
| Autographiviridae | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|

Escherichia-Virus T7 | ||||||||||||||
| Systematik | ||||||||||||||
| ||||||||||||||
| Taxonomische Merkmale | ||||||||||||||
| ||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||
| Autographiviridae | ||||||||||||||
| Links | ||||||||||||||
|
Forschungsgeschichte
Seit den 1990er Jahren hat sich der Begriff T7-Supergruppe für die wachsende Gruppe der mit dem Coliphagen T7 verwandten Bakteriophagen der Familie Podoviridae etabliert. Salmonella virus SP6 (alias Enterobacteriaceae-Phage SP6) und Escherichia virus K1-5 (alias Enterobacteriaceae-Phage K1-5) wurden als erste als eine weitläufig verwandte Untergruppe (die heutige Gattung Zindervirus) dieser ‚T7-Supergruppe‘ betrachtet.[4] Das Pseudomonas-Virus phiKMV (alias Bakteriophage phiKMV) zeigte auf der Ebene der Genomorganisation ebenfalls Gemeinsamkeiten. Basierend auf den verfügbaren morphologischen und proteomischen Daten wurde diese Virusklade als eine Unterfamilie in der Familie Podoviridae etabliert.
Etymologie
Der Name der Unterfamilie Autographiviridae kommt von griechisch αὐτο-γράφειν (aúto-gráphein, selbst-schreibend) und bezieht darauf, dass diese Phagen ihre eigene RNA-Polymerase kodieren, also selbst-transkribierend sind; dies ist ein gemeinsames Merkmal all ihrer Mitglieder.
Aufbau

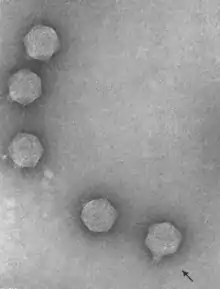
Die Virusteilchen (Virionen) der Autographiviridae sind nicht umhüllt mit ikosaedrischem Kopf-Schwanz-Aufbau und eine Symmetrie (Triangulationszahl) T=7. Der Durchmesser beträgt ca. 60 nm. Das Genom ist linear und hat eine Länge von ca. 40–42 kb.[2]
Vermehrungszyklus
Die Virusreplikation geschieht im Zytoplasma. Das Virus tritt durch Lyse (Auflösung) der Wirtszelle vermöge Holin-, Endolysin- bzw. Spanin-Proteinen aus dieser aus. Die Übertragung geschieht durch passive Diffusion.[2]
Systematik
Die folgende Liste führt die Spezies in der Unterfamilie Autographiviridae auf gemäß der ICTV Master Species List (MSL) #35 2019 v1:[5]
Familie Autographiviridae (veraltet: Autographivirinae, T7-Supergruppe)
- Gattung Daemvirus
- Gattung Friunavirus (veraltet Fri1virus)
- Spezies Acinetobacter-Virus AB3
- Spezies Acinetobacter-Virus Abp1
- Spezies Acinetobacter-Virus Fri1 (*)
- Spezies Acinetobacter-Virus IME200
- Spezies Acinetobacter-Virus PD6A3
- Spezies Acinetobacter-Virus PDAB9
- Spezies Acinetobacter-Virus phiAB1
- Gattung Pettyvirus
- Spezies Acinetobacter-Virus Petty (*)
- Gattung Gutovirus
- Spezies Vibrio-Virus Vc1 (*)
- Gattung Kaohsiungvirus
- Spezies Vibrio-Virus A318 (*)
- Gattung Murciavirus
- Spezies Marinomonas-Virus CB5A (*)
- Gattung Trungvirus
- Spezies Vibrio-Virus VEN (*)
- Gattung Uliginvirus
- Spezies Pseudomonas-Virus uligo (*)
- Gattung Gutovirus
- Gattung Kantovirus
- Spezies Pseudomonas-Virus C171 (*)
- Gattung Kotilavirus
- Spezies Pectobacterium-Virus PP16 (*)
- Gattung Phimunavirus (*)
- Spezies Pectobacterium-Virus fM1 (*)
- Gattung Stompvirus
- Spezies Dickeya-Virus BF25-12 (*)
- Gattung Kantovirus
- Gattung Kirikabuvirus
- Spezies Pseudomonas-Virus NV3 (*)
- Gattung Phikmvvirus (veraltet Phikmvlikevirus, PhiKMV-like viruses, PhiKMV-ähnliche Viren)
- Spezies Pantoea-Virus Limelight (mit Pantoea-Phage Limelight)
- Spezies Pantoea-Virus Limezero (mit Pantoea-Phage Limezero)
- Spezies Pseudomonas-Virus LKA1 (mit Pseudomonas-Phage LKA1)
- Spezies Pseudomonas-Virus phiKMV (*, mit Pseudomonas-Phage phiKMV)
- Gattung Stubburvirus
- Spezies Pseudomonas-Virus LKA1 (*)
- Gattung Tunggulvirus (ICTV-Fehlschreibung Tunggulviirus korrigiert nach den ICTV-Regeln für Gattungsnamen)
- Spezies Pseudomonas-Virus f2 (*)
- Gattung Kirikabuvirus
- Gattung Aerosvirus
- Spezies Aeromonas-Virus AS7 (*, mit Aeromonas-Phage phiAS7)[8]
- Gattung Aghbyvirus
- Spezies Yersinia-Virus ISAO8 (*)
- Gattung Ahphunavirus
- Spezies Aeromonas-Virus Ahp1 (*)
- Gattung Cronosvirus
- Spezies Cronobacter-Virus GAP227 (*, mit Cronobacter-sakazakii-Phage vB_CsaP_GAP227)[8]
- Gattung Panjvirus
- Spezies Salmonella-Virus Spp16 (*)
- Gattung Pienvirus
- Spezies Yersinia-Virus R8-01 (*, mit Yersinia-Phage ϕR8-01)[8]
- Gattung Pokrovskaiavirus
- Spezies Yersinia-Virus Phi80-18 (*, mit Yersinia-Phage ϕ80-18)[8]
- Gattung Wanjuvirus (*)
- Gattung Aerosvirus
- Gattung Acadevirus
- Spezies Proteus-Virus PM85 (*)
- Gattung Axomammavirus (*)
- Spezies Pectobacterium-Virus PP1 (*)
- Gattung Eracentumvirus
- Spezies Erwinia-Virus Era103 (*)
- Gattung Tuodvirus
- Spezies Lelliottia-Virus phD2B (*)
- Gattung Vectrevirus
- Spezies Escherichia-Virus VEc3 (*)
- Gattung Zindervirus (veraltet Sp6virus, Sp6likevirus, SP6-ähnliche Viren)
- Spezies Erwinia-Virus Era103 (mit Erwinia-amylovora-Phage Era103)
- Spezies Escherichia-Virus K5 (mit Enterobacteria-Phage K5)
- Spezies Escherichia-Virus K1-5 (mit Enterobacteria-Phage K1-5)
- Spezies Escherichia-Virus K1E (mit Enterobacteria-Phage K1E)
- Spezies Salmonella-Virus SP6 (*, mit Enterobacteria-Phage SP6)
- Gattung Acadevirus
- Gattung Ampunavirus
- Spezies Burkholderia-Virus BpAMP1 (*)
- Gattung Higashivirus
- Spezies Ralstonia-Virus RSB1 (*)
- Gattung Mguuvirus
- Spezies Burkholderia-Virus JG068 (*)
- Gattung Risjevirus
- Spezies Ralstonia-Virus RSJ2 (*)
- Gattung Sukuvirus
- Spezies Ralstonia-Virus RSPII1 (*)
- Gattung Ampunavirus
- Gattung Bucovirus
- Spezies Shigella-Virus Buco (*)
- Gattung Drulisvirus (veraltet Kp34virus)
- Spezies Klebsiella-Virus F19
- Spezies Klebsiella-Virus K244
- Spezies Klebsiella-Virus Kp2
- Spezies Klebsiella-Virus KP34 (*)
- Spezies Klebsiella-Virus KpV41
- Spezies Klebsiella-Virus KpV71
- Spezies Klebsiella-Virus KpV475
- Spezies Klebsiella-Virus SU503
- Spezies Klebsiella-Virus SU552A
- Gattung Koutsourovirus
- Spezies Enterobacter-Virus KDA1 (*)
- Gattung Novosibovirus
- Spezies Proteus-Virus PM16 (*)
- Gattung Bucovirus
- Gattung Aarhusvirus
- Spezies Dickeya-Virus Dagda (*)
- Gattung Apdecimavirus
- Spezies Yersinia-Virus AP10 (*)
- Gattung Berlinvirus
- Spezies Kluyvera-Virus Kvp1 (mit Kluyvera-Phage Kvp1)
- Spezies Yersinia-Virus Berlin (*)
- Gattung Caroctavirus
- Spezies Citrobacter-Virus CR8 (*)
- Gattung Chatterjeevirus
- Spezies Vibrio-Virus N4 (*)
- Gattung Eapunavirus
- Spezies Enterobacter-Virus Eap1 (*)
- Gattung Elunavirus
- Gattung Foetvirus
- Spezies Escherichia-Virus SRT7 (*)
- Gattung Aarhusvirus
- Gattung Ghunavirus
- Spezies Pseudomonas-Virus gh1 (*, mit Pseudomonas-Phage gh-1)
- Gattung Helsettvirus
- Spezies Yersinia-Virus fPS9 (*)
- Gattung Jarilovirus
- Spezies Pectobacterium-Virus Jarilo (*)
- Gattung Kayfunavirus
- Spezies Escherichia-Virus K1F (*)
- Gattung Minipunavirus
- Spezies Morganella-Virus MmP1 (*)
- Gattung Ningirsuvirus
- Spezies Dickeya-Virus Ninurta (*)
- Gattung Pektosvirus
- Spezies Pectobacterium-Virus PP81 (*)
- Gattung Phutvirus
- Spezies Pseudomonas-Virus PPpW4 (*)
- Gattung Pifdecavirus
- Spezies Pseudomonas-Virus Pf10 (*)
- Gattung Pijolavirus
- Spezies Pseudomonas-Virus PspYZU08 (*)
- Gattung Przondovirus (veraltet Kp32virus)
- Spezies Escherichia-Virus K30
- Spezies Klebsiella-Virus K5
- Spezies Klebsiella-Virus K11
- Spezies Klebsiella-Virus Kp1
- Spezies Klebsiella-Virus KP32 (*)
- Spezies Klebsiella-Virus KpV289
- Gattung Teetrevirus
- Spezies Escherichia-Virus T3 (*)
- Gattung Ghunavirus
- Gattung Teseptimavirus (veraltet T7virus, T7likevirus, T7-ähnliche Viren)
- Spezies Escherichia-Virus T7 (*, mit Enterobacteria-Phage T7)
- Spezies Escherichia-Virus 13a
- Spezies Escherichia-Virus 64795ec1
- Spezies Escherichia-Virus C5
- Spezies Escherichia-Virus CICC80001
- Spezies Escherichia-Virus Ebrios
- Spezies Escherichia-Virus EG1
- Spezies Escherichia-Virus HZ2R8
- Spezies Escherichia-Virus HZP2
- Spezies Escherichia-Virus N30
- Spezies Escherichia-Virus NCA
- Spezies Escherichia-Virus T7
- Spezies Salmonella-Virus 3A8767
- Spezies Salmonella-Virus Vi06
- Spezies Stenotrophomonas-Virus IME15
- Spezies Yersinia-Virus YpPY
- Spezies Yersinia-Virus YpsPG
- Gattung Teseptimavirus (veraltet T7virus, T7likevirus, T7-ähnliche Viren)
- Kandidaten (Vorschläge nach NCBI, wo nicht anders vermerkt. Stand: 7. Februar 2021)[10]
- Spezies Aeromonas-Phage PZL-Ah1
- Spezies Enterobacteria-Phage W31
- Spezies Enterococcus-Phage EFA-2
- Spezies Escherichia-Phage 13a
- Spezies Escherichia-Phage JB01
- Spezies Escherichia-Phage N13
- Spezies Escherichia-Phage vB_EcoP_PHB20
- Spezies Bacteriophage PhiI (alias Phage PhiI)[11]
- Spezies Prochlorococcus-Virus 4f
- Spezies Prochlorococcus-Virus 5e
- Spezies Prochlorococcus-Virus 5f
- Spezies Prochlorococcus-Virus 6b
- Spezies Prochlorococcus-Virus 6ed6p
- Spezies Prochlorococcus-Virus 7g
- Spezies Prochlorococcus-Virus d67f2
- Spezies Pseudomonas-Phage phiPLS27
- Spezies Pseudomonas-Virus Pf1 ERZ-2017
- Spezies Salmonella-Phage C2
- Spezies Serratia-Phage Pila[12]
- Spezies „Synechococcus-Virus 11bc6“
- Spezies „Synechococcus-Virus 11ec6“
- Spezies „Synechococcus-Virus 2fc6“
- Spezies „Synechococcus-Virus 4dc“
- Spezies „Synechococcus-Virus 5gcp“
- Spezies „Synechococcus-Virus 6bc6“
- Spezies „Synechococcus-Virus 7dc6“
- Spezies „Synechococcus-Virus 9ec6“
- Spezies „Synechococcus-Virus 9ecp“
- Spezies „Synechococcus-Virus c7e4“
- Spezies T7-like bacteriophage Eptesicus fuscus/P1/IT/USA/2009
- Spezies Teseptimavirus S2B
- Spezies Yersinia-Phage phiA1122
- Spezies Yersinia-Phage phiR3
- Spezies Yersinia-Phage R
- Spezies Yersinia-Phage Y
- Spezies Yersinia-Phage YpP-R
- Gattung Troedvirus
- Spezies Pseudomonas-Virus Phi15 (*)
- Gattung Unyawovirus
- Spezies Pectobacterium-Virus DUPPII (*)
- Gattung Cyanopodovirus (Vorschlag, abgetrennt von Teseptimavirus, siehe unten)[13]
- Gattung Aegirvirus
- Spezies Synechococcus-Virus SCBP42 (*)
- Gattung Aqualcavirus
- Gattung Ashivirus
- Gattung Atuphduovirus
- Gattung Ayakvirus
- Gattung Ayaqvirus
- Gattung Banchanvirus
- Gattung Bifseptvirus
- Gattung Bonnellvirus
- Gattung Cheungvirus
- Gattung Chosvirus
- Gattung Cuernavacavirus
- Gattung Cyclitvirus
- Gattung Ermolevavirus
- Gattung Foturvirus
- Gattung Foussvirus
- Gattung Fussvirus
- Gattung Gajwadongvirus
- Gattung Gyeongsanvirus
- Gattung Igirivirus
- Gattung Jalkavirus
- Gattung Jiaoyazivirus
- Gattung Kafavirus
- Gattung Kajamvirus
- Gattung Kakivirus
- Gattung Kalppathivirus
- Gattung Kelmasvirus
- Gattung Kembevirus
- Gattung Krakvirus
- Gattung Lauvirus
- Gattung Limelightvirus
- Gattung Lingvirus
- Gattung Lirvirus
- Gattung Lullwatervirus
- Gattung Maculvirus
- Gattung Napahaivirus
- Gattung Nohivirus
- Gattung Oinezvirus
- Gattung Paadamvirus
- Gattung Pagavirus
- Gattung Pairvirus
- Gattung Pedosvirus
- Gattung Pekhitvirus
- Gattung Pelagivirus
- Gattung Percyvirus
- Gattung Piedvirus
- Gattung Podivirus
- Gattung Pollyceevirus
- Gattung Poseidonvirus
- Gattung Powvirus
- Gattung Pradovirus
- Spezies Xanthomonas-Virus f20
- Spezies Xanthomonas-Virus f30
- Spezies Xylella-Virus Prado (*)
- Gattung Qadamvirus
- Gattung Scottvirus
- Gattung Sednavirus
- Gattung Serkorvirus
- Gattung Sieqvirus
- Gattung Stompelvirus
- Gattung Stopalavirus
- Gattung Stopavirus
- Gattung Stupnyavirus
- Gattung Tangaroavirus
- Gattung Tawavirus
- Gattung Aegirvirus
- Gattung Tiamatvirus
- Gattung Tiilvirus
- Gattung Tritonvirus
- Gattung Voetvirus
- Gattung Votkovvirus
- Gattung Waewaevirus
- Gattung Wuhanvirus (wohl zu unterscheiden von SARS-CoV-2, dem Erreger von COVID-19)
- Spezies Pasteurella-Virus PHB01
- Spezies Pasteurella-Virus PHB02 (*)
- ohne Gattungszuweisung
Typusspezies sind mit ‚*‘ markiert.
Anmerkung: Eine vorgeschlagene Gruppe in dieser Familie sind die Cyanopodoviren (vorgeschlagene Gattung Cyanopodovirus), deren Ähnlichkeit zu Escherichia-Virus T7 eine Zuordnung zu den Autographiviridae (Unterfamilie Studiervirinae) nahelegt.[13][28]
Weblinks
- Autographiviridae. Viralzone.
- ICTV.
Einzelnachweise
- ICTV: ICTV Taxonomy history: Enterobacteria phage T4, EC 51, Berlin, Germany, July 2019; Email ratification March 2020 (MSL #35)
- Viral Zone. ExPASy, abgerufen am 29. Dezember 2018.
- ICTV: Virus Taxonomy. Abgerufen am 29. Dezember 2018.
- D. Scholl, J. Kieleczawa, P. Kemp, J. Rush, C. C. Richardson, C. Merril, S. Adhya, I. J. Molineux: Genomic analysis of bacteriophages SP6 and K1-5, an estranged subgroup of the T7 supergroup. In: Journal of Molecular Biology. Band 335, Nr. 5, 2004, S. 1151–1171, doi:10.1016/j.jmb.2003.11.035, PMID 14729334.
- ICTV: ICTV Master Species List 2019.v1, New MSL including all taxa updates since the 2018b release, March 2020 (MSL #35)
- Yunfen Hua, Tingting Luo, Yiqi Yang, Dong Dong, Rui Wang, Yanjun Wang, Mengsha Xu, Xiaokui Guo, Fupin Hu, Ping He: Phage Therapy as a Promising New Treatment for Lung Infection Caused by Carbapenem-Resistant Acinetobacter baumannii in Mice, in: Front. Microbiol., 9. Januar 2018; doi:10.3389/fmicb.2017.02659
- Natalia Bagińska, Anna Pichlak, Andrzej Górski1, Ewa Jończyk-Matysiak: Specific and Selective Bacteriophages in the Fight against Multidrug-resistant Acinetobacter baumannii (PDF) in: Virologica Sinica, Band 34, S. 347–357; doi:10.1007/s12250-019-00125-0, Tbl. 1
- R. Abbasifar, A. M. Kropinski, P. M. Sabour, H. W. Ackermann, A. Alanis Villa, A. Abbasifar, M. W. Griffiths: The Genome of Cronobacter sakazakii Bacteriophage vB_CsaP_GAP227 Suggests a New Genus within the Autographivirinae. In: Genome Announc. Band 1, Nr. 1, 2013, doi:10.1128/genomeA.00122-12, PMID 23409275, PMC 3569369 (freier Volltext).
- Mary Ann Liebert: New bacteriophage fully characterized and sequenced, auf: EurekAlert vom 27. Januar 2020, Quelle: Inc./Genetic Engineering News
- NCBI: unclassified Teseptimavirus
- NCBI: Phage PhiI (species)
- NCBI: Serratia phage Pila (species)
- S. J. Labrie, K. Frois-Moniz, M. S. Osburne, L. Kelly, S. E. Roggensack, M. B. Sullivan, G. Gearin, Q. Zeng, M. Fitzgerald, M. R. Henn, S. W. Chisholm: Genomes of marine cyanopodoviruses reveal multiple origins of diversity. In: Environ. Microbiol. Band 15, Nr. 5, 2013, S. 1356–1376, doi:10.1111/1462-2920.12053, PMID 23320838 (mit.edu [PDF]).
- Sijun Huang et al.: Temporal transcriptomes of a marine cyanopodovirus and its Synechococcus host during infection, in: MicrobiologyOpen, 30. Dezember 2020, doi:10.1002/mbo3.1150
- NCBI: Synechococcus phage S-SBP1 (species)
- NCBI: Synechococcus phage S-RIP2 (species)
- John H. Paul, Matthew B. Sullivan: Marine phage genomics: what have we learned? In: Curr. Op. in Biotechnology, Band 16, Nr. 3, Juni 2005, S. 299–307, doi:10.1016/j.copbio.2005.03.007, PMID 15961031, PMC 15961031 (freier Volltext), siehe Fig. 3 (Genomkarte)
- NCBI: Prochlorococcus virus PSSP7 (species)
- Preeti Gipson, Matthew L. Baker, Desislava Raytcheva, Cameron Haase-Pettingell, Jacqueline Piret, Jonathan A. King, Wah Chiu: Protruding knob-like proteins violate local symmetries in an icosahedral marine virus. In: Nature Communications. 5, Nr. 4278, 2. Juli 2014. doi:10.1038/ncomms5278., Corrigendum, in: Nature Communications, Band 6, Nr. 6040, 12. Januar 2015, doi:10.1038/ncomms7040.
- NCBI: Synechococcus virus P60 (species)
- John H. Paul, Matthew B. Sullivan: Marine phage genomics: what have we learned?, Curr. Op. in Biotechnology Band 16, Nr. 3, Juni 2005, S. 299–307, doi:10.1016/j.copbio.2005.03.007, PMID 15961031, PMC 15961031 (freier Volltext), siehe Fig. 1 (Genomkarte)
- Jacqueline Z.-M. Chan, Andrew D. Millard, Nicholas H. Mann, Hendrik Schäfer: Comparative genomics defines the core genome of the growing N4-like phage genus and identifies N4-like Roseophage specific genes (PDF; 3,9 MB) In: Frontiers in Microbiology, Band 5, Nr. 506, 10. Oktober 2014, doi:10.3389/fmicb.2014.00506
- NCBI: Synechococcus virus Syn5 (species)
- Desislava A. Raytcheva, Cameron Haase-Pettingell, Jacqueline Piret, Jonathan A. King: Two Novel Proteins of Cyanophage Syn5 Compose Its Unusual Horn Structure. In: ASM Journal of Virology. 88, Nr. 4, 15. Februar 2014, ISSN 0022-538X, S. 2047–2055. doi:10.1128/JVI.02479-13. PMID 24307583. PMC 3911526 (freier Volltext). Epub 31. Januar 2014, insbes. Fig. 1 (mit B: Schemazeichnung)
- Desislava A. Raytcheva, Cameron Haase-Pettingell, Jacgueline Piret, Jonathan A. King: Two Novel Proteins of Cyanophage Syn5 Compose Its Unusual Horn Structure, in: ASM Journals / Journal of Virology, Band 88, Nr. 4, 31. Januar 2014, doi:10.1128/JVI.02479-13
- NCBI: Roseobacter virus SIO1 (species)
- NCBI: Vibrio-Virus VpV262 (species)
- Stephen T. Abedon, Richard Calendar (Hrsg.): The Bacteriophages. 2. Auflage. Oxford University Press, 2005, ISBN 0-19-803385-0, S. 518f. (books.google.de)