Caudoviricetes
Caudoviricetes (von lateinisch cauda ‚Schwanz‘) ist eine Klasse von Viren, die Bakterien und Archaeen als Wirte nutzen und eine Kopf-Schwanz-Struktur besitzen, weshalb die Mitglieder der Caudoviricetes einerseits als Bakteriophagen klassifiziert, andererseits gelegentlich informell als Schwanzviren bezeichnet werden. Die Klasse umfasst derzeit (Stand 10. April 2021) nur eine einzige vom International Committee on Taxonomy of Viruses (ICTV) bestätigte Ordnung, Caudovirales. Diese Ordnung war 1998 von Hans-Wolfgang Ackermann aufgrund von EM-Aufnahmen der Virionen als Supergruppe aller geschwänzten Bakteriophagen vorgeschlagen worden, was in der Folge vom ICTV bestätigt wurde. Die klassische Unterteilung dieser Gruppe in Familien erfolgt ebenfalls hauptsächlich aufgrund der genauen Morphologie des am Kapsid sitzenden Schwanzstückes: Myoviridae (Morphotyp A: langes kontraktiles Schwanzstück), Siphoviridae (Morphotyp B: langes nicht-kontraktiles Schwanzstück) und Podoviridae (Morphotyp C: kurzes nicht-kontraktiles Schwanzstück). In zunehmendem Maße erlangen aber Gen- und Genom- bzw. Proteom-Vergleiche für die Taxonomie an Bedeutung. Es zeigte sich dabei, dass diese traditionellen morphologisch begründeten Familien nicht monophyletisch waren. Dies hatte zur Folge, dass Genomsequenzen (sog. Contigs) aus der Metagenomik im bestehenden System – mit fehlender Information über die Morphologie – nicht zugeordnet werden konnten.[4]
| Caudoviricetes | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|

Caudoviricetes | ||||||||||||
| Systematik | ||||||||||||
| ||||||||||||
| Taxonomische Merkmale | ||||||||||||
| ||||||||||||
| Wissenschaftlicher Name | ||||||||||||
| Caudoviricetes | ||||||||||||
| Links | ||||||||||||
|

von links nach rechts.
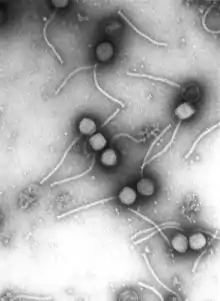
Um dieses Problem zu lösen, hatte das ICTV zunächst verschiedene Gruppen aus diesen herkömmlichen Familien – zunächst formell noch innerhalb der Ordnung Caudovirales – in neue geschaffene Familien verschoben, darunter die Ackermannviridae, Autographiviridae, Chaseviridae, Demerecviridae und Herelleviridae. Im März 2021 resultierten diese Bestrebungen dann in dem Vorschlag einer kompletten Reorganisation der Klasse unter Abschaffung der Ordnung Caudovirales; sowie der Auflösung der polphyletischen Familien (Myoviridae, Podoviridae und Siphoviridae), wobei diese durch neue monophyletische Familien ersetzt werden und nur noch informall als nicht-taxonomische Gruppen zur morphologischen Klassifizierung bestehen bleiben.[4]
Der Klade der aus der Metagenomik identifizierten crAss-like phages soll dabei dann, so der Vorschlag, selbst der Rang einer Ordnung „Crassvirales“ gegeben werden.[4]
Merkmale
Das Genom der Caudoviricetes ist unsegmentiert (monopartit), d. h. es besteht aus einem einzigen Molekül einer (gewöhnlich) doppelsträngigen DNA mit einer Größe von 18 bis 500 kBp (Kilobasenpaare). Es ist gewöhnlich linear, eine Ausnahme stellen die crAss-like phages (der vorgeschlagenen Ordnung „Crassvirales“) dar, die eine ringförmig geschlossene (zirkuläre) DNA besitzen.
Neben den üblichen Nukleotiden des genetischen Codes besitzen die Caudoviricetes auch modifizierte, z. B. glykosylierte Basen (d. h. mit angehängten zusätzlichen Zuckerresten) oder 5-Hydroxymethylcytosin an Stelle von Cytosin. Die DNA befindet sich in einem 45 bis 170 nm im Durchmesser großen ikosaedrischen Kapsid aus meist 72 Kapsomeren; das Schwanzstück kann bis zu 230 nm lang sein.
Das Genom kodiert für 27 bis über 600 Gene, deren Anordnung stark variiert. Das Genom kann von einzelsträngigen Lücken durchbrochen sein und kovalent gebundene terminale Proteine besitzen. Im Bakterium können die Caudoviricetes in das Nukleoid integrieren (Prophage) oder als lineares bzw. zirkuläres Plasmid in der Zelle verbleiben. Die Anzahl der isolierten Genome ist verhältnismäßig hoch, da die Caudoviricetes einem ständigen genetischen Austausch zwischen viralem und bakteriellem Genom wie auch dem horizontalen Austausch als Plasmid zwischen Bakterien unterliegen (siehe horizontaler Gentransfer). Es entsteht so eine Vielzahl von so genannten „Mosaiktypen“.
Verbreitung
Die Caudoviricetes sind stammesgeschichtlich sehr alte Viren mit großen Populationsvarianten und weltweiter Verbreitung. Man schätzt, dass sich etwa 1031 Virionen der Caudoviricetes in der Biosphäre befinden, die aneinandergelegt einer Strecke von 2×108 Lichtjahren entsprächen. Die Caudoviricetes machen den überwiegenden Anteil des Virioplanktons in den Meeren und Gewässern aus.
Systematik
Mit Stand Anfang Juni 2021 kennt das International Committee on Taxonomy of Viruses (ICTV) die folgende Familien und Unterfamilien (ergänzt um einige Vorschläge in doppelten Anführungszeichen), wobei etliche Gattungen (insbesondere wegen der oben beschriebenen Problematik) noch ohne Zuordnung sind:[2]
- Klasse Caudoviricetes
- Ordnung Caudovirales
- Familie Ackermannviridae[5]
- Unterfamilie Aglimvirinae
- Unterfamilie Cvivirinae
- ohne zugewiesene Unterfamilie (7 Gattungen)
- Familie Autographiviridae
- Unterfamilie Beijerinckvirinae
- Unterfamilie Colwellvirinae
- Unterfamilie Corkvirinae
- Unterfamilie Krylovirinae
- Unterfamilie Melnykvirinae
- Unterfamilie Molineuxvirinae
- Unterfamilie Okabevirinae
- Unterfamilie Slopekvirinae
- Unterfamilie Studiervirinae
- ohne zugewiesene Unterfamilie (70 Gattungen)
- Familie Chaseviridae
- Familie Demerecviridae
- Unterfamilie Ermolyevavirinae
- Unterfamilie Markadamsvirinae
- Unterfamilie Mccorquodalevirinae
- ohne zugewiesene Unterfamilie (4 Gattungen)
- Familie Drexlerviridae
- Unterfamilie Braunvirinae
- Unterfamilie Rogunavirinae
- Unterfamilie Tempevirinae
- Unterfamilie Tunavirinae
- ohne zugewiesene Unterfamilie (9 Gattungen)
- Familie Guelinviridae
- Unterfamilie Denniswatsonvirinae
- ohne zugewiesene Unterfamilie (2 Gattungen)
- Familie Herelleviridae
- Unterfamilie Bastillevirinae
- Unterfamilie Brockvirinae
- Unterfamilie Jasinkavirinae
- Unterfamilie Spounavirinae
- Unterfamilie Twortvirinae
- ohne zugewiesene Unterfamilie (7 Gattungen, 1 Spezies)
- Familie Myoviridae
- Unterfamilie Emmerichvirinae
- Unterfamilie Eucampyvirinae
- Unterfamilie Gorgonvirinae
- Unterfamilie Ounavirinae
- Unterfamilie Peduovirinae
- Unterfamilie Tevenvirinae
- Unterfamilie Twarogvirinae
- Unterfamilie Vequintavirinae
- ohne zugewiesene Unterfamilie (153 Gattungen)
- Familie Podoviridae
- Unterfamilie Beephvirinae
- Unterfamilie Eekayvirinae
- Unterfamilie Sepvirinae
- ohne zugewiesene Unterfamilie (44 Gattungen, 1 Spezies)
- Familie Rountreeviridae
- Unterfamilie Rakietenvirinae
- Unterfamilie Sarlesvirinae
- ohne zugewiesene Unterfamilie (2 Gattungen)
- Familie Salasmaviridae
- Unterfamilie Northropvirinae
- Unterfamilie Picovirinae[7]
- Unterfamilie Tatarstanvirinae
- ohne zugewiesene Unterfamilie (4 Gattungen)
- Familie Schitoviridae[8][9][4]
- Unterfamilie Enquatrovirinae
- Unterfamilie Erskinevirinae
- Unterfamilie Fuhrmanvirinae
- Unterfamilie Humphriesvirinae
- Unterfamilie Migulavirinae
- Unterfamilie Pontosvirinae
- Unterfamilie Rhodovirinae
- Unterfamilie Rothmandenesvirinae
- ohne zugewiesene Unterfamilie (14 Gattungen)
- Familie Siphoviridae
- Unterfamilie Arquatrovirinae
- Unterfamilie Azeredovirinae
- Unterfamilie Bclasvirinae
- Unterfamilie Bronfenbrennervirinae
- Unterfamilie Chebruvirinae
- Unterfamilie Dclasvirinae
- Unterfamilie Deejayvirinae
- Unterfamilie Dolichocephalovirinae
- Unterfamilie Gochnauervirinae
- Unterfamilie Guernseyvirinae
- Unterfamilie Gutmannvirinae
- Unterfamilie Hendrixvirinae
- Unterfamilie Langleyhallvirinae
- Unterfamilie Mccleskeyvirinae
- Unterfamilie Mclasvirinae
- Unterfamilie Nclasvirinae
- Unterfamilie Nymbaxtervirinae
- Unterfamilie Pclasvirinae
- Unterfamilie Queuovirinae
- Unterfamilie Skryabinvirinae
- Unterfamilie Trabyvirinae
- Unterfamilie Tybeckvirinae
- ohne zugewiesene Unterfamilie (299 Gattungen)
- Familie Zobellviridae
- Unterfamilie Cobavirinae
- ohne zugewiesene Unterfamilie (6 Gattungen)
- Familie „Flandersviridae“[10]
- Familie „Gratiaviridae“[10]
- Familie „Quimbyviridae“[10]
- Familie „Saltoviridae“[11]
- ohne Familienzuordnung:
- Gattung Lilyvirus
- Gattung „Alteavirus“ (mögliche Schwesterklade der Schitoviridae)[8]
- Gattung „Chungbukvirus“[12] – war früher den Siphoviridae zugeordnet, weicht laut ICTV erheblich von diesen ab und hat Gemeinsamkeiten mit den Myoviridae; sie wird mit ihren Spezies vom ICTV aktuell nicht gelistet.
- Spezies „Streptococcus-Phage ω1“ (alias „Pneumococcus phage ω1“, auch „ω-1“)[15][16][17]
- „Escherichia-Phage P4“ ist ein Satellitenvirus, das den Coliphagen P2 (Myoviridae, Gattung Peduovirus) als Helfervirus benötigt, d. h. ein Satellitenphage.[18][19]
- Familie Ackermannviridae[5]
- Ordnung „Crassvirales“ – Klade „crAss-like phages“ mit 6 Familien und (mindestens) 10 Gattungen und zirkulärem Genom, ursprünglich als Mitglieder der Podoviridae vorgeschlagen.[20][21][22][4][23]
- Familie „Crevaviridae“
- Unterfamilie „Coarsevirinae“
- Unterfamilie „Doltivirinae“
- Familie „Intestiviridae“
- Unterfamilie „Churivirinae“
- Unterfamilie „Crudevirinae“
- Gattung „Carjivirus“
- Spezies „Carjivirus crAssphage“ (mit Phage cr5_ERR589774)[24][25][26]
- Gattung „Carjivirus“
- Unterfamilie „Obtuvirinae“
- Familie „Jelitoviridae“
- ohne zugewiesene Unterfamilie (16 Gattungen)
- Familie „Steigviridae“
- ohne zugewiesene Unterfamilie (21 Gattungen)
- Familie „Suoliviridae“
- Unterfamilie „Bearivirinae“
- Unterfamilie „Boorivirinae“
- Unterfamilie „Loutivirinae“
- Unterfamilie „Oafivirinae“
- Unterfamilie „Uncouvirinae“
- Familie „Tinaiviridae“
- ohne zugewiesene Unterfamilie (2 Gattungen)
- Familie „Crevaviridae“
- ohne weitere Zuordnung:
- Klade „Gubaphagen“: Camarillo-Guerrero, Almeida et al. beschreiben 2019/2020 die Ergebnisse ihrer Metagenomanalysen der menschlichen Darmflora hinsichtlich Bakteriophagen. Sie machen dabei eine neue Klade, genannt „Gubaphagen“ (englisch Gut Bacteroidales phage, „Gubaphage clade)“ (mit zwei Gattungen, G1 – infiziert Bacteroides und G2 – infiziert Parabacteroides) aus, die nach den crAssphagen die zweithäufigsten Viren (d. h. Bakteriophagen) in dieser Umgebung darstellen. Die Merkmale der Gubaphagen erinnern dabei an die von „p-crAssphage“.[27][7] Wegen der Ähnlichkeiten mit den crAssphagen sind dies wahrscheinlich ebenfalls Mitglieder der Caudoviricetes (oder gar „Crassvirales“?).
- Ordnung Caudovirales
Galerie
 Teseptimavirus, Autographiviridae
Teseptimavirus, Autographiviridae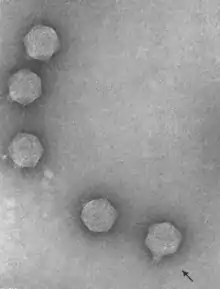 Pseudomonas-Virus gh1, Autographiviridae
Pseudomonas-Virus gh1, Autographiviridae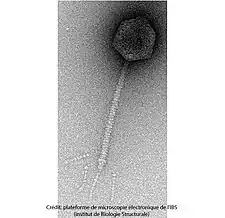 Tequintavirus, Demerecviridae
Tequintavirus, Demerecviridae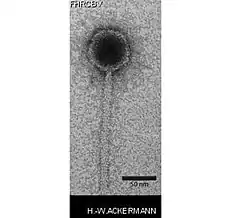 Tunavirus, Drexlerviridae
Tunavirus, Drexlerviridae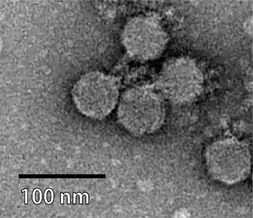 Clostridium-Phage CPS2, Guelinviridae
Clostridium-Phage CPS2, Guelinviridae Okubovirus, Herelleviridae
Okubovirus, Herelleviridae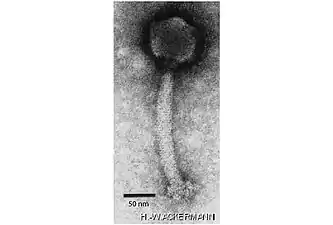 Twortvirus, Herelleviridae
Twortvirus, Herelleviridae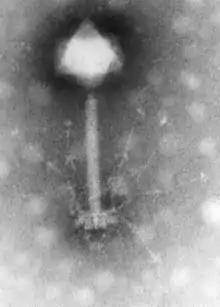 Synechococcus-Phage S-PM2, Myoviridae
Synechococcus-Phage S-PM2, Myoviridae Escherichia phage T4, Myoviridae
Escherichia phage T4, Myoviridae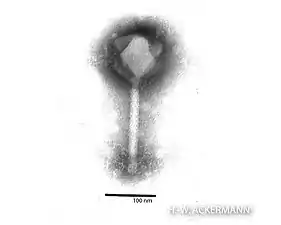 Phikzvirus, Myoviridae
Phikzvirus, Myoviridae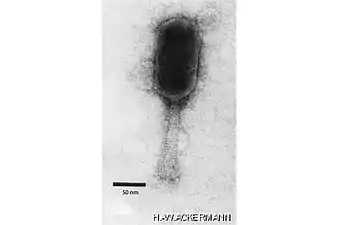 Schizotequatrovirus, Myoviridae
Schizotequatrovirus, Myoviridae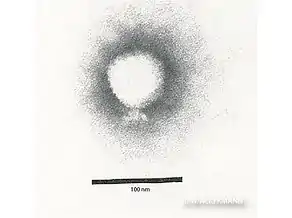 Lederbergvirus, Podoviridae
Lederbergvirus, Podoviridae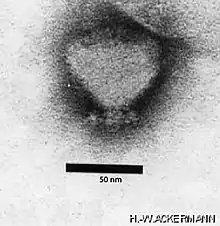 Uetakevirus, Podoviridae
Uetakevirus, Podoviridae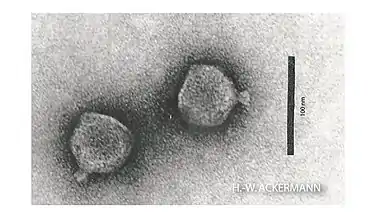 Bruynoghevirus, Podoviridae
Bruynoghevirus, Podoviridae Phage CSA13, Rountreeviridae
Phage CSA13, Rountreeviridae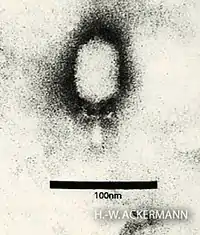 Salasvirus, Salasmaviridae
Salasvirus, Salasmaviridae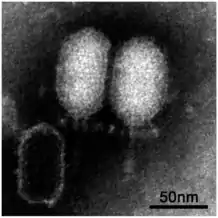 Bacillus-Virus Goe4, Salasmaviridae
Bacillus-Virus Goe4, Salasmaviridae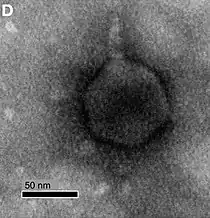 Roseovarius-Virus RPP1, Schitoviridae
Roseovarius-Virus RPP1, Schitoviridae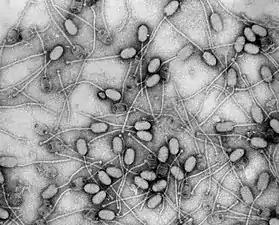 Staphylococcus-aureus-Phage 3A, Siphoviridae
Staphylococcus-aureus-Phage 3A, Siphoviridae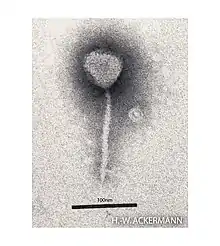 Escherichia-Phage λ, Siphoviridae
Escherichia-Phage λ, Siphoviridae Lentibacter-Virus ICBM1, Zobellviridae
Lentibacter-Virus ICBM1, Zobellviridae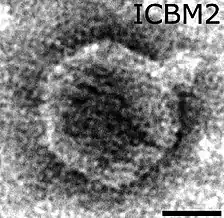 Lentibacter-Virus ICBM2, Zobellviridae
Lentibacter-Virus ICBM2, Zobellviridae
Literatur
- C. M. Fauquet, M. A. Mayo et al.: Eighth Report of the International Committee on Taxonomy of Viruses. San Diego / London 2004.
- Bernard N. Fields, David M. Knipe, Peter Howley (Hrsg.): Fields Virology. 4. Auflage. Philadelphia 2001.
Weblinks
Einzelnachweise
- ICTV: ICTV Master Species List 2019.v1, New MSL including all taxa updates since the 2018b release, March 2020 (MSL #35)
- ICTV: ICTV Master Species List 2020.v1, New MSL including all taxa updates since the 2019 release, March 2021 (MSL #36)
- NCBI: Bacillus phage Gamma (species)
- Dann Turner, Andrew M. Kropinski, Evelien M. Adriaenssens: A Roadmap for Genome-Based Phage Taxonomy, in: MDPI Viruses Band 13, Nr. 3, Section Bacterial Viruses, 18. März 2021, 506, doi:10.3390/v13030506
- SIB: Ackermannviridae, auf: ViralZone
- Evelien M. Adriaenssens, Mart Krupovic et al.: Taxonomy of prokaryotic viruses: 2018–2019 update from the ICTV Bacterial and Archaeal Viruses Subcommittee. In: Archives of Virology, 165, 11. März 2020, S. 1253–1260, doi:10.1007/s00705-020-04577-8, PDF (PDF)
- Luis Fernando Camarillo Guerrero: Integrative Analysis of the Human Gut Phageome Using a Metagenomics Approach. Doktorarbeit, Gonville & Caius College, University of Cambridge, August 2020, doi:10.17863/CAM.63973
- ICTV: Proposals ratification list 2021
- ICTV: Bacterial and archaeal virus proposals (zip): 2020.146B.R.Schitoviridae.xlsx
- Sean Benler, Natalya Yutin, Dmitry Antipov, Mikhail Raykov, Sergey Shmakov, Ayal B. Gussow, Pavel Pevzner, Eugene V. Koonin: Thousands of previously unknown phages discovered in whole-community human gut metagenomes (PDF) auf: bioRxiv, Preprint vom 7. Oktober 2020, doi:10.1101/2020.10.07.330464
- ICTV: 2018.139B.Ud.v1.Saltoviridae.xlsx, ICTV Proposals: Saltoviridae, vom 27. August 2018
- ICTV: ICTV Taxonomy history: Chungbukvirus, ICTV Taxonomy History
- NCBI: Pseudomonas virus Ptobp6g (species)
- Pseudomonas phage phiPto-bp6g (no rank) NCBI
- Jean-Gerard Tiraby, E. Tiraby, M. S. Fox: Pneumococcal bacteriophages, in: Virology Band 68, Dezember 1975, S. 566–569, doi:10.1016/0042-6822(75)90300-1, PMID 844
- Rubens López López: Streptococcus pneumoniae and its bacteriophages: one long argument. In: Int. Microbiol.. 7, Nr. 3, 2004, S. 163–71. PMID 15492930. freier Volltext (PDF) via Web-Archiv, 9. August 2017
- Rubens López, Ernesto García: Recent trends on the molecular biology of pneumococcal capsules, lytic enzymes, and bacteriophage, Oxford Academic FEMS Microbiology Reviews. Band 28, Nr. 5, 1. November 2004, S. 554–580, doi:10.1016/j.femsre.2004.05.002 (Freier Volltext)
- Renata Filipa Cruz de Matos: Enterococcus faecalis V583 prophages: Dynamic interactions and contribution to bacterial pathogenic traits (PDF; 10 MB) Dissertation, Universidade Nova de Lisboa (UNL), Juli 2013
- NCBI: Phage P4 satellite (no rank)
- crAss-like phages/viruses (clade) NCBI
- crAss-like phages. SIB. Double Strand DNA Viruses. ViralZone
- Eugene V. Koonin, Natalya Yutin: The crAss-like Phage Group: How Metagenomics Reshaped the Human Virome. In: Trends in Microbiology, Band 28, Nr. 5, 28. Februar/1. Mai 2020, S. 349–359, doi:10.1016/j.tim.2020.01.010
- A. N. Shkoporov, S. R. Stockdale, E. M. Adriaenssens, N. Yutin, E. V. Koonin, B. E. Dutilh, M. Krupovic, R. A. Edwards, I. Tolstoy, C. Hill: 2020.039B.Ud.v1.Crassvirales.docx, 2020.039B.Ud.v1.Crassvirales.xlsx, Proposal: Create one new order (Crassvirales) including six new families, ten new subfamilies, 78 new genera and 279 new species. 6. Dezember 2020
- NCBI: uncultured crAssphage (species)
- Ajeng K. Pramono, Hirokazu Kuwahara, Takehiko Itoh, Atsushi Toyoda, Akinori Yamada, Yuichi Hongoh: Discovery and Complete Genome Sequence of a Bacteriophage from an Obligate Intracellular Symbiont of a Cellulolytic Protist in the Termite Gut. In: Microbes and Environments. 32, Nr. 2, 2017, ISSN 1342-6311, S. 112–117. doi:10.1264/jsme2.ME16175. PMID 28321010. PMC 5478533 (freier Volltext).
- CrAssphage: Previously Unknown Ancient Gut Virus Lives in Half World’s Population, auf: sci-news vom 11. August 2014 (englisch)
- Luis Fernando Camarillo-Guerrero, Alexandre Almeida, Guillermo Rangel-Pineros, Robert D. Finn, Trevor D. Lawley: Massive expansion of human gut bacteriophage diversity. In: Cell Resource, Band 184, Nr. 4, S. 1098–1109.e9, 18. Februar 2021, doi:10.1016/j.cell.2021.01.029. PrePrint vom 3. September 2020: bioRxiv, Europe PMC, doi:10.1101/2020.09.03.280214. Dazu:
- Martin Vieweg: Tausende Virenarten der Darmflora entdeckt. wissenschaft.de, 18. Februar 2021 (deutsch)
- Daniel Lingenhöhl: Virobiom: Darm beherbergt zehntausende unbekannte Virenarten. spektrum.de vom 19. Februar 2021 (deutsch)
- Peter Dockrill: Scientists Find 140,000 Virus Species in The Human Gut, And Most Are Unknown. sciencealert, 28. Februar 2021 (englisch)
- Biologists Find Almost 143,000 Bacteriophage Species in Human Gut. sci-news, 19. Februar 2021 (englisch)
- Scientists identify more than 140,000 virus species in the human gut. ScienceDaily, 18. Februar 2021 (englisch)