Siphoviridae
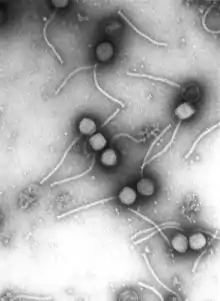
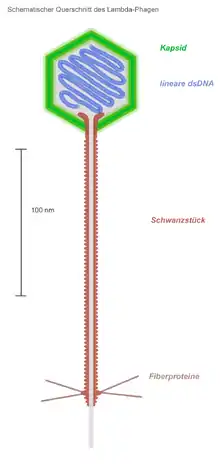
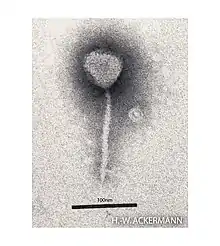
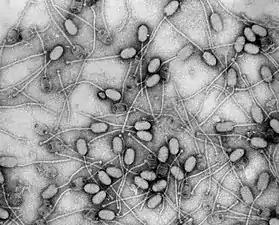
Die Virusfamilie Siphoviridae (griechisch σίφων siphon, deutsch ‚Wasserröhre‘) umfasst Viren der Ordnung Caudovirales mit einem linearen, doppelsträngigen DNA-Genom von ca. 22–121 kBp Länge und einem 50–60 nm im Durchmesser großen ikosaedrisches Kapsid mit einem 56–570 nm langen, nicht-kontraktilen Schwanzteil. Dieses Schwanzteil besteht aus übereinander gestapelten Scheiben von 7 bis 10 nm Durchmesser, die aus sechs identischen Untereinheiten aufgebaut sind.
| Siphoviridae | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ||||||||||||||
| Systematik | ||||||||||||||
| ||||||||||||||
| Taxonomische Merkmale | ||||||||||||||
| ||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||
| Siphoviridae | ||||||||||||||
| Links | ||||||||||||||
|
Die Siphoviridae haben Prokaryoten zum Wirt. Gewöhnlich sind dies Bakterien, was sie nicht-taxonomisch als Bakteriophagen klassifiziert. Es gibt aber auch Mitglieder, die Archaeen infizieren. Eine künstlerische Darstellung von Virusteilchen der Siphoviridae, eine Bakterienzelle angreifend, findet sich zusammen mit Details des Schwanzaufbaus bei EurekAlert (Nov. 2020).[5]
Die Gattungen innerhalb der Siphoviridae unterscheiden sich hinsichtlich der Organisation des Genoms, den Mechanismen der DNA-Verpackung und dem Vorhandensein einer DNA-Polymerase. Einige Gattungen (λ-ähnliche bis L5-ähnliche Viren) haben ein isometrisches Kapsid, bei den anderen ist das Kapsid langgestreckt und nicht-isometrisch.
Wichtige Spezies der Familie sind das Escherichia-Virus Lambda (Lambda-Phage, Gattung Lambdavirus), der Salmonella-Virus Chi (Chi-Phage, Gattung Chivirus), der Escherichia-Virus HK97 (HK97-Phage, Gattung Hendrixvirus), der Bacillus-Virus Gamma (Gamma-Phage, Gattung Wbetavirus)[1] und der „Enterobacteria-Phage Phi80“ (Phi80-Phage, Status zum 6. Februar 2021: Vorschlag ohne Gattungszuordnung)[6].
Im März 2021 wurde vorgeschlagen, die Familie mitsamt der Ordnung Caudovirales wegen fehlender Monophylie aufzulösen und durch neu zu schaffende Familien zu ersetzen, damit neue Ergebnisse aus der Metagenomik in die Taxonomie aufgenommen werden können.[7]
Systematik
Die Familie Siphoviridae umfasst nach dem ICTV (Stand 11. Juni 2021) folgende Gattungen und beispielhafte Spezies (veraltete Namen sind in Klammern als Aliase bzw. mit Zusatz „ehem.“ angegeben):[4]
Familie Siphoviridae
- Genus Arequatrovirus (früher R4virus)
- Spezies Streptomyces-Virus R4 (en. Streptomyces virus R4)
- Genus Camvirus
- Spezies Streptomyces-Virus phiCAM (en. Streptomyces virus phiCAM)
- Genus Likavirus
- Spezies Streptomyces-Virus Lika (en. Streptomyces virus Lika)
- Genus Acadianvirus
- Genus Coopervirus
- Genus Pegunavirus (ehem. Pg1virus oder Pgonelikevirus)
- Genus Pipefishvirus
- Genus Rosebushvirus
-
- Spezies Staphylococcus-Virus 13 (en. Staphylococcus virus 13)
- Spezies Staphylococcus-Virus 77 (en. Staphylococcus virus 77, Typus)
- Staphylococcus-Phage 77[11]
- Spezies Staphylococcus-Virus 108PVL (en. Staphylococcus virus 108PVL)
- Phage φPVL[11]
- Spezies „Staphylococcus-Phage phiN315“ (en. „Staphylococcus phage phiN315“)[15]
- Spezies „Staphylococcus-Phage Mu50A“ (en. „Staphylococcus phage Mu50A“)
- Spezies „Staphylococcus-Phage PVL“ (en. „Staphylococcus phage PVL“)[16]
- Genus Peeveelvirus
- Genus Brujitavirus
- Spezies Mycobacterium-Virus Babsiella (en. Mycobacterium virus Babsiella)
- Spezies Mycobacterium-Virus Brujita (en. Mycobacterium virus Brujita)
- Genus Hawkeyevirus
- Genus Plotvirus (ehem. Pbi1virus; Pbiunavirus, Pbiunalikevirus)
- Spezies , Mycobacterium virus PLot (alias Mycobacterium virus Plot, Mycobacterium phage PLot; Mycobacterium-Virus PBI1, en. Mycobacterium virus PBI1)[17] – beide Genera und Spezies zusammengefügt
- Genus Kenoshavirus
- Genus Secretariatvirus
- Genus Tanisvirus
- Genus Bertelyvirus
- Genus Colossusvirus
- Genus Poindextervirus
- Genus Shapirovirus
- Genus Dragolirvirus
- Genus Harrisonvirus
- Spezies Paenibacillus-Virus Harrison (en. Paenibacillus virus Harrison, Typus)
- Genus Vegasvirus
- Spezies Paenibacillus-Virus Vegas (en. Paenibacillus virus Vegas, Typus), mit Paenibacillus-Phage Diane, Hayley, Vadim und Vegas (Referenzstamm)[18]
- Genus Wanderervirus
- Genus Cornellvirus (alies Sp31virus)
- Spezies Salmonella-Virus SP31 (en. Salmonella virus SP31)
- Genus Jerseyvirus (ehem. Jerseylikevirus)
- Genus Kagunavirus (ehem. K1gvirus)
- Genus Carmenvirus
- Genus Pebcunavirus
- Genus Limdunavirus (ehem. Lmd1virus)
- Genus Unaquatrovirus (ehem. Una4virus)
- Spezies Leuconostoc-Virus 1A4 (en. Leuconostoc virus 1A4)
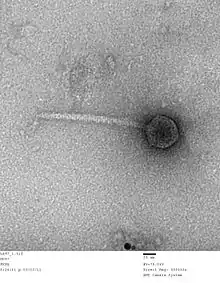
- Genus Byrnievirus
- Spezies Escherichia-Virus HK97 (Typus)
- Enterobacteria-Phage HK97[19]
- Genus Cuauhtlivirus
- Spezies Escherichia-Virus mEpX1
- Genus Kwaitsingvirus
- Spezies Escherichia-Virus HK446
- Spezies Escherichia-Virus HK544
- Genus Nochtlivirus
- Genus Saikungvirus
- Spezies Escherichia-Virus HK75
- Spezies Escherichia-Virus HK633
- Enterobacteria-Phage HK75
- Genus Shamshuipovirus
- Spezies Escherichia-Virus HK022
- Spezies Escherichia-Virus mEpX2
- Enterobacteria-Phage HK022
- Genus Wanchaivirus
- Spezies Escherichia-Virus HK106
- Spezies Escherichia-Virus mEp234
- Genus Wongtaivirus
- Spezies Escherichia-Virus HK542
- Genus Yautsimvirus
- Genus Getalongvirus
- Genus Horusvirus
- Genus Phistoryvirus
- Genus Limdunavirus
- Genus Unaquatrovirus
- Genus Bongovirus
- Genus Reyvirus (ehem. Reylikevirus)
- Genus Buttersvirus
- Genus Charlievirus (ehem. Charlielikevirus)
- Spezies Mycobacterium-Virus Charlie (en. Mycobacterium virus Charlie)[20] im „Cluster N“
- Genus Redivirus
- Genus Baxtervirus
- Genus Nymphadoravirus
- Genus Bignuzvirus (ehem. Bignuzlikevirus)
- Genus Fishburnevirus
- Genus Phayoncevirus
- Genus Amoyvirus
- Genus Nipunavirus (ehem. Np1virus)
- Genus Nonagvirus
- Genus Seuratvirus
- Genus Bembunaquatrovirus
- Genus Pushchinovirus
- Genus Jelitavirus
- Genus Slepowronvirus
- Genus Lenusvirus
- Genus Lidleunavirus
- Genus Maenadvirus
- Species „Lactobacillus-Phage P2“[21] (unterscheide: Lactococcus-Virus P2)

- Genus Fromanvirus (ehem. L5virus, L5likevirus, L5-ähnliche Viren), isometrisches Kapsid, 60 Spezies
- Spezies Mycobacterium-Virus Bxb1(mit Mykobakteriophage Bxb1)[20] im „Subcluster A1“
- Spezies Mycobacterium-Virus Bxz2
- Spezies Mycobacterium-Virus D29 (alias Mycobacterium-Phage D29, Jinga3000 oder Lakes)[20][23] im „Subcluster A2“
- Spezies Mycobacterium-Virus L5 (alias Mycobacterium-Phage L5)
- Spezies Mycobacterium-Virus Gladiator
- Spezies Mycobacterium-Virus Heldan[20][24][25] im „Subcluster A3“
- Spezies Mycobacterium-Virus Peaches[20] im „Subcluster A4“
- Spezies Mycobacterium-Virus U2
- Spezies „Mycobacterium-Phage BabyRay“[20][26]
- Spezies „Mycobacterium-Phage Benvolio“[27][28]
- Spezies „Mycobacterium-Phage Chupacabra“[27][29]
- Spezies „Mycobacterium-Phage D32“[30][31]
- Spezies „Mycobacterium-Phage DaVinci“[20]im „Subcluster A6“
- Spezies „Mycobacterium-Phage EagleEye“[20]im „Subcluster A16“
- Spezies „Mycobacterium-Phage Et2Brutus“[20]im „Subcluster A11“
- Spezies „Mycobacterium-Phage Fernando“[20]im „Subcluster A3“
- Spezies „Mycobacterium-Phage Fred313“[20][32] im „Subcluster A3“
- Spezies „Mycobacterium-Phage Isca“[20][33] im „Subcluster A3“
- Spezies „Mycobacterium-Phage Puppy“[20][34] im „Subcluster A3“
- Spezies „Mycobacterium-Phage TNguyen7“[20][35] im „Subcluster A3“
- Genus Lambdavirus (ehem. Lambdalikevirus, λ‐like phages, Lambda-ähnliche Viren), isometrisches Kapsid
- Spezies Escherichia-Virus DE3 (en. Escherichia virus DE3)
- Spezies Escherichia-Virus HK629 (en. Escherichia virus HK629)
- Spezies Escherichia-Virus HK630 (en. Escherichia virus HK630)
- Spezies Escherichia-Virus Lambda (en. Escherichia virus Lambda)
- Enterobacteria-Phage Lambda (ehem. Bakteriophage Lambda, Lambda-Phage oder Phage λ)
- Spezies „Actinophage RP3“
- Spezies „Actinophage 41C“
- Spezies „Bacillus-Phage Lurz2“ (en. „Bacillus phage Lurz2“)
- Spezies „Bacillus-Phage Lurz3“ (en. „Bacillus phage Lurz3“)
- Spezies „Bacillus-Phage rho11s“ (en. „Bacillus phage rho11s“)
- Spezies „Bacillus-Phage SPO2“ (en. „Bacillus phage SPO2“)
- Spezies „Bacillus-Phage SPR“ (en. „Bacillus phage SPR“)
- Spezies „Campylobacter-Phage B14“ (en. „Campylobacter phage B14“)
- Spezies „Corynephage beta“[36] (en. „Corynebacteriophage β“, „Corynebacterium diphtheriae virus“[37])[13][12][38][39][40]
- Spezies „Corynephage omega“ (en. „Corynebacteriophage ω“)[42]
- Phage omega (Phage ω)
- Spezies „Enterobacteria-Phage H-19B“ (en. „Enterobacteria phage H-19B“)[43]
- Genus Lomovskayavirus (ehem. Phic31virus, Phic3unalikevirus, PhiC31-ähnliche Viren, φC31-ähnliche Viren), nicht-isometrisches Kapsid
- Spezies Streptomyces-Virus phiBT1 (en. Streptomyces virus phiBT1)
- Spezies Streptomyces-Virus phiC31 (en. Streptomyces virus phiC31)
- Spezies „Streptomyces-Phage Shawty“ (en. „Streptomyces phage Shawty“)
- Genus Moineauvirus (ehem. Sfi21dt1virus, Sfi21dtunalikevirus, Sfi21-like phage)
- Spezies Streptococcus-Virus DT1 (en. Streptococcus virus DT1)
- Spezies Streptococcus-Virus Sfi19 (en. Streptococcus virus Sfi19)
- Spezies Streptococcus-Virus Sfi21 (en. Streptococcus virus Sfi21)
- Genus Psimunavirus (ehem. Psimunalikevirus, PsiM1-ähnliche Viren, ψM1-ähnliche Viren), nicht-isometrisches Kapsid, 1 Spezies[44]
- Spezies Methanobacterium-Virus psiM2 (alias ψM2‐like phage, Typus)[45]
- Methanobacterium-Phage PsiM2 (alias Methanobacterium-Phage ψM2, Archaeophage PsiM2, Phage ψM2)
- „Methanobacterium-Phage PsiM1“ (alias „Methanobacterium-Phage ψM1“, „Phage ψM1“, Genomsequenz nicht verfügbar, daher nicht mehr offizielle Typusspezies, PsiM2 rückt nach)[44]
- Genus Skunavirus (ehem. Sk1virus, Skunalikevirus, Sk1likevirus, Sk1-like phage, 936-Gruppe[46])
- Spezies Lactococcus-Virus 936 (en. Lactococcus virus 936)[47][48]
- Spezies Lactococcus-Virus P2 (en. Lactococcus virus P2[49])[50][46] – unterscheide: „Lactobacillus-Phage P2“
- Spezies Lactococcus-Virus sk1 (en. Lactococcus virus sk1)
- Spezies Streptomyces-Virus phiC31
- Streptomyces-Phage PhiC31 (alias Streptomyces-Phage φC31)
- Genus Brussowvirus (ehem. Sfi11virus, Sfi1unalikevirus, Sfi11-like phage)
- Spezies Streptococcus-Virus Sfi11 (en. Streptococcus virus Sfi11)
- Streptococcus-Phage Sfi11 (en. Streptococcus phage Sfi11)
- Genus Abbeymikolonvirus
- Genus Abidjanvirus (ehem. Ab18virus)
- Genus Agmunavirus
- Genus Aguilavirus
- Genus Ahduovirus
- Spezies Burkholderia-Virus AH2
- Genus Alachuavirus
- Genus Alegriavirus
- Genus Amigovirus
- Genus Anatolevirus
- Genus Andrewvirus
- Genus Andromedavirus (ehem. Andromedalikevirus)
- Genus Annadreamyvirus
- Genus Appavirus
- Genus Apricotvirus
- Genus Arawnvirus
- Genus Armstrongvirus
- Genus Ashduovirus
- Genus Attisvirus
- Genus Attoomivirus
- Genus Audreyjarvisvirus
- Genus Austintatiousvirus
- Genus Avanivirus
- Genus Bantamvirus
- Genus Barnyardvirus (ehem. Barnyardlikevirus)
- Genus Beceayunavirus
- Genus Beetrevirus
- Genus Behunavirus
- Genus Bendigovirus
- Genus Bernalvirus (ehem. Bernal13virus)
- Genus Betterkatzvirus
- Genus Bievrevirus
- Genus Bingvirus
- Genus Bowservirus
- Genus Bridgettevirus
- Genus Britbratvirus
- Genus Bronvirus (ehem. Bronlikevirus)
- Genus Camtrevirus
- Genus Casadabanvirus (ehem. D3112virus, D3112likevirus)
- Spezies Pseudomonas virus D3112 (Typus)
- Genus Cbastvirus (ehem. Cba13unalikevirus)
- Spezies Cellulophaga-Virus ST (Typus)
- Cellulophaga phage phiST
- Cellulophaga-Phage phi13:1
- Cellulophaga-Phage phi19:2
- Genus Cecivirus (ehem. Iebhlikevirus)
- Spezies Bacillus-Virus 250
- Bacillus-Phage 250
- Spezies Bacillus-Virus IEBH
- Bacillus-Phage IEBH
- Genus Ceduovirus (ehem. C2virus, C2likevirus, C2-ähnliche Viren, C2-Gruppe[48]), nicht-isometrisches Kapsid, 2 Spezies, etwa 3 vorgeschlagene, und ca. 200 Subtypen
- Spezies Lactococcus-Virus bIL67
- Lactococcus-Phage bIL67
- Spezies Lactococcus-Virus c2 (Typus)
- Lactococcus-Phage c2
- Genus Ceetrepovirus
- Spezies Corynebacterium-Virus C3PO (Typus)
- Spezies Corynebacterium-Virus Darwin
- Spezies Corynebacterium-Virus Zion
- Genus Cequinquevirus (ehem. C5virus, C5likevirus, C5-ähnliche Viren)
- Spezies Lactobacillus-Virus c5 (Typus)
- Lactobacillus-Phage c5
- Genus Chenonavirus (ehem. Che9cvirus, Che9clikevirus)
- Spezies Mycobacterium-Virus Che9c (Typus)[20] im „Subcluster I2“
- Genus Cheoctovirus (ehem. Che8virus, Che8likevirus)
- Spezies Mycobacterium-Virus Che8 (Typus)
- Spezies Mycobacterium-Virus Tweety[20] im „Subcluster E6“
- Genus Chivirus (ehem. Chilikevirus)
- Spezies Salmonella-Virus Chi (en. Salmonella virus Chi, Typus)
- Spezies Salmonella-Virus YSD1 (en. Salmonella virus YSD1, mit Salmonella phage YSD1)[8][53]
- Salmonella-Phage Chi (alias Phage χ)
- Spezies Salmonella-Virus FSLSP030
- Spezies Salmonella-Virus FSLSP088
- Spezies Salmonella-Virus iEPS5
- Spezies Salmonella-Virus SPN19
- Genus Chunghsingvirus
- Genus Cimpunavirus
- Genus Cinunavirus
- Genus Coetzeevirus (ehem. Phijl1virus, Phijlunalikevirus)
- Spezies Lactobacillus-Virus phiJL1 (Typus)
- Genus Colunavirus
- Genus Coralvirus
- Genus Corndogvirus (ehem. Corndoglikevirus)
- Genus Cornievirus
- Genus Coventryvirus
- Genus Cronusvirus
- Genus Cukevirus
- Spezies Mycobacterium virus Cuke (ehem. Mycobacterium-Phage Indlulamithi)
- Genus Daredevilvirus
- Genus Decurrovirus
- Genus Delepquintavirus
- Genus Demosthenesvirus
- Genus Detrevirus (ehem. D3virus, D3likevirus)
- Spezies Pseudomonas-Virus D3 (Typus)
- Genus Deurplevirus
- Genus Dhillonvirus (ehem. Hk578virus, Hk578likevirus)
- Spezies Escherichia-Virus HK578 (Typus)
- Genus Dinavirus
- Genus Dismasvirus
- Genus Doucettevirus
- Genus Edenvirus
- Genus Efquatrovirus
- Genus Eiauvirus
- Genus Eisenstarkvirus
- Spezies Xanthomonas-Virus PhiL7 (Typus)
- Genus Elerivirus
- Genus Emalynvirus
- Genus Eyrevirus
- Genus Fairfaxidumvirus
- Genus Farahnazvirus
- Genus Fattrevirus
- Genus Feofaniavirus
- Genus Fernvirus (ehem. Sitaravirus)
- Spezies Paenibacillus-Virus BN12
- Spezies Paenibacillus-Virus Diva
- Spezies Paenibacillus-Virus Eltigre
- Spezies Paenibacillus-Virus Fern,[18] mit Paenibacillus-Phage Willow
- Spezies Paenibacillus-Virus Hb10c2
- Spezies Paenibacillus-Virus Jacopo
- Spezies Paenibacillus-Virus Kawika
- Spezies Paenibacillus-Virus Leyra
- Spezies Paenibacillus-Virus Likha
- Spezies Paenibacillus-Virus Lucielle
- Spezies Paenibacillus-Virus P123
- Spezies Paenibacillus-Virus Pagassa
- Spezies Paenibacillus-Virus PBL1c
- Spezies Paenibacillus-Virus Rani
- Spezies Paenibacillus-Virus Shelly
- Spezies Paenibacillus-Virus Sitara
- Spezies Paenibacillus-Virus Tadhana
- Spezies Paenibacillus-Virus Yyerffej
- Genus Fibralongavirus
- Genus Fowlmouthvirus
- Genus Franklinbayvirus
- Genus Fremauxvirus
- Genus Fromanvirus
- Genus Gaiavirus
- Genus Galaxyvirus
- Genus Galunavirus
- Spezies Gordonia-Virus GAL1 (en. Gordonia virus GAL1, Typus)
- Genus Gamtrevirus
- Spezies Gordonia-Virus GMA3 (en. Gordonia virus GMA3, Typus)
- Genus Gesputvirus
- Spezies Gordonia-Virus Gsput1 (en. Gordonia virus Gsput1, Typus)
- Genus Getseptimavirus
- Spezies Gordonia-Virus GTE7 (en. Gordonia virus GTE7, Typus)
- Genus Ghobesvirus
- Spezies Gordonia-Virus Ghobes (en. Gordonia virus Ghobes, Typus)
- Genus Gilesvirus
- Genus Gillianvirus
- Genus Gilsonvirus
- Genus Glaedevirus
- Genus Godonkavirus
- Genus Goodmanvirus
- Genus Gordonvirus
- Spezies Arthrobacter-Virus Captnmurica (en. Arthrobacter virus Captnmurica)
- Spezies Arthrobacter-Virus Gordon (en. Arthrobacter virus Gordon, Typus)
- Genus Gordtnkvirus
- Spezies Gordonia-Virus GordTnk2 (en. Gordonia virus GordTnk2, Typus)
- Genus Gorganvirus
- Spezies Proteus-Virus Isfahan (en. Proteus virus Isfahan, Typus)
- Genus Gorjumvirus
- Spezies Gordonia-Virus Jumbo (en. Gordonia virus Jumbo, Typus)
- Genus Gustavvirus
- Spezies Gordonia-Virus Gustav (en. Gordonia virus Gustav, Typus)
- Spezies Gordonia-Virus Mahdia (en. Gordonia virus Mahdia)
- Genus Halcyonevirus (früher Trippvirus)
- Genus Hattifnattvirus
- Genus Hedwigvirus
- Genus Helsingorvirus (ehem. Cba181virus, Cba18unalikevirus)
- Spezies Cellulophaga-Virus Cba121 (en. Cellulophaga virus Cba121)
- Cellulophaga-Phage phi12:1
- Cellulophaga-Phage phi12:3
- Spezies Cellulophaga-Virus Cba171 (en. Cellulophaga virus Cba171)
- Cellulophaga-Phage phi17:1
- Spezies Cellulophaga-Virus Cba181 (en. Cellulophaga virus Cba181, Typus)
- Cellulophaga-Phage phi18:1
- Cellulophaga-Phage phi18:2
- Genus Hiyaavirus
- Genus Hnatkovirus
- Spezies Mycobacterium-Virus DS6A (en. Mycobacterium virus DS6A, Typus)
- Genus Holosalinivirus
- Genus Homburgvirus (ehem. P70virus)
- Spezies Listeria-Virus P70
- Genus Hubeivirus
- Genus Iaduovirus
- Genus Ikedavirus
- Genus Ilzatvirus
- Genus Incheonvrus (ehem. P12002virus)
- Spezies Polaribacter-Virus P12002L
- Spezies Polaribacter-Virus P12002S
- Genus Indlulamithivirus
- Genus Inhavirus (ehem. P12024virus)
- Spezies Nonlabens-Virus P12024L
- Spezies Nonlabens-Virus P12024S
- Genus Jacevirus
- Genus Jarrellvirus
- Genus Jenstvirus
- Genus Jouyvirus
- Genus Juiceboxvirus
- Genus Junavirus
- Genus Kairosalinivirus
- Genus Kamchatkavirus
- Genus Karimacvirus
- Genus Kelleziovirus
- Genus Kilunavirus
- Spezies Burkholderia-Virus KL1 (Burkholderia-Phage KL1)[54]
- Genus Klementvirus
- Genus Knuthellervirus
- Genus Kojivirus
- Genus Konstantinevirus
- Genus Korravirus
- Genus Kostyavirus (ehem. Cjw1virus, Cjwunalikevirus)
- Spezies Mycobacterium-Virus CJW1
- Genus Krampusvirus
- Genus Kryptosalinivirus
- Genus Kuleanavirus
- Genus Labanvirus
- Genus Lacnuvirus
- Genus Lacusarxvirus
- Genus Lafunavirus
- Genus Lambovirus
- Genus Lanavirus
- Genus Larmunavirus
- Genus Laroyevirus
- Genus Latrobevirus
- Genus Leicestervirus
- Genus Lentavirus
- Genus Liebevirus
- Genus Liefievirus (ehem. Halolikevirus)
- Spezies Mycobacterium-Virus Halo
- Spezies Mycobacterium-Virus Liefie[27]
- Spezies „Mycobacterium-Phage BPs“ (vorgeschlagen)[20][57][2][58] im „Cluster G“
- BPsΔ33HTH (gantechnische Mutante) mit Varianten BPsΔ33HTH-HRM1 und BPsΔ33HTH-HRM10
- Genus Lillamyvirus
- Genus Lokivirus
- Genus Luckybarnesvirus
- Genus Luckytenvirus
- Genus Lughvirus
- Genus Lwoffvirus (ehem. Tp21virus, Tp2unalikevirus)
- Spezies Bacillus virus TP21
- Genus Magadivirus
- Genus Majavirus
- Genus Manhattanvirus
- Genus Mapvirus (ehem. Ff47virus)
- Genus Mardecavirus (ehem. Ssp2virus)
- Spezies Vibrio-Virus SSP002
- Genus Marienburgvirus
- Genus Marvinvirus
- Genus Maxrubnervirus
- Genus Mementomorivirus
- Genus Metamorphoovirus
- Genus Minunavirus
- Genus Montyvirus
- Genus Mudcatvirus
- Genus Mufasoctovirus
- Genus Muminvirus
- Genus Murrayvirus
- Genus Nanhaivirus
- Genus Nazgulvirus
- Genus Neferthenavirus
- Genus Nesevirus
- Genus Nevevirus
- Genus Nickievirus
- Genus Nonanavirus
- Genus Nyceiraevirus
- Genus Oengusvirus
- Genus Omegavirus (ehem. Omegalikevirus)
- Spezies Mycobacterium-Virus Courthouse[20] im „Cluster E“
- Spezies Mycobacterium-Virus Omega (alias Mycobacterium phage Omega)
- Genus Oneupvirus
- Genus Orchidvirus
- Genus Oshimavirus (ehem. P23virus, P23likevirus)
- Spezies Thermus-Virus P23-45
- Genus Pahexavirus (ehem. Pa6virus)
- Spezies Propionibacterium-Virus PA6
- Genus Pamexvirus (ehem. Pamx74virus)
- Spezies Pseudomonas-Virus PaMx28 (en. Pseudomonas virus PaMx28)
- Spezies Pseudomonas-Virus PaMx74 (en. Pseudomonas virus PaMx74)
- Genus Pankowvirus
- Genus Papyrusvirus (ehem. Send513virus)
- Spezies Mycobacterium-Virus Send513 (en. Mycobacterium virus Send513)
- Genus Patiencevirus
- Genus Pepyhexavirus (ehem. Pepy6virus)
- Spezies Rhodococcus-Virus Pepy6 (en. Rhodococcus virus Pepy6)
- Genus Phicbkvirus (ehem. Phicbklikevirus)
- Genus Phietavirus (ehem. Phietalikevirus)[10]
- Genus Phifelvirus (ehem. Phifllikevirus)
- Genus Picardvirus
- Genus Pikminvirus
- Genus Pleeduovirus
- Genus Pleetrevirus
- Genus Poushouvirus
- Genus Predatorvirus
- Genus Priunavirus
- Genus Psavirus
- Genus Pulverervirus (ehem. Pfr1virus)
- Spezies Propionibacterium-Virus PFR1
- Genus Questintvirus
- Genus Quhwahvirus
- Genus Radostvirus
- Genus Raleighvirus
- Genus Ravarandavirus
- Genus Ravinvirus (ehem. N15virus, N15likevirus, N15-ähnliche Viren), nicht-isometrisches Kapsid
- Spezies Escherichia-Virus N15 (alias Enterobacteria-Phage N15)
- Spezies „Yersinia-Phage PY54“ (alias „Bacteriophage PY54“, alias „Yersinia enterocolitica phage PY54“ – vorgeschlagen)[63]
- Genus Rerduovirus (ehem. Rer2virus)
- Spezies Rhodococcus-Virus RER2
- Spezies Rhodococcus-Virus RGL3 (alias Rhodococcus-Phage RGL3), früher zu Gattung Fromanvirus (damals L5virus bzw. L5likevirus)[64]
- Genus Rigallicvirus
- Genus Rimavirus
- Genus Rockefellervirus
- Genus Rockvillevirus
- Genus Rogerhendrixvirus
- Genus Ronaldovirus
- Genus Roufvirus (ehem. Pis4avirus)
- Spezies Aeromonas-Virus pIS4A
- Genus Rowavirus
- Genus Ruthyvirus
- Genus Samistivirus
- Genus Samunavirus
- Genus Samwavirus
- Genus Sandinevirus
- Genus Sanovirus
- Genus Sansavirus
- Genus Saphexavirus (ehem. Sap6virus, Sap6likevirus)
- Spezies Enterococcus-Virus SAP6
- Genus Sashavirus
- Genus Sasvirus
- Genus Saundersvirus (ehem. Tp84virus)
- Spezies Geobacillus-Virus Tp84
- Genus Sawaravirus
- Genus Scapunavirus
- Genus Schnabeltiervirus
- Genus Schubertvirus
- Genus Seongbukvirus
- Genus Septimatrevirus (ehem. Septima3virus)
- Genus Seussvirus
- Genus Sextaecvirus
- Genus Slashvirus
- Genus Sleepyheadvirus
- Genus Smoothievirus
- Genus Sonalivirus
- Genus Soupsvirus
- Genus Sourvirus
- Genus Sozzivirus
- Genus Sparkyvirus
- Genus Spbetavirus (ehem. Spbetalikevirus, SPbeta-ähnliche Viren), 1 Spezies
- Spezies Bacillus-Virus SPbeta
- Bacillus-Phage SPβ
- Genus Spizizenvirus
- Genus Squashvirus
- Genus Squirtyvirus
- Genus Stanholtvirus (ehem. E125virus, Phie125likevirus)
- Spezies Burkholderia-Virus phi6442 (en. Burkholderia virus phi6442)
- Burkholderia-Phage phi6442
- Spezies Burkholderia-Virus phiE125 (en. Burkholderia virus phiE125)
- BBurkholderia-Phage phie125
- Genus Steinhofvirus (ehem. Jwxvirus)
- Genus Sukhumvitvirus
- Genus Tandoganvirus
- Genus Tankvirus
- Genus Tantvirus
- Genus Terapinvirus
- Genus Teubervirus
- Genus Thetabobvirus
- Genus Tigunavirus
- Genus Timquatrovirus (ehem. Tm4virus, Tm4likevirus)
- Spezies Mycobacterium-Virus Larva[20] im „Subcluster K5“
- Spezies Mycobacterium-Virus TM4[20] im „Subcluster K2“
- Spezies „Mycobacterium-Phage Adephagia“[20] im „Subcluster I2“
- Spezies „Mycobacterium-Phage Amgine“[20] im „Subcluster K6“
- Spezies „Mycobacterium-Phage Keshu“[20] im „Subcluster K3“
- Spezies „Mycobacterium-Phage Krueger“[20] im „Subcluster K6“
- Spezies „Mycobacterium-Phage SamScheppers“[20] im „Subcluster K4“
- Spezies „Mycobacterium-Phage ShedlockHolmes“[20] im „Subcluster K3“
- Spezies „Mycobacterium-Phage Validus“[20] im „Subcluster K1“
- Spezies „Mycobacterium-Phage Wintermute“[20] im „Subcluster K4“
- Spezies „Mycobacterium-Phage ZoeJ“ (vorgeschlagen)[20][57][2][75][76] im „Subcluster K2“
- ZoeJΔ45 (gentechnische Mutante)
- Genus Timquatrovirus
- Genus Tinduovirus (ehem. Tin2virus)
- Spezies Tsukamurella-Virus TIN2
- Genus Titanvirus
- Genus Tortellinivirus
- Genus Triavirus (ehem. 3alikevirus, sic!)[10]
- Spezies Staphylococcus-Virus 3a (en. Staphylococcus virus 3a)[77]
- Staphylococcus-aureus-Phage 3A[11]
- Genus Trigintaduovirus
- Genus Trigintaduovirus
- Genus Trinavirus
- Genus Trinevirus
- Genus Triplejayvirus
- Genus Unahavirus
- Genus Uwajimavirus
- Genus Vashvirus
- Genus Vedamuthuvirus
- Genus Vendettavirus
- Genus Vhulanivirus
- Genus Vidquintavirus
- Genus Vieuvirus
- Genus Vividuovirus
- Genus Vojvodinavirus
- Genus Waukeshavirus
- Genus Wbetavirus (ehem. Wbetalikevirus)
- Genus Weaselvirus
- Genus Whackvirus
- Genus Whiteheadvirus
- Genus Wildcatvirus
- Genus Wilnyevirus
- Genus Wizardvirus
- Genus Woesvirus
- Genus Woodruffvirus (ehem. Ydn12virus)
- Genus Xiamenvirus (ehem. Rdjlvirus),
- Spezies Roseobacter-Virus RDJL1 (en. Roseobacter virus RDJL1, RDJLΦ1)[80]
- Spezies Roseobacter-Virus RDJL2 (en. Roseobacter virus RDJL2, RDJLΦ2)
- Genus Xipdecavirus (ehem. Xp10virus, Xp10likevirus)
- Spezies Xanthomonas-Virus Xp10 (en. Xanthomonas virus Xp10)
- Genus Yangvirus
- Genus Yonseivirus
- Genus Yuavirus (ehem. Yualikevirus)
- Genus Yvonnevirus
- Genus Zetavirus
- vorgeschlagene Genera:[81][10][82][83][84][85]
- Genus „Cba39unalikevirus“
- Spezies: „Cellulophaga-Phage phi39:1“[86]
- Genus „Cba46unalikevirus“
- Spezies: „Cellulophaga-Phage phi46:1“[87]
- Genus „Cba10unalikevirus“
- Genus „R1tlikevirus“
- Spezies: „Lactococcus-Phage r1t“ (alias Bacteriophage r1t)[90]
- Genus „Cyanostylovirus“ (informell: nicht zugeodnete Cyanophagen mit Siphoviren-Morphologie, evtl. eigene Familie „Cyanostyloviridae“ oder „Styloviridae“)
- Spezies „Cyanophage KBS-S-2A“[91]
- Spezies „Cyanophage MED4-117“[92]
- Spezies „Cyanophage PSS2“[93]
- Spezies „Cyanophage S-1“[94][95][96]
- Spezies: „Cyanophage S-2L“ (alias „Cyanobacteria phage S-2L“)[97][98][99][100][101][102] – Wirte aus der Gattung Synechocystis, alle Adenosinbasen im Genom durch 2,6-Diaminopurin ersetzt.[103][104][105][106][107] (Zusammenfassungen auf sciencealert und ScienceNews).[108][109]
- Genus nicht bestimmt (Auswahl von ICTV-unbestätigten Spezies):[113]
- Spezies „Acinetobacter phage SH-Ab 15497“[115][109][105][114][116]
- ?Spezies „Microbacterium-Phage ChickenNugget“ (veraltet „Microbacterium-Phage CrispyBacon“)[27][117]
- ?Spezies „Microbacterium-Phage IAmGroot“[27][118] im „Cluster EH“
- Spezies „Mycobacterium-Phage Cborch11“
- Spezies „Mycobacterium-Phage Damien“
- Spezies „Mycobacterium-Phage Lixy“[27][119]
- Spezies „Mycobacterium-Phage Oaker“
- Spezies „Mycobacterium-Phage Phreeze“
- Spezies „Mycobacterium-Phage Thumb“
- Spezies „Mycobacterium-Phage TGIPhriday“[27][120]
- Spezies „Achromobacter-Phage JWF“[121][73] (Gattung Steinhofvirus?)
- Spezies „Achromobacter-Phage phiAxp-1“[122][54]
- Spezies „Achromobacter-Phage phiAxp-2“[123]
- Spezies „Achromobacter phage vB_AchrS_AchV4“[124]
- Spezies „Arthrobacter-Phage Auxilium“
- Spezies „Arthrobacter-Phage Beagle“
- Spezies „Arthrobacter-Phage Lego“
- Spezies „Arthrobacter-Phage Liebe“
- Spezies „Arthrobacter-Phage Maja“
- Spezies „Arthrobacter-Phage Mimi“ – zu unterscheiden von Acanthamoeba polyphaga Mimivirus (APMV)
- Spezies „Arthrobacter-Phage Sputnik“ – zu unterscheiden Mimivirus-dependent virus Sputnik, einem Virophagen
- Spezies „Bacillus-Phage Basilisk“ (en. „Bacillus cereus phage Basilisk“)[125][126]
- Species „Bacillus-Phage SPP1“ (en. „Bacillus phage SPP1“)[8][127][50][48]
- Spezies „Clostridium phage vB_CpeS-CP51“ (alias „Clostridium phage phiCP51“)[128][129]
- Spezies „Enterobacteria-Phage Phi80“ („Phi80-Phage“, „Phage φ80“)[6]
- Spezies „Enterobacteria phage phi-C3208“ (en. „Enterobacteria phage phi-C3208“, mit Phage ΦC3208 alias φFC3208)[130][12][13][14]
- Spezies „Escherichia-Phage C1“ (en. „Escherichia phage C1“, mit Phage C1)[131][12][14]
- Spezies „Gordonia-Phage DekHockey33“ (en. „Gordonia phage DekHockey33“)[132]
- Spezies „Pseudomonas-Phage phi297“ (en. „Pseudomonas phage phi297“)[133][134]
- Spezies „Streptococcus-Phage Dp-1“
- Spezies „Ruegeria-Phage DSS3-P1“ (en. „Ruegeria phage DSS3-P1“, alias „Roseophage DSS3-P1“)[135]
- Spezies „Roseobacter-Phage DSS3phi1“ (en. „Roseobacter phage DSS3phi1“, DSS3Φ1)[136] – identisch mit DSS3-P1?
- Spezies „Streptococcus-Phage Dp-1“ (en. „Pneumococcus phage Dp-1“, „Diplophage 1“)[137][138]
- Spezies „Streptococcus-pyogenes-Phage T12“ (alias „Bakteriophage T12“, en. „Streptococcus pyogenes phage T12“)[139] — siehe Scharlach
- Species „Tetraselmis viridis virus S20“[140] — womöglich ein Tetraselmis-assoziierter Bakteriophage
- Species „Tetraselmis viridis virus SI1“[141]
- Spezies „Vibrio-Phage 149 (Typ IV)“ (alias „Phage ϕ149“, ehemals Gattung T5virus alias T5likevirus, vom ICTV 2015 aus dieser Gattung genommen, da zu wenig Datenmaterial für die Zuordnung vorliegt)[142][143]
- Spezies „Mycobacterium abscessus subsp. bolletii phage Araucaria“[144]
Die Genom-Organisation in den Gattungen Fromanvirus (alt: L5virus), Lambdavirus (alt: Lambdalikevirus), Lomovskayavirus (alt: Phic31virus), Brussowvirus (altr: Sfi11virus), Moineauvirus (alt: Sfi21dt1virus), Skunavirus (alt: Sk1virus) und Timquatrovirus (alt: Tm4virus), sowie Psimunavirus (alt: „ψM2‐like phage“) legt nahe, dass diese eine Lambda-Supergruppe (oder -Unterfamilie) bilden könnten.[22]
Die Gattung Cetovirus wurde im März 2020 vom ICTV in die neue Familie Demerecviridae, Unterfamilie Ermolyevavirinae verschoben,[145] ebenso die Gattung Jesfedecavirus; die Gattung Tequintavirus (ehem. T5virus, T5likevirus) mit isometrischem Kapsid und den Spezies Escherichia-Virus T5 (mit 'Enterobacteria-Phage T5) sowie Salmonella-Virus Stitch etc. wurde in die Unterfamilie Markadamsvirinae dieser Familie verschoben; die Gattungen Novosibvirus und Sugarlandvirus ebenso, aber ohne Unterfamilienzuordnung. Die Gattung „Chungbukvirus“ mit Typusart „Pseudomonas-Virus Ptobp6g“ (Pseudomonas-Phage phi_Pto-bp6g)[146] weicht laut ICTV erheblich von den Sipohoviridae ab und hat Gemeinsamkeiten mit den Myoviridae; sie wird vom ICTV aktuell nicht mehr gelistet. Paenibacillus-Virus Lily wird aus der Gattung Fernvirus (früher Sitaravirus) verschoben in die neue Gattung Lilyvirus (weiter unter den Caudovirales, aber zunächst ohne Familienzuordnung).
Die Unterfamilie Tunavirinae wurde mit MSL#36 (1. Jahreshälfte 2021) zur Familie Drexlerviridae verschoben, die Gattungen teilweise dort anderen Unterfamilien zugeordnet.
Der „Megasphaera-Phage A9“ (alias „Huge Phage A9“) ist als Riesenphage vermutlich ebenfalls Mitglied der Siphoviridae, infiziert Megasphaera NM10 ([en], Klasse Clostridia);[147] nicht zu verwechseln mit dem Brochothrix-Phagen A9 (Spezies Brochothrix-Virus A9, Herelleviridae).
Literatur
- C. M. Fauquet, M. A. Mayo et al.: Eighth Report of the International Committee on Taxonomy of Viruses. London / San Diego, 2004.
- David M. Knipe, Peter M. Howley et al. (Hrsg.): Fields’ Virology. 4.& Auflage, Philadelphia 2001.
Weblinks
Einzelnachweise
- NCBI: Bacillus phage Gamma (species)
- Daniel Bojar: Spektrum der Wissenschaft, Juni 2020, Spektrum der Wissenschaft, Juni 2020, S. 40–45
- ICTV: ICTV Taxonomy history: Enterobacteria phage T4, EC 51, Berlin, Germany, July 2019; Email ratification March 2020 (MSL #35)
- ICTV: ICTV Master Species List 2020.v1, New MSL including all taxa updates since the 2019 release, March 2021 (MSL #36)
- Light shed on the atomic resolution structure of phage DNA tube, auf: EurekAlert! vom 13. November 2020. Quelle: Forschungsverbund Berlin, Forschungsinstitut für Molekulare Pharmakologie (FMP)
- NCBI: Enterobacteria phage phi80 (species)
- Dann Turner, Andrew M. Kropinski, Evelien M. Adriaenssens: A Roadmap for Genome-Based Phage Taxonomy, in: MDPI Viruses Band 13, Nr. 3, Section Bacterial Viruses, 18. März 2021, 506, doi:10.3390/v13030506
-
Maximilian Zinke, Katrin A. A. Sachowsky, Carl Öster, Sophie Zinn-Justin, Raimond Ravelli, Gunnar F. Schröder, Michael Habeck, Adam Lange: Architecture of the flexible tail tube of bacteriophage SPP1, in: Nature Communications, Band 11, Nr. 5759, 13. November 2020, doi:10.1038/s41467-020-19611-1. Dazu:
- Light shed on the atomic resolution structure of phage DNA tube, auf: EurekAlert vom 13. November 2020. Quelle: Forschungsverbund Berlin (FVB): Leibniz-Forschungsinstitut für Molekulare Pharmakologie (FMP), Berlin (englisch)
- Struktur von Phagen-DNA-Tunnel in atomarer Auflösung aufgeklärt – methodischer Meilenstein gelungen, FV Berlin (FVB), FMP, 13. November 2020 (deutsch)
- NCBI: Staphylococcus virus 80alpha (species)
- D Gutiérrez, EM Adriaenssens, B Martínez, A Rodríguez, R Lavigne, AM Kropinski, P García: Three proposed new bacteriophage genera of staphylococcal phages: "3alikevirus", "77likevirus" and "Phietalikevirus". In: Archives of Virology. 159, Nr. 2, 11. September 2013, S. 389–398. doi:10.1007/s00705-013-1833-1. PMID 24022640.
- Roman Pantůček, J. Doskar, V. Růzicková, P. Kaspárek et al.: Identification of bacteriophage types and their carriage in Staphylococcus aureus, in: Archives of Virology 149(9), S. 1689–1703, Oktober 2004, doi:10.1007/s00705-004-0335-6, PMID 15593413, Abstract und Table 1
- SIB: Viral exotoxin, Expasy: ViralZone
- SIB: Modulation of host virulence by virus, Expasy: ViralZone
- Harald Brüssow, Carlos Canchaya, Wolf-Dietrich Hard: Phages and the Evolution of Bacterial Pathogens: from Genomic Rearrangements to Lysogenic Conversion, in: Microbiol Mol Biol Rev. 68(3), September 2004, S. 560–602, doi:10.1128/MMBR.68.3.560-602.2004, PMC 515249 (freier Volltext), PMID 15353570, siehe insbes. Table 1 und Fig. 6
- NCBI: Staphylococcus phage phiN315 (species)
- NCBI: Staphylococcus phage PVL (species)
- ICTV: ICTV Taxonomy history: Pbi1virus, Proposal (zip)
- Casey Stamereilers, Lucy LeBlanc, Diane Yost, Penny S. Amy, Philippos K. Tsourkas: Comparative genomics of 9 novel Paenibacillus larvae bacteriophages, in: Bacteriophage, Band 6, Nr. 3, e1220349, Epub 5. August 2016, doi:10.1080/21597081.2016.1220349
- Preeti Gipson, Matthew L. Baker, Desislava Raytcheva, Cameron Haase-Pettingell, Jacqueline Piret, Jonathan A. King, Wah Chiu: Protruding knob-like proteins violate local symmetries in an icosahedral marine virus. In: Nature Communications. 5, Nr. 4278, 2. Juli 2014. doi:10.1038/ncomms5278., Corrigendum, in: Nature Communications, Band 6, Nr. 6040, 12. Januar 2015, doi:10.1038/ncomms7040.
- Rebekah M. Dedrick, Carlos A. Guerrero-Bustamante, Rebecca A. Garlena, Daniel A. Russell, Katrina Ford, Kathryn Harris, Kimberly C. Gilmour, James Soothill, Deborah Jacobs-Sera, Robert T. Schooley, Graham F. Hatfull, Helen Spencer: Engineered bacteriophages for treatment of a patient with a disseminated drug-resistant Mycobacterium abscessus, in: Nat Med 25, S. 730–733, 8. Mai 2019, doi:10.1038/s41591-019-0437-z. Freies Autoren-Manuskript vom 8. November 2019, PMC 6557439 (freier Volltext), PMID 31068712. ResearchGate
- NCBI: Lactobacillus phage P2 (species)
- Harald Brüssow, Frank Desiere: Comparative phage genomics and the evolution of Siphoviridae: insights from dairy phages. In: Mol Microbiol. 39, Nr. 2, 2001, S. 213–222. doi:10.1046/j.1365-2958.2001.02228.x, PMID 11136444, 21. Dezember 2001, Stichwort „λ supergroup“.
- PhagesDb: Mycobacterium phage D29
- NCBI: Mycobacterium virus Heldan (species)
- ICTV: ICTV Taxonomy history: Mycobacterium-Virus Heldan (MSL #35)
- NCBI: Mycobacterium phage BabyRay (species)
- Ed Yong: A Huge Discovery in the World of Viruses, auf: The Atlantic vom 20. Februar 2020
- NCBI: Mycobacterium phage Benvolio (species)
- NCBI: Mycobacterium phage Chupacabra (species)
- NCBI: Mycobacterium phage D32 (species)
- PhagesDb: Mycobacterium phage D32
- NCBI: Mycobacterium virus Fred313 (species)
- NCBI: Mycobacterium virus Isca (species)
- NCBI: Mycobacterium virus Puppy (species)
- NCBI: Mycobacterium virus TNguyen7 (species)
- NCBI: Corynephage beta (species)
- NCBI: Corynebacterium diphtheriae virus/phage (species)
- Julie K. Segman (Hrsg.): Stedman's Medical Dictionary, 28th. Auflage, Lippincott Williams & Wilkins, Baltimore, Maryland 2006, ISBN 978-0-7817-3390-8, S. 449.
- TABLE 1. Bacterial virulence properties altered by bacteriophages from P. L. Wagner, M. K. Waldor: Bacteriophage control of bacterial virulence. In: Infection and Immunity. 70, Nr. 8, August 2002, S. 3985–3993. doi:10.1128/IAI.70.8.3985-3993.2002. PMID 12117903. PMC 128183 (freier Volltext).
- L. P. Johnson, M. A. Tomai, P. M. Schlievert: Bacteriophage Involvement in Group A Streptococcal Pyrogenic Exotoxin A Production, in: Journal Of Bacteriology, Band 166, Nr. 2, Mai 1986, S. 623-627
- J. J. Costa, J. L. Michel, R. Rappuoli, J. R. Murphy: Restriction map of corynebacteriophages beta c and beta vir and physical localization of the diphtheria tox operon. In: Journal of Bacteriology. 148, Nr. 1, 1981, S. 124–130. doi:10.1128/JB.148.1.124-130.1981. PMID 6270058. PMC 216174 (freier Volltext).
- NCBI: Corynephage omega (species)
- NCBI: Enterobacteria phage H-19B (species)
- ICTV: ICTV Taxonomy history: Methanobacterium virus psiM1, EC 51, Berlin, Germany, July 2019; Email ratification March 2020 (MSL #35). Siehe 2019: Proposal – psiM2 ersetzt psiM1
- NCBI: Methanobacterium phage psiM2 (species)
- Stephen Hayes, Jennifer Mahony, Renaud Vincentelli, Laurie Ramond, Arjen Nauta Douwe van Sinderen, Christian Cambillau: Ubiquitous Carbohydrate Binding Modules Decorate 936 Lactococcal Siphophage Virions, in: MDPI Viruses, Band 11, Br. 7, Section Bacterial Viruses, 631, 9. Juli 2019, doi:10.3390/v11070631
- NCBI: Lactococcus virus 936 (species)
- https://www.nature.com/articles/s41467-020-19611-1 Silvia Spinelli, David Veesler, Cecilia Bebeacua, Christian Cambillau: Structures and host-adhesion mechanisms of lactococcal siphophages, in: Front. Microbiol., 16. Januar 2014, Part of research topic Gram-positive phages: From isolation to application, doi:10.3389/fmicb.2014.00003
- NCBI: Lactococcus virus P2 (species)
- Matthew Dunne, Mario Hupfeld, Jochen Klumpp, Martin J. Loessner: Molecular Basis of Bacterial Host Interactions by Gram-Positive Targeting Bacteriophages, in: MDPI Viruses, Band 10, Nr. 8, Special Issue Phage-Host Interactions, 397, 28. Juli 2018, doi:10.3390/v10080397. Anm.: B. subtilis phage PBS1 gehört nach ICTV zu den Myoviridae.
- NCBI: Mycobacterium virus Gabriela (species)
- NCBI: Mycobacterium virus Itos (species)
- NCBI: Salmonella virus YSD1 (species)
- Erna Li, Jiangtao Zhao, Yanyan Ma, Xiao Wei et al.: Characterization of a novel Achromobacter xylosoxidans specific siphoviruse: phiAxp-1, in: Scientific Reports, Band 6, 24. Februar 2016, S. 21943, doi:10.1038/srep21943
- Kjærgaard Nielsen, Alexander Byth Carstens, Patrick Browne, René Lametsch, Horst Neve, Witold Kot, Lars Hestbjerg Hansen: The first characterized phage against a member of the ecologically important sphingomonads reveals high dissimilarity against all other known phages, in: Scientific Reports Band 7, Nr. 13566, 19. Oktober 2017, doi:10.1038/s41598-017-13911-1
- Stuart Siddell: Why virus taxonomy is important, Microbiology Society, 13. Februar 2018 (Bilder)
- Graham F. Hatfull: Phage Therapy Treats Patient with Drug-Resistant Bacterial Infection, auf: Howard Hughes Medical Institute vom 8. Mai 2019
- NCBI: Mycobacterium phage BPs (species)
- NCBI: Acinetobacter virus IMEAB3 (species)
- NCBI: Acinetobacter phage IME_AB3 (isolate)
- Jason Godyer: Bacteria-killing viruses can help us win the fight against antibiotic resistance, auf: BBC ScienceFocus vom 16. September 2021
- NCBI: Staphylococcus virus phiETA (species)
- dsDNA Viruses > Siphoviridae, in: ICTV 9th Report (2011)
- ICTV: ICTV Taxonomy history: Rhodococcus virus RGL3, EC 51, Berlin, Germany, July 2019; Email ratification March 2020 (MSL #35)
- NCBI: Pseudomonas virus Ab26 (species)
- NCBI: Pseudomonas phage vB_PaeS_SCH_Ab26 (isolate)
- NCBI: Pseudomonas virus Kakheti25 (species)
- NCBI: Pseudomonas phage vB_Pae-Kakheti25 (isolate)
- NCBI: Gordonia phage Mariokart (species)
- PhageDb: Gordonia phage Mariokart
- Evan Bennett, Colton White, Shallee Page: Analysis of the Complete Genome of Gordonia Phage Mariokart, Franklin Pierce University 2020
- NCBI: Achromobacter phage JWX (species)
- Brigitte Dreiseikelmann, Boyke Bunk, Cathrin Spröer, Manfred Rohde, Manfred Nimtz, Johannes Wittmann: Characterization and Genome Comparisons of Three Achromobacter Phages of the Family Siphoviridae, in: Arch Virol 162(8), 29. März 2017, S. 2191–2201, doi:10.1007/s00705-017-3347-8, PMID 28357512
- NCBI: Achromobacter virus 83-24 (species)
- NCBI: Mycobacterium phage ZoeJ (species)
- PhagesDb: Mycobacterium phage ZoeJ
- NCBI: Staphylococcus virus 3a (species)
- NCBI: Streptomyces phage phiSAJS1 (species)
- Nana Lu, Choljin Kim, Zhi Chen, Ying Wen, Qing Wei, Yi Qiu, Shiwei Wang, Yuan Song: Characterization and genome analysis of the temperate bacteriophage φSAJS1 from Streptomyces avermitilis, in: Virus Res Band 265, Mai 2019, S. 34-42, doi:10.1016/j.virusres.2019.03.006, Epub 6. März 2019, PMID 30851301
- Jacqueline Z.-M. Chan, Andrew D. Millard, Nicholas H. Mann, Hendrik Schäfer: Comparative genomics defines the core genome of the growing N4-like phage genus and identifies N4-like Roseophage specific genes, in: Frontiers in Microbiology, Band 5, Nr. 506, 10. Oktober 2014, doi:10.3389/fmicb.2014.00506. Siehe insbes. Phylogenetischen Baum Fig. 5
- Proux C, van Sinderen D, Suarez J, Garcia P, Ladero V, Fitzgerald GF, Desiere F, Brüssow H: The dilemma of phage taxonomy illustrated by comparative genomics of Sfi21-like Siphoviridae in lactic acid bacteria. In: J. Bacteriol.. 184, Nr. 21, 2002, S. 6026–6036. doi:10.1128/JB.184.21.6026-6036.2002. PMID 12374837. PMC 135392 (freier Volltext).
- Taxonomy Proposals Awaiting Ratification at ICTV
- Taxonomy Proposals Pending at ICTV
- K. Holmfeldt, N. Solonenko, M. Shah, K. Corrier, L. Riemann, N. C. Verberkmoes, M. B. Sullivan: Twelve previously unknown phage genera are ubiquitous in global oceans. In: Proceedings of the National Academy of Sciences. 110, Nr. 31, 2013, S. 12798–803. doi:10.1073/pnas.1305956110. PMID 23858439. PMC 3732932 (freier Volltext).
- Y. D. Niu, T. A. McAllister, J. H. E. Nash, A. M. Kropinski, K. Stanford: Four Escherichia coli O157:H7 Phages: A New Bacteriophage Genus and Taxonomic Classification of T1-Like Phages. In: PLoS ONE. 9, Nr. 6, 2014, S. e100426. doi:10.1371/journal.pone.0100426. PMID 24963920. PMC 4070988 (freier Volltext).
- NCBI: Cellulophaga phage phi39:1 (species)
- NCBI: Cellulophaga phage phi46:1 (species)
- NCBI: Cellulophaga phage phi10:1 (species)
- NCBI: Cellulophaga phage phi19:1 (species)
- NCBI: Lactococcus phage r1t (species)
- NCBI: Cyanophage KBS-S-2A (species)
- NCBI: Cyanophage MED4-117 (species)
- NCBI: Cyanophage PSS2 (species)
- T. A. Sarma: Cyanophages, in: Handbook of Cyanobacteria, CRC Press, 2012, ISBN 1466559411
- Curtis A. Suttle: Cyanophages and Their Role in the Ecology of Cyanobacteria. In: Brian A. Whitton (Hrsg.): The Ecology of Cyanobacteria (en). Springer Netherlands, 1. Januar 2000, ISBN 9780792347354, S. 563–589, doi:10.1007/0-306-46855-7_20.
- Markus G. Weinbauer (Department of Biological Oceanography, Netherlands Institute for Sea Research): Ecology of prokaryotic viruses, in: FEMS Microbiology Reviews, Band 28, Nr. 2, 1. Mai 2004, Epub 27. Oktober 2003, S. 127–181. doi:10.1016/j.femsre.2003.08.001. Offenbar Gleichsetzung von „Cyanophage AS-1“ und „S-1“, in der Folge „Cyanostylovirus“ zu Myoviridae.
- NCBI: Cyanophage S-2L (species)
- US20060270005A1, Genomic library of cyanophage s-2l and functional analysis
- Han Xia et al.: Freshwater cyanophages, in: Virologica Sinica, 28(5), S. 253–259, Oktober 2013, doi:10.1007/s12250-013-3370-1
- M. H. V. van Regenmortel et al.: ICTV 7th Report (PDF; 857 kB) 2000
- R. S. Safferman, R. E. Cannon, P. R. Desjardins, B. V. Gromov, R. Haselkorn, L. A. Sherman, M. Shilo: Classification and Nomenclature of Viruses of Cyanobacteria. In: Intervirology. 19, Nr. 2, 1983, S. 61–66. doi:10.1159/000149339. PMID 6408019.
- Roger Hull, Fred Brown, Chris Payne (Hrsg.): Virology: A Directory and Dictionary of Animal, Bacterial and Plant Viruses, Macmillan Reference Books, ISBN 978-1-349-07947-6, doi:10.1007/978-1-349-07945-2, Treffer
- Ivan Y. Khudyakov, M. D. Kirnos, N. I. Alexandrushkina, B. F. Vanyushin: Cyanophage S-2L contains DNA with 2,6-diaminopurine substituted for adenine. In: Virology. 88, Nr. 1, 1978, S. 8–18. PMID 676082.
- Andrew M. Kropinski et al.: The Sequence of Two Bacteriophages with Hypermodified Bases Reveals Novel Phage-Host Interactions. In: Viruses, Mai 2018, 10(5), S. 217. PMC 5977210 (freier Volltext), PMID 29695085, PMC 5977210 (freier Volltext).
- Yan Zhou, Xuexia Xu, Yifeng Wei, Yu Cheng, Yu Guo, Ivan Khudyakov, Fuli Liu, Ping He, Zhangyue Song, Zhi Li, Yan Gao, Ee Lui Ang, Huimin Zhao, Yan Zhang, Suwen Zhao: A widespread pathway for substitution of adenine by diaminopurine in phage genomes. In: Science. 372, Nr. 6541, 30. April 2021. doi:10.1126/science.abe4882.
- Dona Sleiman, Pierre Simon Garcia, Marion Lagune, Jerome Loc’h, Ahmed Haouz, Najwa Taib, Pascal Röthlisberger, Simonetta Gribaldo, Philippe Marlière, Pierre Alexandre Kaminski: A third purine biosynthetic pathway encoded by aminoadenine-based viral DNA genomes. In: Science. 372, Nr. 6541, 30. April 2021. doi:10.1126/science.abe6494.
- Valerie Pezo, Faten Jaziri, Pierre-Yves Bourguignon, Dominique Louis, Deborah Jacobs-Sera, Jef Rozenski, Sylvie Pochet, Piet Herdewijn, Graham F. Hatfull, Pierre-Alexandre Kaminski, Philippe Marliere: Noncanonical DNA polymerization by aminoadenine-based siphoviruses. In: Science. 372, Nr. 6541, 30. April 2021. doi:10.1126/science.abe6542.
- Jacinta Bowler: Some Viruses Have a Completely Different Genome to The Rest of Life on Earth, auf: sciencealert vom 4. Mai 2021
- Tina Hesman Saey: Some viruses thwart bacterial defenses with a unique genetic alphabet, auf: ScienceNews vom 5. Mai 2021
- NCBI: Lactococcus phage P335 (species)
- NCBI: Lactococcus phage TP901-1 (species)
- NCBI: Lactococcus phage Tuc2009 (species)
- NCBI: unclassified Siphoviridae
- Yunfen Hua, Tingting Luo, Yiqi Yang, Dong Dong, Rui Wang, Yanjun Wang, Mengsha Xu, Xiaokui Guo, Fupin Hu, Ping He: Phage Therapy as a Promising New Treatment for Lung Infection Caused by Carbapenem-Resistant Acinetobacter baumannii in Mice, in: Front. Microbiol., 9. Januar 2018; doi:10.3389/fmicb.2017.02659
- NCBI: Acinetobacter phage SH-Ab 15497 (species)
- Natalia Bagińska, Anna Pichlak, Andrzej Górski1, Ewa Jończyk-Matysiak: Specific and Selective Bacteriophages in the Fight against Multidrug-resistant Acinetobacter baumannii, in: Virologica Sinica, Band 34, S. 347–357; doi:10.1007/s12250-019-00125-0, Tbl. 1
- PhageDb: Microbacterium phage ChickenNugget
- PhageDb: Microbacterium phage IAmGroot
- PhageDb: Mycobacterium phage Lixy (Siphoviridae)
- PhageDb: Mycobacterium phage TGIPhriday (Siphoviridae)
- NCBI: Achromobacter phage JWF (species)
- NCBI: Achromobacter phage phiAxp-1 (species)
- NCBI: Achromobacter phage phiAxp-2 (species)
- Laura Kaliniene, Algirdas Noreika, Algirdas Kaupinis, Mindaugas Valius, Edvinas Jurgelaitis, Justas Lazutka, Rita Meškienė, Rolandas Meškys: Analysis of a Novel Bacteriophage vB_AchrS_AchV4 Highlights the Diversity of Achromobacter Viruses, in: Viruses 13(3), 374, 27. Februar 2021, doi:10.3390/v13030374
- NCBI: Bacillus phage Basilisk (species)
- Viruses Like Herpes and Zika Will Need6 to Be Reclassified, Here’s Why, auf: SciTechDaily vom 27. September 2019, Quelle: San Diego State University, Fig. 2a
- NCBI: Bacillus phage SPP1 (species)
- NCBI: Clostridium phage vB_CpeS-CP51 (species)
- Eunsu Ha, Bokyung Son, Sangryeol Ryu: Clostridium perfringens Virulent Bacteriophage CPS2 and Its Thermostable Endolysin LysCPS2, in: MDPI Viruses, Band 10, Nr. 5, Special Issue Biotechnological Applications of Phage and Phage-Derived Proteins, 251, 11. Mai 2018, doi:10.3390/v10050251
- NCBI: Enterobacteria phage phi-C3208 (species)
- NCBI: Escherichia phage C1 (species)
- PhagesDb: Gordonia phage DekHockey33, auf: The Actinobacteriophage Database. Cluster A, Subcluster A15, Temperate
- NCBI: Pseudomonas phage phi297 (species)
- Victor Krylov, Olga Shaburova, Sergey Krylov, Elena Pleteneva: A Genetic Approach to the Development of New Therapeutic Phages to Fight Pseudomonas Aeruginosa in Wound Infections, in: MDPI Viruses Band 5, Nr. 1, 21. Dezember 2012, Special Issue Recent Progress in Bacteriophage Research, S. 15-53, doi:10.3390/v5010015
- NCBI: phage DSS3-P1 (species)
- NCBI: Roseobacter phage DSS3phi1 (species)
- NCBI: Streptococcus phage Dp-1 (species)
- Maureen McDonnell, Concepcion Ronda-Lain, Alexander Tomasz: “Diplophage”: a bacteriophage of Diplococcus pneumoniae, Virology Band 63, Nr. 2, Februar 1975, S. 577–582, doi:10.1016/0042-6822(75)90329-3
- NCBI: Bacteriophage T12 (species)
- NCBI: Tetraselmis viridis virus S20 (species)
- NCBI: Tetraselmis viridis virus SI1 (species)
- ICTV: ICTV Taxonomy history: Vibrio phage 149 (type IV), EC 47, London, UK, July 2015; Email ratification 2016 (MSL #30)
- Andrew M. Kropinski et al.: To amend the description of the T5likevirus; and, add four (4) new species, auf: Talk ICTVonline
- Mohamed Sassi, Cecilia Bebeacua, Michel Drancourt, Christian Cambillau: The first structure of a mycobacteriophage, the Mycobacterium abscessus subsp. bolletii phage Araucaria, in: ASM J Virol, Band 87, Nr. 14, S. 8099-8109, 27. Juni 2013/Juli 2013, doi:10.1128/JVI.01209-13, PMC 3700213 (freier Volltext), PMID 23678183, Epub 15. Mai 2013
- ICTV: ICTV Taxonomy history: Cetovirus, EC 51, Berlin, Germany, July 2019; Email ratification March 2020 (MSL #35)
- NCBI: Pseudomonas phage phiPto-bp6g
- Basem Al-Shayeb, Rohan Sachdeva, Lin-Xing Chen, Cindy J. Castelle, Alexander L. Jaffe, Jennifer A. Doudna, Jillian F. Banfield et al.: Clades of huge phage from across Earth’s ecosystems, in: Nature 578, 12. Februar 2020, S. 425–431, doi:10.1038/s41586-020-2007-4. Dazu:
- bioRxiv, PrePrint vom 11. März 2019, doi:10.1101/572362
- Huge bacteria-eating viruses close gap between life and non-life – Large bacteriophages carry bacterial genes, including CRISPR and ribosomal proteins; auf: EurekAlert! vom 12. Februar 2020
