Phosphoenolpyruvatcarboxylase
Das Enzym Phosphoenolpyruvatcarboxylase (PEPCase, PEPC) carboxyliert irreversibel Phosphoenolpyruvat zu Oxalacetat in Pflanzen, Bakterien und Archaeen. Es ist eine wesentliche Schaltstelle im pflanzlichen Stoffwechsel, weshalb sie bei Pflanzen als Biomarker eingesetzt werden kann. Es können verschiedene Schadbilder (Pflanzenkrankheiten mit ursächlichem Bezug zu Mangelversorgung) mit beachtlicher Genauigkeit gemessen und die Einschätzung von Waldschäden kann gegenüber der ausschließlichen Beobachtung der Zeigerpflanzen verbessert werden.
| Phosphoenolpyruvat-Carboxylase (Zea mays) | ||
|---|---|---|
|
Vorhandene Strukturdaten: 1jqo | ||
| Masse/Länge Primärstruktur | 970 Aminosäuren | |
| Sekundär- bis Quartärstruktur | Homotetramer | |
| Kofaktor | Mg2+ | |
| Bezeichner | ||
| Gen-Name(n) | pep (MaizeGDB) | |
| Enzymklassifikation | ||
| EC, Kategorie | 4.1.1.31, Carboxy-Lyase | |
| Substrat | H2O + Phosphoenolpyruvat + CO2 | |
| Produkte | Phosphat + Oxaloacetat | |
| Vorkommen | ||
| Übergeordnetes Taxon | Pflanzen, div. Mikroorganismen[1] | |
Entdeckung
1953 wurde PEPC aus Spinatblättern zum ersten Mal isoliert und in allen bereits untersuchten Pflanzen, Algen und Bakterien entdeckt, jedoch nicht in Tieren oder Pilzen. Auch in Genom von Archaeen wurde PEPC identifiziert, dieses unterscheidet sich jedoch erheblich von der PEPC aus Bakterien und Pflanzen.
Einführung
PEPC ist ein wichtiges Enzym von Bakterien, Archaeen und höheren Pflanzen, insbesondere zur reduktiven Kohlenstoffdioxid-Fixierung bei Pflanzen mit C4- oder Crassulaceen-Säurestoffwechsel. Darüber hinaus spielt es auch eine Rolle im Metabolismus bei der Synthese von Kohlenstoffverbindungen während der Stickstofffixierung bei Leguminosen, Regelung des Turgors, Aufrechterhaltung des Ionenhaushaltes oder Regulation des pH-Wertes.
Zunächst ist man davon ausgegangen, dass die PEPC in Eukaryoten gemäß Endosymbiontentheorie ursprünglich aus Protocyanobakterien stamme. Jedoch ähneln PEPC aus Pflanzen eher denen aus γ-Proteobakterien. Entweder hat der für das Mitochondrium verantwortliche Vorgänger des α-Proteobakteriums bzw. der für den Chloroplasten verantwortliche Vorgänger des Cyanobakteriums die PEPC-Gene (ppc) via horizontalen Gentransfer (HGT) von jenem γ-Proteobakterium erhalten. Alternativ sind die PEPC-Gene aus γ-Proteobakterien relativ früh in der Pflanzengeschichte via HGT eingebracht worden.
Eigenschaften
Das Enzym ist strukturell ein Homotetramer mit vier aktiven Zentren. Es nutzt zweiwertiges Magnesium (Mg2+) als Cofaktor. Aus Mais und Escherichia coli liegen dreidimensionale Strukturen vor. Das Monomer besteht aus einem β-Fass mit acht β-Faltblättern und aus 40 α-Helices. Das aktive Zentrum ist am C-terminalen Bereich des β-Fasses verortet. Die aktive Seite ähnelt auch der aus Pyruvatkinase oder Pyruvat-Phosphat-Dikinase, auch wenn sie sich in ihrer Aminosäurensequenz unterscheiden.
Bei den PEPC verschiedener Organismen liegen Unterschiede vor: In höheren Pflanzen ist jede Untereinheit von PEPC bis 1.000 Aminosäuren groß (bis zu 110 kDa). Die größte berichtete PEPC-Untereinheit stammt aus der Alge Chlamydomonas reinhardtii (1.221 Aminosäuren mit 131 kDa). Die Untereinheiten in Methanothermobacter thermautotrophicus, einem Archeon, ist 55 kDa schwer, was etwa der Hälfte der sonst anzutreffenden Varianten entspricht. Allgemein sind archaelle PEPC viel kleiner als bakterielle oder pflanzliche PEPC.
In Pflanzen sind die ppc-Gene stark konserviert, und weisen üblicherweise 10 bis 11 Exone und 9 bis 10 Introne auf. Auch die Anordnung dieser Exone und Introne ist höchst konserviert. Die Anzahl der ppc-Gene unterscheidet sich in verschiedenen Pflanzenarten, beispielsweise sechs in Reis oder vier in Acker-Schmalwand. Zudem finden sich auch verschiedene Klassen an PEPC. So liegt in vollständig sequenzierten Genomen von Acker-Schmalwand und Reis ein ppc-Gen vor, das eher bakterieller PEPC ähnelt.
Die große Bedeutung der PEPC zeigt sich auch in ihrer Verbreitung. PEPC kommt in Methanopyrus kandleri vor, ein Archaeon das gewöhnlich in den heißen Wasserpfützen von Vulkanen lebt, was angesichts der Temperaturabhängigkeit der PEPC ungewöhnlich ist.
Reaktion
PEPC ist eine Lyase, welche funktional spezifiziert eine C–C-Lyase bzw. eine Carboxy-Lyase ist. Sie setzt PEP und Bicarbonat (HCO3−) irreversibel in folgender Reaktion um:
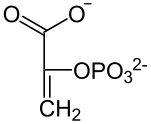 | HCO3− Pi | 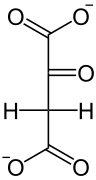 |
| Phosphoenolpyruvat (PEP) | Oxalacetat |
Die Michaeliskonstante für die Umsetzung von PEP (Phosphoenolpyruvat) zu Oxalacetat liegt bei 5 – 6 × 10−4 mol/l für PEP und bei 3,1 × 10−3 mol/l für HCO3−
Mechanismus
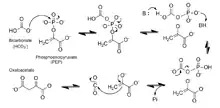
Die Reaktanten werden in das reaktive Zentrum des Enzyms transportiert. Dort wird das PEP relativ zentral positioniert, das Mg2+ wird als Cofaktor in die Nähe des Reaktionszentrums gebracht und dann das Hydrogencarbonat in die Nähe des Reaktionsortes bewegt. Für den genauen Reaktionsmechanismus ist aber nicht ganz geklärt, ob PEP und Magnesium getrennt oder als Komplex ans Enzym binden. Es wird diskutiert, ob eventuell sogar die energetische Steuerung der Reaktion durch das Enzym beeinflusst wird. Nach Ablauf der Reaktion werden die Produkte wieder ausgeschleust, was vermutlich durch das Enzym begünstigt oder initiiert wird.
Regulation
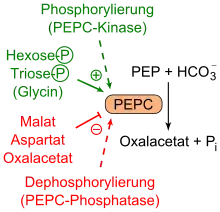
PEPC wird durch viele Faktoren reguliert. So sind Glucose-6-phosphat und andere photosynthetisch erzeugte Zuckerphosphate (Triosephosphate) Aktivatoren, während Aspartat, Oxalacetat und Malat Feedback-Hemmer sind. In nicht-photosynthetischen Isoenzymen wirken jene Inhibitoren wesentlich stärker. In Einkeimblättrigen wird es zudem durch Glycin inhibiert. Glucose-6-phosphat erhöht die Affinität des Enzyms zu PEP und moduliert die seiner allosterischen Inhibitoren.
Der Grad dieser metabolitischen Aktivatoren und Inhibitoren wird durch Phosphorylierung an einem N-terminalen Serinrest von PEPC beeinflusst. Eine Phosphorylierung bewirkt ein Einschalten des Enzyms, da die Sensitivität gegenüber den allosterischen Inhibitoren Malat und Aspartat gesenkt wird. Die Stelle, an der phosphoryliert wird, ist in Eukaryoten höchst konserviert, wurde aber in Bakterien nicht gefunden. Katalysiert wird diese Phosphorylierung durch eine Ca2+-unabhängige, nur 30 kDa leichte Serin-/Threoninkinase, der PEPC-Kinase (PEPC-K).
Die in Archaeen gefundene PEPC unterscheidet sich darin, dass sie sich nicht analog wie bei Pflanzen oder Bakterien regulieren lässt, außerdem fehlen die dafür nötigen allosterischen Bindungsstellen.
Nutzen
Durch die genetischen Differenzen gibt es geringfügige Abweichungen innerhalb botanisch identischer Vertreter derselben Art, was sich neben strukturellen Unterschieden auch in der messbaren enzymatischen Aktivität und bestimmten Toleranzen von Bäumen und anderen Pflanzen gegenüber Umweltgiften niederschlägt. Diese Unterschiede sind, bei ausreichend genauen Messmethoden, sehr zuverlässige Maße für den Gesundheitszustand der Pflanze.
ppc ist ein molekularer Marker, der in Wochen- bis Monatsfrist auf Schadeinwirkungen aus der Umwelt reagiert. Signifikant reagiert das Enzym auf ein (erhöhtes, und mit leicht verringerter Genauigkeit auch erniedrigtes) Stickstoffangebot sowie auf die Phosphatregulation; des Weiteren kann man zeigen, dass PEPC auf Ozon, das Magnesiumangebot sowie Schwermetalle und Pestizide anspricht. Die Ansprache ist durchaus quantitativ messbar, indem man entsprechende Klone der zu untersuchenden Pflanze als Referenz nutzt. Selbst mit künstlichen Essays (Modellsubstanzen) kann man eine für praktische Schadensbegutachtung ausreichende Genauigkeit erreichen.
Literatur
- Udo Gowik und Peter Westhoff: C4-Phosphoenolpyruvate Carboxylase. In: Agepati S. Raghavendra, Rowan F. Sage (Hrsg.): C4 photosynthesis and related CO2 concentrating mechanisms. Springer, Dordrecht 2011, ISBN 978-90-481-9406-3 (Reihe Advances in photosynthesis and respiration Band 32).
- O'Leary B., Park, J. und Plaxton, WC. (2011): The remarkable diversity of plant PEPC (phosphoenolpyruvate carboxylase): recent insights into the physiological functions and post-translational controls of non-photosynthetic PEPCs. In: Biochem J. 436(1); 15–34; PMID 21524275; doi:10.1042/BJ20110078
Einzelnachweise
- PROSITE documentation PDOC00330. Phosphoenolpyruvate carboxylase. Swiss Institute of Bioinformatics (SIB), abgerufen am 12. August 2011 (englisch).