Escherichia-Virus HK97
Escherichia-Virus HK97 (Akronym HK97) ist eine Spezies bakterieller Viren. Ihre Wirte sind entsprechend der Bezeichnung Bakterien der Gattung Escherichia (Escherichien), d. h. Colibakterien (Bakterienspezies E. coli) und Verwandte. Die Viren dieser Spezies werden daher auch als Bakteriophagen (oder kurz Phagen) klassifiziert, daher auch der alte Name Bakteriophage HK97. Das Akronym HK steht für Hongkong, dem Ort, an dem diese Viren erstmals isoliert wurden. Wie die Lambda-Phagen und andere lamboide Phagen (Gattung Lambdavirus) besitzt HK97 ein doppelsträngiges DNA-Genom. Aufgrund größerer genetischer Unterschiede ordnet man die Spezies heute aber nicht mehr in diese, sondern in eine Gattung Hendrixvirus derselben Virusfamilie Siphoviridae ein, deren Typusspezies HK97 ist.[2] Zusammen mit anderen Phagen mit Kopf-Schwanz-Struktur gehört die Siphoviridae zur Ordnung Caudovirales. Dieses bakterielle Virus ist zu unterscheiden vom Riesenvirus Hokovirus (HKV oder HokV) und der humanpathogenen Gattung Tetraparvovirus, früher auch Hongkong-Virus oder Hokovirus genannt.
| Escherichia-Virus HK97 | ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
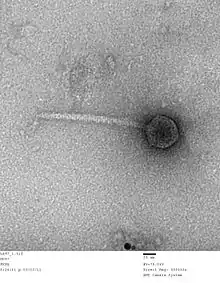
TME-Aufnahme eines Virusteilchens von Escherichia-Virus HK97 | ||||||||||||||||
| Systematik | ||||||||||||||||
| ||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||
| ||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||
| Escherichia virus HK97 | ||||||||||||||||
| Kurzbezeichnung | ||||||||||||||||
| HK97 | ||||||||||||||||
| Links | ||||||||||||||||
|
Vermehrungszyklus
Die Virusteilchen (Virionen) von HK97 haben eine Kopf-Schwanz-Struktur: Der Kopf (das eigentliche Kapsid) beherbergt das genetische Material, hier Doppelstrang-DNA (dsDNA). Durch den Schwanz wird nach Andocken an die bakterielle Wirtszelle dieses in den Wirt injiziert. Im Einzelnen sind die Vorgänge wie folgt:
Das Kapsidprotein gp5 von HK97 vernetzt sich bei der Reifung zu einer verketteten kettenhemdartigen Struktur.[3] Der Bakteriophage durchläuft bei der DNA-Verpackung in das Kapsid einen Reifungsprozess, bei dem er sich um fast 5 nm ausdehnt und seine Geometrie von sphärisch symmetrisch zu ikosaedrisch symmetrisch wechselt.
Der Zusammenbau (Assemblierung) der Virusteilchen (Virionen) beginnt mit der Selbstorganisation des Kapsidproteins gp5 zu Pentameren und Hexameren. Die Protease gp4 spaltet gp5 an ihrem Amino-Terminus. Die anschließende Anlagerung eines so genannten Portalproteins (englisch portal protein, hier mit Bezeichnung gp3) bewirkt Konformationsänderungen, die zur Bildung einer Prohead- oder Procapsid-Struktur – dem noch nicht mit genetischem Material gefüllte Kapsidkopf – führen. Weitere Konformationsänderungen und die Vernetzung von gp5-Monomeren bewirken eine weitere Kapsidreifung und führen zur Bildung eines fertigen Phagenkopfes.[4] Im Gegensatz zu den meisten anderen Viren mit Kopf-Schwanz-Struktur (Ordnung Caudovirales) ist für die Kapsid-Assemblierung kein Gerüstprotein (englisch scaffolding protein) erforderlich (ein solches würde das Procapsid wie ein Gerüst stützen und wird nach dem Verpacken meist abgebaut).[5][6]
SIE
Superinfection exclusion (SIE)[7] ist ein Phänomen, bei dem eine bereits bestehende Virusinfektion (Erstinfektion) in einer Konkurrenzsituation eine Sekundärinfektion mit demselben oder einem eng verwandten Virus verhindert. Dieser Effekt ist also ein Phänotyp des betreffenden Virus, allerdings in vielen Fällen noch schlecht verstanden. SIE kann eine Art antiviralen Zustand der infizierten Zelle widerspiegeln.[8][9]
Es gibt eine Reihe von Phagen (d. h. bakteriellen Viren), die Proteine kodieren, um SIE zu erreichen. Diese viralen Proteine können die DNA-Freisetzung in das Wirtszytoplasma stören oder den Eintrittsrezeptor modifizieren. Sie können auch als Inhibitoren von viralem Peptidoglycan-Abbauenzymen – mit denen Phagen die Wand der Bakterienzelle durchlöchern – wirken, und so den Eintritt von weiteren, neu ankommenden Phagen verhindern. Zu diese Gruppe von Phagen gehört auch HK97 mit seinem SIE-Protein gp15.[8]
Systematik
Die Gruppe der HK97-ähnlichen Phagen wurde früher aufgrund morphologischer Ähnlichkeiten der Gattung der lambdoiden Phagen (Lambdalikevirus, heute Lambdavirus) zugeschlagen. Aufgrund genetischer Analysen hat das ICTV inzwischen für diese Gruppe in den Rang einer eigenständigen Gattung erhoben. Der heutige Gattungsname Hendrixvirus ehrt den Virologen Roger W. Hendrix, der sich besonders um die Erforschung dieser Phagen verdient gemacht hat.[2][10]
- Gattung Hendrixvirus (veraltet Hk97virus, HK97-ähnliche Viren)
- Spezies Escherichia virus HK022 (Enterobacteria Phage HK022)
- Spezies Escherichia virus HK75 mit Escherichia phage HK75
- Spezies Escherichia virus HK97 (Enterobacteria Phage HK97)
- Spezies Escherichia virus HK106 mit Enterobacteria phage HK106
- Spezies Escherichia virus HK446 mit Escherichia phage HK446
- Spezies Escherichia virus HK542 mit Escherichia virus HK542
- Spezies Escherichia virus HK544 mit Escherichia phage HK544
- Spezies Escherichia virus HK633 mit Escherichia phage HK633
- Spezies Escherichia virus mEp234 mit Escherichia phage mEp234
- Spezies Escherichia virus mEpX1 mit Escherichia phage mEpX1
- Spezies Escherichia virus mEpX2 mit Escherichia phage mEpX2
- nicht-klassifizierte Kandidaten zur Gattung Hendrixvirus:[11]
- Spezies „Enterobacteria phage HK140“
- Spezies „Enterobacteria phage mEp235“
- Spezies „Escherichia phage ECP1“
Einzelnachweise
- ICTV: ICTV Taxonomy history: Enterobacteria phage T4, EC 51, Berlin, Germany, July 2019; Email ratification March 2020 (MSL #35)
- ICTV: ICTV Master Species List 2018b.v2 MSL #34v
- Charlotte Helgstrand, William R. Wikoff, Robert L. Duda, Roger W. Hendrix, John E. Johnson, Lars Liljas: The Refined Structure of a Protein Catenane: The HK97 Bacteriophage Capsid at 3.44Å Resolution. In: Journal of Molecular Biology. 334, Nr. 5, 1. Dezember 2003, S. 885–899. doi:10.1016/j.jmb.2003.09.035. PMID 14643655.
- Roger W. Hendrix, John E. Johnson: Bacteriophage HK97 capsid assembly and maturation.. In: Advances in Experimental Medicine and Biology. 726, 2012, S. 351–363. doi:10.1007/978-1-4614-0980-9_15. PMID 22297521.
- Robert L. Duda, K. Martincic, Roger W. Hendrix: Genetic basis of bacteriophage HK97 prohead assembly. In: J. Mol. Biol.. 247, Nr. 4, 1995, S. 636–647. doi:10.1006/jmbi.1994.0169. PMID 7723020.
- B. Oh, C. L. Moyer, Roger W. Hendrix, Robert L. Duda: The delta domain of the HK97 major capsid protein is essential for assembly. In: Virology. 456–457, 2014, S. 171–178. doi:10.1016/j.virol.2014.03.022. PMID 24889236. PMC 4044616 (freier Volltext).
- Knut J. Heller, Horst Neve: Superinfection exclusion und DNA-Injektion bei Siphoviridae-Phagen, Biospektrum Heft 1 (2014), S. 26–29
- SIB: Superinfection exclusion, auf: ExPASy ViralZone
- Vergleiche auch Influenza-A- und Rhinoviren: Nadja Podbregar: Grippe schützt vor Erkältung – Forscher weisen erstmals Wechselwirkung zweier viraler Krankheitserreger nach, auf: scinexx.de vom 17. Dezember 2019
- NCBI: Hendrixvirus (genus)
- NCBI: unclassified Hendrixvirus
Weblinks
- Rober L. Duda: Dr. Bob Duda's Publications
- Roger W. hendrix: Dr. Roger Hendrix's Publications
- UniProt: UniProt Taxonomy: Enterobacteria phage HK97