EcoRV
EcoRV ist ein Restriktionsenzym aus dem Bakterium Escherichia coli.
| Endonuklease EcoRV | ||
|---|---|---|
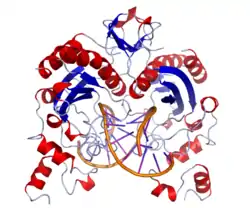 | ||
| nach PDB 1AZ0 | ||
| Masse/Länge Primärstruktur | 29.500 Da | |
| Sekundär- bis Quartärstruktur | Homodimer | |
| Bezeichner | ||
| Gen-Name(n) | EcoRV (Rebase) | |
| Enzymklassifikation | ||
| EC, Kategorie | 3.1.21.4, Restriktionsenzym | |
| Reaktionsart | Hydrolyse | |
| Substrat | DNA | |
| Produkte | Zweisträngige DNA-Fragmente mit terminalen 5'-Phosphatgruppen | |
| Vorkommen | ||
| Übergeordnetes Taxon | Escherichia coli | |
Eigenschaften
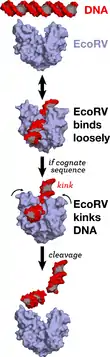
EcoRV ist eine Endonuklease (Typ II, Subtyp P), die DNA an einer palindromischen DNA-Erkennungsequenz schneidet. In suboptimalen Pufferbedingungen sinkt die Affinität von EcoRV zu der Erkennungssequenz und die DNA wird unspezifisch geschnitten (Star-Aktivität). Durch den Schnitt der DNA durch EcoRV entsteht ein blunt end (Ende ohne Überhang) und je einer Phosphatgruppe an beiden 5'-Enden der doppelsträngigen DNA-Produkte. EcoRV wird weder durch eine DAM-Methylierung, noch durch eine DCM- oder eine CpG-Methylierung gehemmt. Nach einer Restriktion von DNA in vitro kann EcoRV durch 20-minütiges Erhitzen auf 80 °C denaturiert und somit inaktiviert werden. Eco32I ist ein Isoschizomer von EcoRV und kann alternativ verwendet werden. Meistens wird EcoRV als rekombinantes Protein in E.coli hergestellt.
| Erkennungssequenz | Restriktionsschnitt |
|---|---|
5'-GATATC-3' 3'-CTATAG-5' |
5'-GAT ATC-3' 3'-CTA TAG-5' |
Struktur
EcoRV besitzt am N-Terminus eine Dimerisierungs-Proteindomäne, bestehend aus einer kurzen α-Helix, einem zweisträngigen antiparallelen β-Faltblatt und einer langen α-Helix.[1] Sie bindet – wie alle typischen Endonukleasen des Typs II – als Homodimer an die DNA.[2] Die DNA wird beim Schneiden an der Schnittstelle zwischen den Basen Thymin und Adenin in einem Winkel von 50° gebogen, was einzigartig unter den Endonukleasen des Typs II ist.[1] Nach einem Gleiten der EcoRV entlang der DNA werden die äußeren Nukleinbasen erkannt und anschließend das Vorhandensein der zentralen und vergleichsweise kleinen Basen TA geprüft, die für eine Biegung der DNA notwendig sind.[3]
Anwendungen
EcoRV wird für Restriktionsverdaue im Rahmen von Klonierungen[4] oder Restriktionsanalysen verwendet.[5]
Einzelnachweise
- Alfred Pingoud, A. Jeltsch: Structure and function of type II restriction endonucleases. In: Nucleic acids research. Band 29, Nummer 18, September 2001, S. 3705–3727, PMID 11557805, PMC 55916 (freier Volltext).
- J. Bitinaite, D. A. Wah, A. K. Aggarwal, I. Schildkraut: FokI dimerization is required for DNA cleavage. In: Proceedings of the National Academy of Sciences. Band 95, Nummer 18, September 1998, S. 10570–10575, PMID 9724744, PMC 27935 (freier Volltext).
- M. Zahran, I. Daidone, J. C. Smith, P. Imhof: Mechanism of DNA recognition by the restriction enzyme EcoRV. In: Journal of molecular biology. Band 401, Nummer 3, August 2010, S. 415–432, doi:10.1016/j.jmb.2010.06.026, PMID 20600128.
- Keith Wilson: Principles and Techniques of Biochemistry and Molecular Biology. Cambridge University Press, 2010, ISBN 978-0-521-51635-8, S. 218.
- Henri Grosjean: DNA and RNA Modification Enzymes. Taylor & Francis, 2009, ISBN 978-1-587-06329-9, S. 82–83.