Clostridium ljungdahlii
Clostridium ljungdahlii ist eine Bakterienart aus der Gattung der Clostridien. Sie lebt in sauerstofffreien Substraten und wurde 1988 erstmals aus dem Abfall eines Hühnerzuchtbetriebs isoliert. Unter ungünstigen Lebensbedingungen bildet sie nach wenigen Stunden Endosporen, die mehrere Jahre als Überdauerungsstadien überleben und unter anaeroben Bedingungen wieder zu vegetativen Bakterien werden.
| Clostridium ljungdahlii | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Systematik | ||||||||||||
| ||||||||||||
| Wissenschaftlicher Name | ||||||||||||
| Clostridium ljungdahlii | ||||||||||||
| Tanner et al. 1993 |
Sämtliche Informationen über das Bakterium entstammen der Untersuchung von Laborkulturen, Beobachtungen aus den Ursprungssubstraten liegen nicht vor. Aufgrund seiner Fähigkeit, fermentativ Ethanol und Essigsäure auf der Basis von Synthesegas herzustellen (Synthesegas-Fermentation), hat es eine potenzielle Bedeutung für die industrielle Biotechnologie, Produktionsanlagen existieren allerdings bislang nur im Pilotmaßstab. Im Jahr 2010 wurde zum besseren Verständnis des Stoffwechsels und zur Optimierung des Bakteriums als Produktionsorganismus das Genom von Clostridium ljungdahlii sequenziert und publiziert.[1]
Merkmale
Innerhalb der Bakterien und insbesondere auch innerhalb der Clostridien werden die Arten vor allem aufgrund ihrer Stoffwechseleigenschaften und molekularbiologischer Merkmale unterschieden, während morphologische Merkmale nur begrenzt für die Bestimmung einzelner Arten nutzbar sind.
Morphologie
Clostridium ljungdahlii ist ein stäbchenförmiges, motiles, grampositives Bakterium. Die Breite der Bakterienzelle beträgt 0,6 μm bei einer Länge von 2 bis 3 μm. Die Zelloberfläche ist von einer 0,1 bis 0,2 μm dicken Zellumhüllung umgeben. Wie andere Clostridien kann sich auch dieses mit den auf der gesamten Bakterienoberfläche gleichmäßig angeordneten Geißeln (peritrich) aktiv bewegen und bildet keine Zellkolonien. An den beiden Zellpolen (terminal und subterminal) kann das Bakterium Endosporen bilden.[2][3]
Stoffwechsel
Clostridium ljungdahlii lebt unter anoxischen (anaeroben) Bedingungen, also in einer Umgebung ohne Sauerstoff. Das pH-Optimum für das Wachstum des Bakteriums liegt im leicht sauren Bereich von 6,0 mit einem Toleranzbereich zwischen 4,0 und 7,0, in der Wachstum erfolgt. Die Optimaltemperatur beträgt etwa 37 °C mit einem Toleranzbereich von 30 bis 40 °C. Unter diesen Optimalbedingungen hat der Stamm ATCC 49587T (T steht für Typus) eine Teilungsrate von 0,26 Teilungen pro Stunde auf einem Fructosemedium oder einem Medium mit Wasserstoff und Kohlenstoffdioxid, die Anzahl der Bakterien verdoppelt sich also etwa alle vier Stunden.[2]
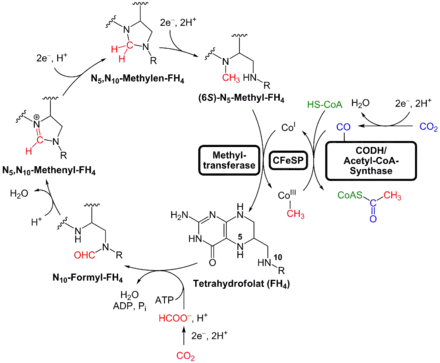
Clostridium ljungdahlii ist ein homoacetogenes Bakterium und in der Lage, auf unterschiedlichen kohlenstoffhaltigen Substraten zu wachsen. Diese umfassen Ethanol, Pyruvat, Arabinose, Xylose, Fructose und Glucose sowie Kohlenstoffmonoxid oder Kohlenstoffdioxid gemeinsam mit Wasserstoff (gasförmig in Flüssigmedium). Methanol, Ferulasäure, Milchsäure, Galactose, Mannose, Saccharose und Stärke führen dagegen nicht zu einer Wachstumsförderung.[2]
Die Aufnahme von Kohlenstoffmonoxid und Kohlenstoffdioxid in Gegenwart von Wasserstoff und ihre Umsetzung zu Ethanol und Essigsäure über Acetyl-CoA erfolgt über den reduktiven Acetyl-CoA-Weg. Dieser ist nach seinen Hauptentdeckern Harland G. Wood und Lars G. Ljungdahl auch als „Wood-Ljungdahl-Weg“ bekannt.[4][5] Neben Kohlenstoff ist dabei die Gabe von Vitaminen sowie von Proteinen (z. B. als Hefeextrakt) als Stickstoffquelle notwendig.[2] Die Bakterien produzieren während ihrer Wachstumsphase vor allem Essigsäure, während Ethanol vor allem während der stationären Phase gebildet wird. Daneben ist das Hauptprodukt abhängig vom pH-Wert des Substrats: Während die Bakterien bei höheren pH-Werten zwischen 5 und 7 vor allem Essigsäure bilden, produzieren sie bei niedrigeren pH-Werten zwischen 4 und 4,5 vor allem Ethanol.[6][3]
Die biochemische Umsetzung von Kohlenstoffmonoxid und Kohlenstoffdioxid erfolgt dabei entsprechend den folgenden Reaktionsgleichungen:[7]
- Für Ethanol:
- Für Essigsäure:
Bei einem Wasserstoff/Kohlenstoffdioxid-Gemisch werden durch Clostridium ljungdahlii 4 mmol Wasserstoff und 2 mmol Kohlenstoffdioxid in 1 mmol Essigsäure umgesetzt, während bei einem Fructose-Medium eine Umsetzung von 1 mmol Fructose in durchschnittlich 2,44 mmol Essigsäure erfolgt. Die Umsetzung ähnelt derjenigen von anderen acetogenen Bakterien: Acetobacterium carbinolicum setzt ein Molekül Fructose in 2,1 bis 2,3 Moleküle Essigsäure um, Acetobacterium woodi in 2,2 bis 2,8; bei Clostridium thermoaceticum erfolgt eine Umsetzung von einem Molekül Glucose in durchschnittlich 2,55 und bei Clostridium thermoautotrophicum in 2,5 Moleküle Essigsäure.[2] Kulturen zur Herstellung von Ethanol aus Synthesegas produzieren in der Regel bis etwa 2 mmol Ethanol aus 1 mmol Fructose.[2] Über den pH-Wert lässt sich allerdings das Wachstum der Bakterien und die Produktionsrate für Ethanol steuern – so konnte nachgewiesen werden, dass bei einem pH von 7,8 im Vergleich zu pH 5,5 die Zelldichte um etwa das 1,5fache erhöht ist und sich zugleich die im Medium enthaltene Ethanolmenge um etwa 110 % erhöht.[8]
Genetik
Das Genom des Bakteriums wurde 2010 als zweites Genom eines acetogenen Bakteriums nach Moorella thermoacetica vollständig sequenziert. Es weist 4.630.065 bp auf und gehört damit zu den größten der bekannten Genome innerhalb der Clostridien. 25,2 % der 4.184 identifizierten Gene besitzen keine bekannte Funktion. Bemerkenswerterweise liegen mehr als 3/4 der codierenden Sequenzen auf einem Strang der Doppelhelix. Zu den identifizierten Genen gehören die Gene für die Biosynthese der Flagellen, die in zwei Clustern angelegt sind, sowie ein benachbarter Gencluster für die Chemotaxis der Bakterien. Des Weiteren wurde das Gen spo0A nachgewiesen, welches das gleichnamige, für die Sporenbildung notwendige Regulationsprotein Spo0A codiert. Zudem konnten die Gene der für die Sporulation typischen Sigma-Faktoren nachgewiesen werden, während die Gene spo0F und spo0B wie bei allen sequenzierten Clostridien fehlen.[1]
Die Arbeit von Köpke und Mitarbeitern konzentrierte sich vor allem auf die codierenden Sequenzen, die für die Stoffwechseleigenschaften des Bakteriums interessant sind. Dabei konnte nachgewiesen werden, dass Clostridium ljungdahlii über ein spezifisches Rnf-System verfügt. Es weist damit einen gegenüber anderen bekannten acetogenen Bakterien modifizierten Stoffwechsel auf, der für die Energiebereitstellung weder ein Cytochrom-System noch Natriumionen benötigt.[1]
Ökologie
Über die Ökologie von Clostridium ljungdahlii liegen nur sehr wenige Daten vor, da das Bakterium nach seiner Isolierung ausschließlich unter Laborbedingungen untersucht wurde. Es lebt obligat anaerob, benötigt also zur Bildung von Fortpflanzungszellen ein sauerstofffreies Substrat. Unter aeroben Bedingungen bildet es nach wenigen Stunden Endosporen, die mehrere Jahre als Überdauerungsstadien auch in sauerstoffreichen Substraten überleben und unter anaeroben Bedingungen wieder zu vegetativen Bakterien werden.
Der Stamm ATCC 49587T, der zur Erstbeschreibung genutzt wurde, wurde im Abfall eines Hühnerzuchtbetriebs isoliert.[2]
Systematik
Die wissenschaftliche Erstbeschreibung von Clostridium ljungdahlii erfolgte 1993 durch die Arbeitsgruppe um Ralph S. Tanner von der University of Oklahoma.[2] Namensgebend ist Lars G. Ljungdahl, der zentrale Arbeiten zur Aufklärung des Stoffwechsels acetogener Bakterien sowie der Clostridien im Allgemeinen veröffentlicht hat und gemeinsam mit Harland G. Woods auch Namensgeber des Woods-Ljungdahl-Stoffwechselweges ist.
Clostridium ljungdahlii wird taxonomisch der mit über 160 beschriebenen Arten sehr artenreichen Gattung der Clostridien zugeordnet. Dabei stellt die Gruppe der Clostridien eine sehr diverse Gruppe dar, deren phylogenetische Klassifizierung und eventuelle Aufspaltung in mehrere Gattungen in stetiger Diskussion ist.[9][10] Die Unterscheidung der Arten innerhalb der Gattung beruht vor allem auf der molekularbiologischen Analyse der 23S rRNA, nach der die Gattung in 12 Homologie-Gruppen aufgeteilt wird. Clostridium ljungdahlii stellt dabei das erste bekannte acetogene Bakterium in der Homologie-Gruppe I dar.[2] Es besteht eine enge Verwandtschaft mit Clostridium tyrobutyricum, einem Essig- und Buttersäure produzierenden Bakterium, und Clostridium pasteurianum, die durch einen Vergleich der Sequenz 16S rRNA nachgewiesen wurde.[2] Das ebenfalls als eigene Art beschriebene Clostridium autoethanogenum ist wahrscheinlich ein Synonym von Clostridium ljungdahlii, da es von diesem nicht unterscheidbar ist.[10]
Technische Bedeutung und Forschungsgeschichte
Das Bakterium wurde von Suhakar Barik von der University of Arkansas aufgrund seiner physiologischen Eigenschaften isoliert. Er suchte gezielt nach Bakterien, die in der Lage sind, Synthesegas aus der Kohlevergasung für die Produktion potenziell interessanter Produkte zu nutzen.[2] Gemeinsam mit J. L. Gaddy, ebenfalls an der University of Arkansas, und weiteren Forschern konnte Barik bereits 1988 die Ethanolproduktion durch die von ihm isolierte und der Gattung Clostridium zugeordnete Bakterienkultur nachweisen.[11] Ralph S. Tanner und D. Yang stellten die neue Art 1990 anhand Bariks Kultur als Clostridium ljungdahlii PETCT in einer Präsentation und einem Abstract auf der 90. Jahrestagung der American Society for Microbiology in Washington D.C. erstmals wissenschaftlich vor.[12] Die offizielle Erstbeschreibung erfolgte durch Tanner und Mitarbeitern erst 1993 als Clostridium ljungdahlii Stamm ATCC 49587T. Unter dieser Bezeichnung wurde die Kultur in die American Type Culture Collection aufgenommen.[2] Bereits 1992 patentierte Gaddy die technische Ethanol- und Essigsäureproduktion mit der Bakterienkultur.[3] In den Folgejahren wurden mit den Stämmen DSM 13528 und PETC weitere Stämme isoliert und kultiviert.[13] 2010 wurde das Genom des Stamms DSM 13528 sequenziert.[1]
Insbesondere aufgrund der potenziellen Nutzung von Wasserstoff/Kohlenstoffmonoxid (Synthesegas-Fermentation) und Wasserstoff/Kohlenstoffdioxid als Wachstumssubstrat besteht ein sehr großes wirtschaftliches Interesse an dem Bakterium für die Nutzung im Bereich der Industriellen Biotechnologie. Es soll zur Herstellung von Ethanol, Butanol und Essigsäure genutzt werden,[1] wobei eine Nutzung zur Butanolherstellung erst durch eine Genübertragung der für die Butanolherstellung wichtigen Gene von Clostridium acetobutylicum ermöglicht wurde.[14][15] Es handelt sich hierbei um die Gene bcd für eine Butyryl-CoA-Dehydrogenase, hbd für eine 3-Hydroxybutyryl-CoA-Dehydrogenase, thlA für eine Thiolase, bdha für eine Butanol-Dehydrogenase und adhE für eine Butyraldehyd-/Butanol-Dehydrogenase.[14][15] Nach dem Einbau des artfremden Plasmids mit Hilfe einer Elektroporation konnte bei dem neu entwickelten Stamm eine Butanolproduktion auf der Basis von Synthesegas festgestellt werden, die in weiteren Schritten optimiert werden soll.[15]
Im Gegensatz zu anderen acetogenen Bakterien wird Clostridium ljungdahlii bereits technisch zur Ethanolproduktion genutzt, wobei die Synthesegas-Fermentation mit einer Biomassevergasung verbunden wird. Anlagen der Unternehmen INEOS Bio in Großbritannien, LanzaTech in Neuseeland und China sowie BriEnergy und Coskata in den Vereinigten Staaten stehen im Pilot- und Demonstrationsmaßstab zur Verfügung.
Einzelnachweise
- Michael Köpke, Claudia Held, Sandra Hujer, Heiko Liesegang, Arnim Wiezer, Antje Wollherr, Armin Ehrenreich, Wolfgang Liebl, Gerhard Gottschalk, Peter Dürre: Clostridium ljungdahlii represents a microbial production platform based on syngas. In: Proceedings of the National Academy of Sciences of the United States of America. Band 107, Nr. 29, 2010, ISSN 1091-6490, S. 13087–13092, doi:10.1073/pnas.1004716107, PMID 20616070.
- Ralph S. Tanner, Letrisa M. Miller, Decheng Yang: Clostridium ljungdahlii sp. nov., an Acetogenic Species in Clostridial rRNA Homology Group I. In: International Journal of Systematic and Evolutionary Microbiology,. Band 43, Nr. 2, 1993, ISSN 1466-5026, S. 232–236, doi:10.1099/00207713-43-2-232.
- Patent US5173429: Clostridium ljungdahlii, an anaerobic ethanol and acetate producing micoorganism.. Veröffentlicht am 22. Dezember 1992, Erfinder: J.L. Gaddy, E.C. Clausen.
- L. G. Ljungdahl: The autotrophic pathway of acetate synthesis in acetogenic bacteria. In: Annual Review of Microbiology. Band 40, 1986, ISSN 0066-4227, S. 415–450, doi:10.1146/annurev.mi.40.100186.002215, PMID 3096193.
- Harland G. Wood, Steve W. Ragsdale, Ewa Pezacka: The acetyl-CoA pathway of autotrophic growth. In: FEMS Microbiology Letters. Band 39, Nr. 4, 1986, ISSN 0378-1097, S. 345–362, doi:10.1111/j.1574-6968.1986.tb01865.x.
- Asma Ahmed, Allyson White, Peng Hu, Randy Lewis, Raymond Huhnke: Ethanol from Syngas – Microbial Conversion. (Memento vom 26. Juni 2010 im Internet Archive) SynGrant BioWeb, 2008.
- Pradeep Chaminda Munasinghe, Samir Kumar Khanal: Biomass-derived syngas fermentation into biofuels: Opportunities and challenges. In: Bioresource Technology. Band 101, Nr. 13, 2010, ISSN 1873-2976, S. 5013–5022, doi:10.1016/j.biortech.2009.12.098, PMID 20096574.
- Jacqueline L. Cotter, Mari S. Chinn, Amy M. Grunden: Influence of process parameters on growth of Clostridium ljungdahlii and Clostridium autoethanogenum on synthesis gas. In: Enzyme and Microbial Technology. Band 44, Nr. 5, 2009, ISSN 0141-0229, S. 281–288, doi:10.1016/j.enzmictec.2008.11.002.
- M. D. Collins, P. A. Lawson, A. Willems, J. J. Cordoba, J. Fernandez-Garayzabal, P. Garcia, J. Cai, H. Hippe, J. A. E. Farrow: The Phylogeny of the Genus Clostridium: Proposal of Five New Genera and Eleven New Species Combinations. In: International Journal of Systematic and Evolutionary Microbiology,. Band 44, Nr. 4, 1994, ISSN 1466-5026, S. 812–826, doi:10.1099/00207713-44-4-812.
- E. Stackebrandt, I. Kramer, J. Swiderski, H. Hippe: Phylogenetic basis for a taxonomic dissection of the genus Clostridium. In: FEMS immunology and medical microbiology. Band 24, Nr. 3, 1999, ISSN 0928-8244, S. 253–258, doi:10.1111/j.1574-695X.1999.tb01291.x, PMID 10397308.
- S. Barik, S. Prieto, S. B. Harrison, E. C. Clausen, J. L. Gaddy: Biological production of alcohols from coal through indirect liquefaction. In: Applied Biochemistry and Biotechnology. Band 18, Nr. 1, 1988, ISSN 1559-0291, S. 363–378, doi:10.1007/BF02930840.
- Ralph S. Tanner, Letrisa M. Miller, Decheng Yang: Clostridium ljungdahlii sp. nov., an Acetogenic Species in Clostridial rRNA Homology Group I. In: International Journal of Systematic and Evolutionary Microbiology,. Band 43, Nr. 2, 1993, ISSN 1466-5026, S. 232–236, doi:10.1099/00207713-43-2-232.
- UniProt.: "Clostridium ljungdahlii", aufgerufen am 1. März 2011.
- Michael Köpke: Genetische Veränderung von Clostridium ljungdahlii zur Produktion von 1-Butanol aus Synthesegas. Dissertation der Fakultät für Naturwissenschaften der Universität Ulm, 2009. (Volltext; PDF; 10,0 MB)
- Michael Köpke, Sandra Hujer, Peter Dürre: Klimaschutz durch innovative Biotechnologie. Laborwelt 6/2009.
Weblinks
- Clostridium ljungdahlii auf der Website des National Center for Biotechnology Information (NCBI)
- GenBank-Eintrag CP001666 für Clostridium ljungdahlii DSM 13528, complete genome auf der Website des National Center for Biotechnology Information (NCBI)