Zobellviridae
Zobellviridae ist die Bezeichnung für eine 2021 vom International Committee on Taxonomy of Viruses (ICTV) neu eingerichtete Familie von Viren mit sog. Kopf-Schwanz-Aufbau (Ordnung Caudovirales).[3]
| Zobellviridae | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|

| ||||||||||||||
| Systematik | ||||||||||||||
| ||||||||||||||
| Taxonomische Merkmale | ||||||||||||||
| ||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||
| Zobellviridae | ||||||||||||||
| Links | ||||||||||||||
|
Systematik
Die Familie Zobellviridae setzt sich nach ICTV Master Species List (MSL) #36 mit Stand Ende Juni 2021 wie folgt zusammen (von den Spezies ist meist nur eine Auswahl angegeben):[2][3]
Familie Zobellviridae (veraltet RIO-Klade, en. RIO clade)
- Unterfamilie Cobavirinae (veraltet Cobavirus-Gruppe, Cobavirus group)
- Gattung Siovirus (im Gegensatz zu früheren Vorschlägen im engeren Sinn. Die provisorische Gattung „Siovirus“ im ursprünglichen, weiteren Sinn war äquivalent zur „SIO-Klade“, en. „SIO clade“)
- Spezies Celeribacter-Virus P12053L (en. Celeribacter virus P12053L)
- Spezies Lentibacter-Virus ICBM1 (en. Lentibacter virus ICBM1, früher Lentibacter virus vB_LenP_ICBM1),[1] mit Referenzstamm Lentibacter-Phage vB_LenP_ICBM1 (ICBM1) und Stamm Lentibacter-Phage vB_LenP_ICBM3 (ICBM3).
- Spezies Roseobacter-Virus SIO1 (en. Roseobacter virus SIO1), mit Bakteriophage SIO1[4][5]
- Gattung Veravirus
- Gattung Siovirus (im Gegensatz zu früheren Vorschlägen im engeren Sinn. Die provisorische Gattung „Siovirus“ im ursprünglichen, weiteren Sinn war äquivalent zur „SIO-Klade“, en. „SIO clade“)
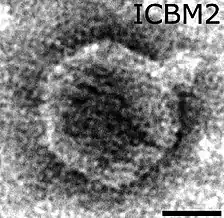
- Spezies Lentibacter-Virus ICBM2 (en. Lentibacter virus ICBM2, früher Lentibacter virus vB_LenP_ICBM2)[1]
- Unterfamilie nicht bestimmt
- Gattung Citrovirus
- Spezies Citrobacter-Virus CVT22 (en. Citrobacter virus CVT22)
- Gattung Icepovirus
- Spezies Vibrio-Virus ICP2 (en. Vibrio virus ICP2)
- Gattung Melvirus (wohl zu unterscheiden von dem Nucleocytoviricota-Kandidaten „Meelsvirus“, einem Riesenvirus, das den Pfeilwurm Adhesisagitta hispida infiziert)
- Spezies Pseudoalteromonas-Virus HP1 (en. Pseudoalteromonas virus HP1), mit (Referenz-)Stamm Pseudoalteromonas-Phage HP1
- Spezies Pseudoalteromonas-Virus RIO1 (en. Pseudoalteromonas virus RIO1)
- Gattung Paundecimvirus
- Spezies Pseudomonas-Virus PA11 (en. Pseudomonas virus PA11)
- Gattung Salinovirus
- Spezies Salinivibrio-Virus CW02 (en. Salinivibrio virus CW02), mit Salinivibrio-Phage CW02 (alias Halophage CW02)
- Gattung Vipivirus
- Spezies Vibrio-Virus VpV262 (en. Vibrio virus VpV262), mit Bakteriophage VpV262[4]
- Gattung Citrovirus
Beschreibung
Unterfamilie Cobavirinae
Die Phagen der vorgeschlagenen Unterfamilie Cobavirinae haben eine konservierte Genom-Organisation. Alle haben an den Enden des Genoms direkte terminale Wiederholungen (englisch direct terminal repeats, DTRs). Dies deutet darauf hin, dass die Cobavirinae eine Verpackungsstrategie ähnlich der des T7-Phagen verwenden könnten. Die Gene sind in zwei Armen der Genomischen DNA mit entgegengesetzten Transkriptionsrichtungen organisiert. Auf dem linken Arm werden Gene für die Replikation und den Nukleotid-Stoffwechsel kodiert, während der rechte Arm Gene für die Lyse und die Struktur und Morphologie des Virions (Kapsid etc.) kodiert.[1][3]
- Gattung Siovirus:
Lentibacter-Phage ICBM1 wurde (wie ICBM2, s. u.) 2019 aus einer Wasserprobe der südlichen Nordsee angereichert, die während einer Algenblüte gesammelt wurde. Der Stamm ICBM3 wurde durch Sequenzierung der Phagenanreicherung gewonnen. Die Transmissionselektronenmikroskopie (TEM) zeigte, dass Phage ICBM1 ein isometrisches Kapsid (d. h. mit ideale-regelmäßigem Ikosaeder) mit hexagonalen Querschnitten von 58,7 ± 3,7 nm und einem kurzen Schwanz besitzt, d. h, eine podovirenartige Morphologie. Im aus den Genomdaten ermittelten Phylogenetischen Baum bildeten die Phagen ICBM1, ICBM2 und ICBM3 zusammen mit dem Roseobacter-Virus SIO1 und seinen Verwandten, dem Celeribacter-Phagen P12053L und mehreren anderen Genomen aus der Metagenomik von Umweltproben eine stark unterstützte Klade. Innerhalb dieser hatten bei den Analysen alle Phagen mit Ausnahme von zwei Umweltgenomen eine Cobalamin-abhängige Ribonukleotidreduktase. ICBM1 infiziert nur Lentibacter sp. SH36 ([en]), den Wirt der ursprünglichen Isolation. ICBM1 hat eine Genomgröße von 40.163 bp (Basenpaaren) und einen G+C-Gehalt von 47,0 %, ICBM3 hat eine Genomgröße von 40.498 bp und einen G+C-Gehalt von 47,30 %.[1][3]
Celeribacter-Phage P12053L hat ein dsDNA-Genom von 35.889 bp Länge, mit einem G+C-Gehalt von 46,1 %.[3]
- Gattung Veravirus:
Lentibacter-ICBM2 wurde (wie ICBM1, s. o.) 2019 während einer Algenblüte aus einer Wasserprobe der südlichen Nordsee isoliert. ICBM2 hat ebenfalls ein isometrisches Kapsid mit hexagonalen Querschnitten von hier 59,2 ± 2,8 nm mit podovirenartig kurzer Schwanzstruktur. ICBM2 infiziert ebenfalls nur den ursprünglichen Isolationswirt Lentibacter sp. SH36. Die Genomgröße beträgt 40.907 bp, der G+C-Gehalt 47,8 %.[1][3]
Mitglieder ohne zugeordnete Unterfamilie
- Gattung Melvirus:
Der Pseudoalteromonas-Phage HP1 wurde 2017 aus Meerwasserproben aus der Nordsee bei Helgoland, Deutschland, isoliert. Er infiziert zwei eng verwandte Linien von Pseudoalteromonas, Pseudoalteromonas sp. H-100[6] und Pseudoalteromonas sp. 13–15.[7] Es hat ebenfalls eine podovirale Morphologie mit einem ikosaedrischen Kapsid und einem kurzen Schwanz. Das Genom hat eine Größe von 45.035 bp, der G+C-Gehalt beträgt 44,67 %.[3]
Der Pseudoalteromonas-Phage RIO-1 wurde 2013 zusammen mit seinem Wirt Pseudoalteromonas marina CL-E25 [en][8] aus Meerwasserproben des Koreanischen Ostmeeres (Japanisches Meer, zwischen Korea, Japan und Südostsibirien) isoliert. Sein ikosaedrischer Kopf hat einen Durchmesser von 51 nm und eine podovirenartig kurze Schwanzstruktur. Sein Genom ist 43.882 bp groß mit einem G+G-Gehalt von 39,6 % und direkten terminalen Wiederholungen direkte terminale Wiederholungen (DTRs).[3]
- Gattung Paundecimvirus:
Phage PA1 infiziert Pseudomonas aeruginosa und hat eine Genomgröße von 49.639 bp mit einem G+C-Gehalt von 44,8 %.[3]
- Gattung Citrovirus:
Phage CVT22 infiziert Citrobacter sp. TM1552.[9] Zusammen mit seinem Wirt wurde er isoliert aus dem Darm von Termiten der Spezies Coptotermes formosanus [en] (Rhinotermitidae) isoliert. Er hat die übliche podovirale Morphologie und ein zirkuläres permutiertes Genom (en. circular permuted genome) von 47.636 bp mit 41,6 % G+C-Gehalt.[3]
- Gattung Salinovirus:
Der Salinivibrio-Phage CW02 infiziert das Salinivibrio-costicola-ähnliche Bakterium SA50.[10] Nächster bekannter Verwandter von SA50 ist der Stamm S. costicola subsp. costicola ATCC 33508,[11][12] ein Bakterium der Familie Vibrionaceae (Gammaproteobacteria). Phage CW02 wurde 2012 mit seinem Wirt im Großen Salzsee, USA, isoliert. Der ikosaedrische Kopf von CW02 hat ein Durchmesser von ca. 60 nm Durchmesser und einen podovirenartig kurzen Schwanz. Das Genom des Phagen CW02 ist 40.547 bp groß mit 47,67 % G+C-Gehalt. Es gibt keine terminalen Wiederholungen.[13][3]
- Gattung Icepovirus:
Vibrio-Phage ICP2 infiziert verschiedene Stämme von Vibrio cholerae. Er wurde aus Stuhlproben von Cholera-Patienten in Bangladesch isoliert, mit dem ursprünglichen Wirt V. cholerae O1 El Tor (alias O1 N16961).[14] Er kann auch den Stamm V. cholerae O139 M010[15] infizieren. Das ikosaedrisches Kapsid von ICP2 hat einen Durchmesser von 60 nm und den podovirenartigen kurzen Schwanz, 13 nm lang, 8 nm im Durchmesser (Breite). Sein Genom hat eine Länge von 49.675 bp und 42,7 % G+C-Gehalt.[3]
Etymologie
- Familie Zobellviridae: Benannt zu Ehren von Claude Ephraim ZoBell, der als erster marine Bakteriophagen isolierte.[3]
- Unterfamilie Cobavirinae: Von Cobalamin wegen der im Phagengenom kodierten Cobalamin-abhängigen Ribonukleotid-Reduktase.[3]
- Gattung Siovirus: Der Gattungsname leitet sich vom Roseobacter-Phagen SIO1 ab, dem ersten beschriebenen Phagen aus dieser Gattung und dem ersten sequenzierten marinen Bakteriophagen überhaupt.[3]
- Gattung Veravirus: Benannt zu Ehren der Forscherin Vera Bischoff, die den Lentibacter-Phagen ICBM2 isolierte.
- Gattung Melvirus: Benannt zu Ehren der Forscherin Melissa Duhaime, die es beschrieben hat.
- Gattung Citrovirus: Der Name leiet sich ab von den Wirtsbakterien dieser Phagen, der Gattung Citrobacter.
- Gattung Salinovirus: Der Name leiet sich ab von den Wirtsbakterien dieser Phagen, der Gattung, Salinivibrio.
Einzelnachweise
- Vera Bischoff, Boyke Bunk, Jan P. Meier-Kolthoff, Cathrin Spröer, Anja Poehlein, Marco Dogs, Mary Nguyen, Jörn Petersen, Rolf Daniel, Jörg Overmann, Markus Göker, Meinhard Simon, Thorsten Brinkhoff, Cristina Moraru: Cobaviruses – a new globally distributed phage group infecting Rhodobacteraceae in marine ecosystems. In: Nature ISME Journal, Band 13, S. 1404–1421, 4. Februar 2019, doi:10.1038/s41396-019-0362-7
- ICTV Master Species List 2020.v1. ICTV, New MSL including all taxa updates since the 2019 release, March 2021 (MSL #36)
- Evelien M. Adriaenssens, I. Tolstoy, M. Łobocka, Liliana Cristina Moraru, Jakub Barylski, Y. Tong, A. M. Kropinski: 2020.187B.R.Zobellviridae.zip (docx, xlsx), Vorschlag an das ICTV: Create one new family (Zobellviridae) including one new subfamily (Cobavirinae), seven new genera and 12 new species (Caudovirales), November 2020
- R. Lavigne, D. Seto, P. Mahadevan, H.-W. Ackermann, A. M. Kropinski: Unifying classical and molecular taxonomic classification: analysis of the Podoviridae using BLASTP-based tools. In: Research in Microbiology. Band 159, Nr. 5, 2008, S. 406–414, doi:10.1016/j.resmic.2008.03.005, PMID 18555669.
- Jacqueline Z.-M. Chan, Andrew D. Millard, Nicholas H. Mann, Hendrik Schäfer: Comparative genomics defines the core genome of the growing N4-like phage genus and identifies N4-like Roseophage specific genes (PDF; 3,9 MB) In: Frontiers in Microbiology, Band 5, Nr. 506, 10. Oktober 2014, doi:10.3389/fmicb.2014.00506
- NCBI: Pseudoalteromonas sp. H100 (species)
- NCBI: Pseudoalteromonas sp. 13-15 (species)
- NCBI: Pseudoalteromonas marina Nam et al. 2007 (species)
- NCBI: Citrobacter sp. TM1552 (species)
- NCBI: uncultured gamma proteobacterium SA50 (species)
- NCBI: Salinivibrio costicola subsp. costicola ATCC 33508 = LMG 11651
- Ida Romano et al.: Salinivibrio costicola subsp. alcaliphilus subsp. nov., a haloalkaliphilic aerobe from Campania Region (Italy). In: Systematic and Applied Microbiology, Band 28, Nr. 1, 28. Januar 2005, S. 34–42, doi:10.1016/j.syapm.2004.10.001
- Peter S. Shen, Matthew J. Domek, Eduardo Sanz-García, Aman Makaju, Ryan M. Taylor, Ryan Hoggan, Michele D. Culumber, Craig J. Oberg, Donald P. Breakwell, John T. Prince, David M. Belnap: Sequence and structural characterization of great salt lake bacteriophage CW02, a member of the T7-like supergroup. In: Journal of Virology, Band 86, Nr. 15, 11. Juli 2012, S. 7907–7917, Epub 16. Mai 2012, doi:10.1128/jvi.00407-12, PMID 22593163, PMC 3421657 (freier Volltext). Mit T7 supergroup sind offenbar die podovirenartigen Phagen gemeint, Phage T7 gehört zu den schon früher aus den Podoviridae ausgegliederten Familie Autographiviridae.
- NCBI: Vibrio cholerae O1 biovar El Tor str. N16961 (strain)
- NCBI: Vibrio cholerae O139 (serogroup)