Crassvirales
„Crassvirales“ (provisorisch auch „crAss-like phages“, auch „crAss-like viruses“, deutsch etwa: „crAss-ähnliche Phagen“, kurz „crAssphagen“) ist die vorgeschlagene Bezeichnung für eine Verwandtschaftsgruppe (Klade) von Bakteriophagen (Viren, die Bakterien infizieren) und 2014 durch rechnergestützte Analyse öffentlich zugänglicher wissenschaftlicher Daten zu Genomen im menschlichen Stuhl entdeckt wurde.[4][5]< Das zirkuläre Doppelstrang-DNA-Genom dieses ersten Vertreters crAssphage (ΦCrAss) ist etwa 97 kbp (Kilo-Basenpaare) groß und enthält nach Vorhersage 80 offene Leserahmen (englisch Open reading Frames, ORFs).[4][3] Derartige Sequenzen sind häufig in menschlichen Stuhlproben zu finden.[4] Zum Zeitpunkt der Entdeckung wurde vorausgesagt, dass crAssphagen Bakterien des Phylums (Abteilung) Bacteroidetes infiziert, die im Darmtrakt vieler Tiere häufig vorkommen, einschließlich des Menschen.[4] Seitdem wurde der erste Bakteriophage dieser Gruppe in vitro isoliert und es wurde bestätigt, dass er Bacteroides intestinalis infiziert.[6] Weitere Vertreter der Gruppe infizieren Bacteroidetes-Bakterien der Gattungen Azobacteroides ([en], Bacteroidales), Cellulophaga ([en], Flavobacteriales) und Flavobacterium (Flavobacteriales).[7]
| „Crassvirales“ | ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|
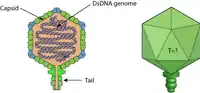
crAssphage (Schemazeichnung) | ||||||||||
| Systematik | ||||||||||
| ||||||||||
| Taxonomische Merkmale | ||||||||||
| ||||||||||
| Wissenschaftlicher Name | ||||||||||
| „Crassvirales“ | ||||||||||
| Links | ||||||||||
|
Basierend auf der Analyse von Metagenomdaten wurden bei etwa der Hälfte aller untersuchten Menschen crAssphage-Sequenzen identifiziert.[4] Das Virus bzw. die Gruppe wurde nach der Software crAss (cross-assembly) benannt,[8][9] mit der das virale Genom gefunden wurde. Damit sind die crAssphagen möglicherweise die ersten Organismen, die zu Werbezwecken nach einem Computerprogramm benannt wurde.[10]
Im März 2021 wurde vorgeschlagen, die bisherige Ordnung Caudovirales im Phylum Caudoviricetes aufzulösen und die Klade der crAssphagen selbst in den Rang einer Ordnung „Crassvirales“ mit sechs Familien zu erheben.[2] Diese Klade (Verwandtschaftsgruppe) von Bakterienviren ist wohl zu unterscheiden vom Phylum Cressdnaviricota der CRESS-DNA-Viren mit einem zirkulären Einzelstrang-DNA-Genom, deren Wirte Eukaryoten (möglicherweise auch Archaeen) sind.
Beschreibung
CrAssphagen benutzen ein eigenes Enzym (RNA-Polymerase), um RNA-Kopien ihrer Gene zu erstellen. Da alle Zellen, von Bakterien bis hin zum Menschen mit Hilfe solcher RNA-Polymerase RNA-Kopien (mRNA) ihrer Gene herstellen, sind diese Enzyme sehr alt und in allen Lebewesen auch sehr ähnlich (hoch konserviert). Die RNA-Polymerase der crAssphagen ähnelt dagegen eher einem Enzym im Menschen und anderen höheren Organismen, das an der RNA-Interferenz beteiligt ist.[11]
Forschungsgeschichte
Zum Zeitpunkt seiner Entdeckung im Jahr 2014 hatte crAssphage keine bekannten Verwandten. Im Jahr 2017 wurde jedoch eine Reihe verwandter Viren entdeckt.[12] Basierend auf einem Screening verwandter Sequenzen in öffentlichen Nukleotiddatenbanken und einer phylogenetischen Analyse wurde zunächst vorgeschlagen, dass crAssphage Teil der expansiven Phagenfamilie Podoviridae (Ordnung Caudovirales) ist. Die Podoviren schienen in ihrer Morphologie (Kopf-Schwanz-Architektur mit kurzer Schwanzstruktur) mit der tatsächlichen oder vermuteten Erscheinungsform von crAssphage und den anderen „crAss-like phages“ übereinzustimmen. Das Genom der Podoviren crAssphagen ist ein Doppelstrang-DNA-Molekül. Hauptunterschied ist, dass dieses bei den Podoviren linear vorliegt, bei den crAssphagen aber zirkulär.[13][3][14][15]
Vertreter wurden in verschiedenen Umgebungen gefunden:
- dem menschlichem Darm und Kot
- dem Termitendarm,[12][16][17]
- in terrestrischen Umgebungen und im Grundwasser
- in Natronseen (hypersaline Sole)
- im marinen Sediment
- an Pflanzenwurzeln[12]
Die Klade der crAssphagen (crAss-like viruses) umfasst nach früheren Schätzungen etwa (mindestens) 10 Gattungen.[3]
Nachdem man zunächst vorgeschlagen hatte, die crAssphagen (etwa als Unterfamilie) der Familie Podoviridae unterzuordnen, zielen neuere Vorschläge darauf ab, diese wegen ihrer hohen Diversität untereinander und der genomischen Unterschiede zu den anderen Vertretern der Caudovirales trotz der podovirenartigen äußeren Erscheinung als eine eigene Ordnung „Crassvirales“ innerhalb der gemeinsamen Klasse Caudoviricetes einzuordnen.
Systematik
Mit Stand Juni 2021, d. h. mit Master Species List (MSL) #36, waren vom International Committee on Taxonomy of Viruses (ICTV) weder die Klade als Ganzes, noch einzelne Vertreter (Gattungen oder Spezies) offiziell bestätigt.[1]
Ordnung „Crassvirales“ (alias crAssphagen, en. „crAss-like phages“), vorgeschlagen mit 6 Familien, 10 ausgewiesenen Unterfamilien und insgesamt 78 Gattungen (nebst einer Auswahl von Spezies):[7][13][3][15][2][18]
- Familie „Crevaviridae“
- Unterfamilie „Coarsevirinae“
- Gattung „Junduvirus“
- Unterfamilie „Doltivirinae“
- Gattung „Kingevirus“
- Familie „Intestiviridae“
- Unterfamilie „Churivirinae“
- Gattung „Jahgtovirus“
- Spezies „Jahgtovirus cr107“, mit Phage cr107_1 (alias crAssphage cr107_1)[19]
- Unterfamilie „Crudevirinae“
- Gattung „Carjivirus“
- Gattung „Dabirmavirus“
- Gattung „Delmidovirus“
- Gattung „Diorhovirus“
- Spezies „Diorhovirus cr3“, mit Phage cr3_1 (alias crAssphage cr31_1)[23]
- Gattung „Drivevirus“
- Gattung „Earahsivirus“
- Gattung „Endlipuvirus“
- Gattung „Esdrolevirus“
- Unterfamilie „Obtuvirinae“
- Familie „Jelitoviridae“
- ohne zugewiesene Unterfamilie:
- Gattung „Ritroivirus“
- Gattung „Schipovirus“
- Gattung „Schnahavirus“
- Gattung „Schupivirus“
- Gattung „Sehjuvirus“
- Gattung „Serblevirus“
- Gattung „Shuhtzevirus“
- Gattung „Siluhkovirus“
- Gattung „Simuyevirus“
- Gattung „Sipelivirus“
- Gattung „Soilyhevirus“
- Gattung „Tehntovirus“
- Gattung „Tirverovirus“
- Gattung „Tohrnjavirus“
- Gattung „Wahchdevirus“
- Gattung „Wehumevirus“
- Familie „Steigviridae“
- ohne zugewiesene Unterfamilie:
- Gattung „Kahnovirus“
- Spezies „Kahnovirus cr85“, mit Phage cr85_1 (alias crAssphage cr85_1)[26]
- Gattung „Kehishuvirus“
- Gattung „Kolpuevirus“
- Spezies „Kolpuevirus cr126“, mit Phage cr126_1 (alias crAssphage cr126_1)[29]
- Gattung „Lahbrevirus“
- Gattung „Lahndsivirus“
- Spezies „Lahndsivirus cr111“, mit Phage cr111_1 (alias crAssphage cr111_1)[30]
- Gattung „Lebriduvirus“
- Gattung „Lehconavirus“
- Gattung „Liunbivirus“
- Gattung „Mahlemavirus“
- Spezies „Mahlunavirus cr128“, mit Phage cr128_1 (alias crAssphage cr128_1)[31]
- Gattung „Mahlunavirus“
- Gattung „Mahstovirus“
- Spezies „Mahstovirus cr106“, mit Phage cr106_1 (alias crAssphage cr106_1)[32]
- Gattung „Nowfuvirus“
- Gattung „Nurbavirus“
- Gattung „Pamirivirus“
- Spezies „Pamirivirus cr116“, mit Phage cr116_1 (alias crAssphage cr116_1)[33]
- Gattung „Paundivirus“
- Spezies „Paundivirus cr117“, mit IAS-Virus (en. IAS virus)[34]
- Gattung „Pedtovirus“
- Gattung „Penjavirus“
- Gattung „Picehavirus“
- Gattung „Piconjevirus“
- Gattung „Pipoluvirus“
- Spezies „Pipoluvirus cr108“, mit Phage cr108_1 (alias crAssphage cr108_1)[35]
- Gattung „Puadlovirus“
- Familie „Suoliviridae“
- Unterfamilie „Bearivirinae“
- Gattung „Afonbuvirus“
- Spezies „Afonbuvirus cr6“, mit Phage cr6_1 (alias crAssphage cr6_1)[36]
- Unterfamilie „Boorivirinae“
- Gattung „Canhaevirus“
- Spezies „Canhaevirus cr10“, mit Phage cr10_1 (alias crAssphage cr10_1)[37]
- Gattung „Cohcovirus“
- Spezies „Cohcovirus cr50“, mit Phage cr50_1 (alias crAssphage cr50_1)[38]
- Gattung „Culoivirus“
- Unterfamilie „Loutivirinae“
- Gattung „Blohavirus“
- Spezies „Blohavirus cr53“, mit Phage cr53_1 (alias crAssphage cr53_1)[41]
- Gattung „Buchavirus“
- Spezies „Buchavirus cr52“, mit Phage cr52_1 (alias crAssphage cr52_1)[42]
- Spezies „Buchavirus cr56“, mit Phage cr56_1 (alias crAssphage cr56_1)[43]
- Spezies „Buchavirus cr60“, mit Phage cr60_1 (alias crAssphage cr60_1)[44]
- Spezies „Buchavirus cr109“, mit Phage cr109_1 (alias crAssphage cr109_1)[45]
- Spezies „Buchavirus cr113“, mit Phage cr113_1 (alias crAssphage cr113_1)[46]
- Gattung „Buorbuivirus“
- Spezies „Buorbuivirus cr4“, mit Phage cr4_1 (alias crAssphage cr4_1)[47]
- Unterfamilie „Oafivirinae“
- Gattung „Belmavirus“
- Gattung „Bohxovirus“
- Gattung „Buhlduvirus“
- Gattung „Buorbuivirus“
- Gattung „Burzaovirus“
- Gattung „Cacepaovirus“
- Spezies „Cacepaovirus cr131“, mit Phage cr131_1 (alias crAssphage cr131_1)[52]
- Gattung „Cahvavirus“
- Gattung „Ceneivirus“
- Gattung „Cerpitivirus“
- Gattung „Chuhaivirus“
- Spezies „Chuhaivirus cr130“, mit Phage cr130_1 (alias crAssphage cr130_1)[53]
- Unterfamilie „Uncouvirinae“
- Familie „Tinaiviridae“
- ohne zugewiesene Unterfamilie:
- Gattung „Pasahvevirus“
- Gattung „Rodbovirus“
- Weitere Vorschläge zu crAss-like viruses nach NCBI ohne Zuordnung zu einer Familie, Unterfamilie oder Gattung (Auswahl):
- Spezies: „Azobacteroides-Phage ProJPt-Bp1“ (englisch „Azobacteroides phage ProJPt-Bp1“)[58]
- Spezies: „Bacteroides-Phage crAss002“ (en. „Bacteroides phage crAss002“)[59]
- Spezies „Cellulophaga-Phage phi14:2“ (en. „Cellulophaga phage phi14:2“)[60]
- Spezies „Flavobacterium-Phage vB_FspP_elemoA_1-5B“ (en. „Flavobacterium phage vB_FspP_elemoA_1-5B“)[61]
- Spezies „Marine virus AFVG_117M62“ (en. „Marine virus AFVG_117M62“)[62]
Gubaphagen
Camarillo-Guerrero, Almeida et al. beschrieben 2019/2020 die Ergebnisse ihrer Metagenomanalysen der menschlichen Darmflora hinsichtlich Bakteriophagen. Sie machen dabei eine weitere Klade, genannt „Gubaphagen“ (englisch Gut Bacteroidales phage, „Gubaphage clade)“ mit zwei Gattungen („G1“ – infiziert Bacteroides und „G2“ – infiziert Parabacteroides, [en]) aus, die nach den crAssphagen die zweithäufigsten Viren (d. h. Bakteriophagen) in dieser Umgebung darstellen. Die Merkmale der Gubaphagen erinnern dabei an die von „p-crAssphage“.[63][64]
Klinik
Es gibt keinen Hinweis darauf, dass crAssphagen an der menschlichen Gesundheit oder Krankheit direkt beteiligt sind,[65] sie beeinflussen aber als Bakteriophagen natürlich die menschliche Darmflora. Die Viren können in ihrer Eignung als Biomarker für die Kontamination des menschlichen Stuhls Indikatorbakterien übertreffen.[66][67][68]
Einzelnachweise
- ICTV: ICTV Master Species List 2020.v1, New MSL including all taxa updates since the 2019 release, March 2021 (MSL #36)
- Dann Turner, Andrew M. Kropinski, Evelien M. Adriaenssens: A Roadmap for Genome-Based Phage Taxonomy, in: MDPI Viruses Band 13, Nr. 3, Section Bacterial Viruses, 18. März 2021, 506, doi:10.3390/v13030506
- SIB: crAss-like phages, auf: Expasy ViralZone – Order: Caudovirales, Estimated about 10 genera
- Bas E. Dutilh, Noriko Cassman, Katelyn McNair, Savannah E. Sanchez, Genivaldo G. Z. Silva, Lance Boling, Jeremy J. Barr, Daan R. Speth, Victor Seguritan, Ramy K. Aziz, Ben Felts, Elizabeth A. Dinsdale, John L. Mokili, Robert A. Edwards: A highly abundant bacteriophage discovered in the unknown sequences of human faecal metagenomes. In: Nature Communications. 5, 2014, S. 4498. bibcode:2014NatCo...5.4498D. doi:10.1038/ncomms5498. PMID 25058116. PMC 4111155 (freier Volltext).
- CrAssphage: Previously Unknown Ancient Gut Virus Lives in Half World’s Population, auf: sci-news vom 11. August 2014 (englisch)
- A. N. Shkoporov, E. V. Khokhlova, C. B. Fitzgerald, S. R. Stockdale, L. A. Draper, P. Ross, C. Hill: ΦCrAss001 represents the most abundant bacteriophage family in the human gut and infects Bacteroides intestinalis. In: Nature Communications. 9, Nr. 1, 2018, S. 4781. bibcode:2018NatCo...9.4781S. doi:10.1038/s41467-018-07225-7. PMID 30429469. PMC 6235969 (freier Volltext).
- NCBI: crAss-like viruses/phages (clade)
- Bas E. Dutilh, Robert Schmieder, Jim Nulton, Ben Felts, Peter Salamon, Robert A. Edwards, John L. Mokili: Cross-Assembly of Metagenomes, in: Bioinformatics 2012. PMID 23074261
- Bas E. Dutilh, Robert Schmieder, Jim Nulton, Ben Felts, Peter Salamon, Robert A. Edwards, John L. Mokili: Reference-independent comparative metagenomics using cross-assembly: crAss. In: Bioinformatics. 28, Nr. 24, 2012, S. 3225–3231. doi:10.1093/bioinformatics/bts613. PMID 23074261. PMC 3519457 (freier Volltext).
- J. V. Chamary: A Common Virus Is Eating Your Gut Bacteria (en) In: Forbes. Abgerufen am 19. April 2019.
- Gut microbiome manipulation could result from virus discovery, auf: EurekAlert! vom 18. November 2020, Quelle: Rutgers University
- Natalia Yutin, Kira S. Makarova, Ayal B. Gussow, Mart Krupovic, Anca Segall, Robert A. Edwards, Eugene V. Koonin: Discovery of an expansive bacteriophage family that includes the most abundant viruses from the human gut. In: Nature Microbiology. 3, Nr. 1, 2018, S. 38–46. doi:10.1038/s41564-017-0053-y. PMID 29133882. PMC 5736458 (freier Volltext).
- SIB: Double Strand DNA Viruses, auf: ViralZone
- Eugene V. Koonin: Behind the paper: The most abundant human-associated virus no longer an orphan, 13. November 2017
- Eugene V. Koonin, Natalya Yutin: The crAss-like Phage Group: How Metagenomics Reshaped the Human Virome, in: Trends in Microbiology Band 28 Nr. 5, S: 349–359, 28. Februar/1. Mai 2020, doi:10.1016/j.tim.2020.01.010
- Chinmay V. Tikhe, Claudia Husseneder: Metavirome Sequencing of the Termite Gut Reveals the Presence of an Unexplored Bacteriophage Community. In: Frontiers in Microbiology. 8, 2018, ISSN 1664-302X, S. 2548. doi:10.3389/fmicb.2017.02548. PMID 29354098. PMC 5759034 (freier Volltext).
- Ajeng K. Pramono, Hirokazu Kuwahara, Takehiko Itoh, Atsushi Toyoda, Akinori Yamada, Yuichi Hongoh: Discovery and Complete Genome Sequence of a Bacteriophage from an Obligate Intracellular Symbiont of a Cellulolytic Protist in the Termite Gut. In: Microbes and Environments. 32, Nr. 2, 2017, ISSN 1342-6311, S. 112–117. doi:10.1264/jsme2.ME16175. PMID 28321010. PMC 5478533 (freier Volltext).
- A. N. Shkoporov, S. R. Stockdale, E. M. Adriaenssens, N. Yutin, E. V. Koonin, B. E. Dutilh, M. Krupovic, R. A. Edwards, I. Tolstoy, C. Hill: 2020.039B.Ud.v1.Crassvirales.docx, 2020.039B.Ud.v1.Crassvirales.xlsx, Proposal: Create one new order (Crassvirales) including six new families, ten new subfamilies, 78 new genera and 279 new species. 6. Dezember 2020
- NCBI: crAssphage cr107_1 (species)
- NCBI: uncultured crAssphage (species)
- NCBI: crAssphage cr11_1 (species)
- NCBI: crAssphage cr110_1 (species)
- NCBI: crAssphage cr3_1 (species)
- NCBI: crAssphage cr8_1 (species)
- NCBI: crAssphage cr271_1 (species)
- NCBI: crAssphage cr85_1 (species)
- NCBI: Bacteroides phage crAss001 (species)
- NCBI: crAssphage cr112_1 (species)
- NCBI: crAssphage cr126_1 (species)
- NCBI: crAssphage cr111_1 (species)
- NCBI: crAssphage cr128_1 (species)
- NCBI: crAssphage cr106_1 (species)
- NCBI: crAssphage cr116_1 (species)
- NCBI: IAS virus (species)
- NCBI: crAssphage cr108_1 (species)
- NCBI: crAssphage cr6_1 (species)
- NCBI: crAssphage cr10_1 (species)
- NCBI: crAssphage cr50_1 (species)
- NCBI: crAssphage cr1_1 (species)
- NCBI: crAssphage cr55_1 (species)
- NCBI: crAssphage cr53_1 (species)
- NCBI: crAssphage cr52_1 (species)
- NCBI: crAssphage cr56_1 (species)
- NCBI: crAssphage cr60_1 (species)
- NCBI: crAssphage cr109_1 (species)
- NCBI: crAssphage cr113_1 (species)
- NCBI: crAssphage cr4_1 (species)
- NCBI: crAssphage cr272_1 (species)
- NCBI: crAssphage cr273_1 (species)
- NCBI: crAssphage cr7_1 (species)
- NCBI: crAssphage cr124_1 (species)
- NCBI: crAssphage cr131_1 (species)
- NCBI: crAssphage cr130_1 (species)
- NCBI: crAssphage cr114_1 (species)
- NCBI: crAssphage cr125_1 (species)
- NCBI: crAssphage cr118_1 (species)
- NCBI: crAssphage cr115_1 (species)
- NCBI: Azobacteroides phage ProJPt-Bp1 (species)
- NCBI: Bacteroides phage crAss002 (species)
- NCBI: Cellulophaga phage phi14:2 (species)
- NCBI: Flavobacterium phage vB_FspP_elemoA_1-5B (species)
- NCBI: Marine virus AFVG_117M62 (species)
-
Luis Fernando Camarillo-Guerrero, Alexandre Almeida, Guillermo Rangel-Pineros, Robert D. Finn, Trevor D. Lawley: Massive expansion of human gut bacteriophage diversity, in: Cell Resource Band 184, Nr. 4, S. 1098–1109.e9, 18. Februar 2021, doi:10.1016/j.cell.2021.01.029. PrePrint vom 3. September 2020: bioRxiv, Europe PMC, doi:10.1101/2020.09.03.280214. Dazu:
- Martin Vieweg: Tausende Virenarten der Darmflora entdeckt, auf: wissenschaft.de vom 18. Februar 2021 (deutsch)
- Daniel Lingenhöhl: Virobiom: Darm beherbergt zehntausende unbekannte Virenarten, auf: spektrum.de vom 19. Februar 2021 (deutsch)
- Biologists Find Almost 143,000 Bacteriophage Species in Human Gut, auf: sci-news vom 19. Februar 2021 (englisch)
- Peter Dockrill: Scientists Find 140,000 Virus Species in The Human Gut, And Most Are Unknown, auf: sciencealert vom 28. Februar 2021 (englisch)
- Scientists identify more than 140,000 virus species in the human gut, auf: ScienceDaily vom 18. Februar 2021 (englisch)
- Luis Fernando Camarillo Guerrero: Integrative Analysis of the Human Gut Phageome Using a Metagenomics Approach, Doktorarbeit, Gonville & Caius College, University of Cambridge, August 2020, doi:10.17863/CAM.63973
- W. Ahmed, A. Lobos, J. Senkbeil, J. Peraud, J. Gallard, V. J. Harwood: Evaluation of the novel crAssphage marker for sewage pollution tracking in storm drain outfalls in Tampa, Florida. In: Water Research. 131, 2017, S. 142–150. doi:10.1016/j.watres.2017.12.011. PMID 29281808.
- C. García-Aljaro, E. Ballesté, M. Muniesa, J. Jofre: Determination of crAssphage in water samples and applicability for tracking human faecal pollution. In: Microbial Biotechnology. 10, Nr. 6, 2017, S. 1775–1780. doi:10.1111/1751-7915.12841. PMID 28925595. PMC 5658656 (freier Volltext).
- E. Stachler, C. Kelty, M. Sivaganesan, X. Li, E. Bibby, O. C. Shanks: Quantitative CrAssphage PCR Assays for Human Fecal Pollution Measurement. In: Environmental Science and Technology. 51, Nr. 16, 2017, S. 9146–9154. bibcode:2017EnST...51.9146S. doi:10.1021/acs.est.7b02703. PMID 28700235.
Weblinks
- Newly-found gut virus 'abundant in humans' - BBC News
- Ed Yong: Why Has This Really Common Virus Only Just Been Discovered? – National Geographic, 24. Juli 2014
- Michael Price: Novel Virus Discovered in Half the World's Population – SDSU, 24. Juli 2014
- Michaleen Doucleff: Globe-Trotting Virus Hides Inside People's Gut Bacteria – NPR, 24. Juli 2014
- nik/dpa: Neues Darmvirus entdeckt: In jedem zweiten Menschen und doch unbekannt – Spiegel Online, 26. Juli 2014
- Amanda Forde, Colin Hill: [Phages of life – the path to pharma], in: British Journal of Pharmakology, 19. Dezember 2017, doi:10.1111/bph.14106
- Benjamin Siranosian: Transmission of crAssphage in the microbiome; What is crAssphage?, Blogs vom 24. Juli 2019 (Stanford Genetics Ph.D Student)
- Haiyang Chen, Lijun Jing, Zhipeng Yao, Fansheng Meng, Yanguo Teng: Prevalence, source and risk of antibiotic resistance genes in the sediments of Lake Tai (China) deciphered by metagenomic assembly: A comparison with other global lakes, in: Environment International, Band 127, Juni 2019, S. 267–275 Februar/März 2019, doi:0.1016/j.envint.2019.03.048 (ARG: Acronym für antibiotic resistance gene)
- Robert A. Edwards, Alejandro A. Vega, Bas E. Dutilh et al.: Global phylogeography and ancient evolution of the widespread human gut virus crAssphage, in: Nature Microbiology, Band 4, 8. Juli 2019, Last updated: 2. Oktober 2019, S. 1727–1736, doi:10.1038/s41564-019-0494-6
- Kata Farkas, Evelien M. Adriaenssens, David I. Walker, James E. McDonald, Shelagh K. Malham, Davey L. Jones: Critical Evaluation of CrAssphage as a Molecular Marker for Human-Derived Wastewater Contamination in the Aquatic Environment, in: Food and Environmental Virology, Band 11, Nr. 2, S. 113–119, Juni 2019, Online: 13. Februar 2019, doi:10.1007/s12560-019-09369-1
- Arina V. Drobysheva, Sofia A. Panafidina, Matvei V. Kolesnik, Maria L. Sokolova et al.: Structure and function of virion RNA polymerase of a crAss-like phage, in: Nature 589 (2021), S. 306–309, Epub 18. November 2020, doi:10.1038/s41586-020-2921-5. Dazu:
- Researchers recognize a viral protein's M.O. by just 3% of its size, auf: EurekAlert vom 18. November 2020