Ackermannviridae
Ackermannviridae ist die Bezeichnung einer Familie von Viren in der Ordnung Caudovirales. Als natürliche Wirte dienen Gammaproteobakterien (Proteobacteria). Derzeit (Stand 8. Februar 2021) gibt es offiziell vom International Committee on Taxonomy of Viruses (ICTV) bestätigt zwei Unterfamilien, vier Gattungen und 21 Arten in dieser Familie.[3][1]
| Ackermannviridae | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Systematik | ||||||||||||||
| ||||||||||||||
| Taxonomische Merkmale | ||||||||||||||
| ||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||
| Ackermannviridae | ||||||||||||||
| Links | ||||||||||||||
|
Etymologie
Der Name der Familie wurde zu Ehren von Hans-Wolfgang Ackermann (1936–2017), einem deutschen Mikrobiologen und Virologen (geboren in Berlin) vergeben. Die Endung ‚-viridae‘ ist die Standard-Endung für Virusfamilien.[4][5]
Aufbau
Die Virionen (Virusteilchen) der Ackermannviridae haben wie bei allen Caudovirales („Schwanzviren“) eine Kopf-Schwanz-Struktur. Der ikosaedrische Kopf (Kapsid) ist nicht umhüllt und hat einen Durchmesser von ca. 93 nm. Der Hals ist ohne Kragen und hat eine kleine Grundplatte. Der Schwanz von etwa 140 nm Breite und 20 nm Länge ist kontraktil. Am Schwanz sind ca. 38 nm lange Fibern angebracht.[6]
Genom
Das Genom ist ein lineares dsDNA-Molekül (Doppelstrang-DNA) von etwa 155 kbp (Kilo-Basenpaare) Länge. Es kodiert für 190 bis 216 Proteine und eine bis sieben tRNAs (Transfer-RNAs).[6]
Replikationszyklus
Die Replikation erfolgt im Zytoplasma der Wirtszellen. Die Gene werden durch Operons transkribiert.
Zunächst heftet sich der Phage über seine Schwanzfasern an die Wirtszelle. Danach erfolgt der Ausstoß (Injektion) der Virus-DNA in das Zytoplasma der Wirtszelle durch den hohlen Schwanz per Kontraktion dieser Schwanzscheide. Dort folgen die Transkription und Translation der „frühen“ Gene, d. h. er werden zunächst diejenigen Proteine erzeugt, die die Kontrolle über die Wirtszelle übernehmen. In der nächsten Phase folgt die Replikation des viralen Genoms sowie die Transkription und Translation der restlichen, „späten“ Gene.
Jetzt kann der Zusammenbau (Assembly) der Virusteilchen beginnen: An dessen Anfang steht die Montage eines noch leeren „Prokapsids“ (= leeres Kapsid ohne Genom darin). Danach folgt die Verpackung des Genoms. Die Assemblierung wird abgeschlossen mit dem Zusammenbau der viralen Schwanzfasern und der Montage des Virus-Schwanzes.
Die reifen Virionen werden schließlich durch Lyse aus der Zelle unter deren Zerstörung (Platzen) freigesetzt.[6]
Systematik
Die Systematik der Ackermannviren ist nach ICTV (ergänzt um einige Vorschläge nach NCBI in Anführungszeichen) wie folgt:[3][2][7]
Ordnung: Caudovirales, Familie: Ackermannviridae
- Unterfamilie: Aglimvirinae
- Gattung: Agtrevirus (veraltet Ag3virus)
- Spezies: Salmonella-Virus SKML39 (englisch Salmonella virus SKML39)
- Spezies: Shigella-Virus AG3 (en. Shigella virus AG3, Typus, mit Shigella phage phiSboM-AG3)
- Spezies: Enterobacter-Phage EspM4VN (en. Enterobacter phage EspM4VN)
- Spezies: Salmonella-Phage P46FS4 (en. Salmonella phage P46FS4)
- Spezies: Shigella-Phage MK-13 (en. Shigella phage MK-13)
- Gattung: Limestonevirus
- Spezies: Dickeya-Virus Limestone (en. Dickeya virus Limestone, Typus)
- Spezies: Dickeya-Virus RC2014 (en. Dickeya virus RC2014)
- Spezies: Dickeya-Phage Coodle (en. Dickeya phage Coodle)
- Unterfamilie: Cvivirinae
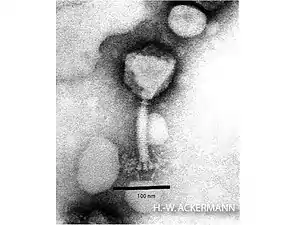
- Gattung: Kuttervirus (veraltet Cba120virus), zusammengelegt mit Viunavirus (veraltet Vi1virus, Viunalikevirus)
- Spezies: Escherichia-Virus CBA120 (en. Escherichia virus CBA120, Typus)
- Spezies: Escherichia-Virus ECML4 (en. Escherichia virus ECML4) – bisher Viunavirus
- Spezies: Escherichia-Virus PhaxI (en. Escherichia virus PhaxI)
- Spezies: Salmonella-Virus 38 (en. Salmonella virus 38)
- Spezies: Salmonella-Virus Det7 (en. Salmonella virus Det7)
- Spezies: Salmonella-Virus GG32 (en. Salmonella virus GG32)
- Spezies: Salmonella-Virus PM10 (en. Salmonella virus PM10)
- Spezies: Salmonella-Virus SFP10 (en. Salmonella virus SFP10)
- Spezies: Salmonella-Virus SH19 (en. Salmonella virus SH19)
- Spezies: Salmonella-Virus SJ3 (en. Salmonella virus SJ3)
- Spezies: Escherichia-Phage EP75 (en. Escherichia phage EP75)
- Spezies: Salmonella-Phage allotria (en. Salmonella phage allotria)
- Spezies: Salmonella-Phage barely (en. Salmonella phage barely)
- Spezies: Salmonella-Phage bering (en. Salmonella phage bering)
- Spezies: Salmonella-Phage dinky (en. Salmonella phage dinky)
- Spezies: Salmonella-Phage heyday (en. Salmonella phage heyday)
- Spezies Salmonella-Virus maane (en. Salmonella virus maane)
- Spezies Salmonella-Virus Marshall (en. Salmonella virus Marshall)
- Spezies Salmonella-Virus Maynard (en. Salmonella virus Maynard)
- Spezies Salmonella-Virus SJ2 (en. Salmonella virus SJ2)
- Spezies Salmonella-Virus STML131 (en. Salmonella virus STML131)
- Spezies Salmonella-Virus ViI (en. Salmonella virus ViI)
- ohne zugewiesene Unterfamilie:
- Gattung: Campanilevirus
- Gattung: Kujavirus
- Gattung: Miltonvirus
- Spezies: Serratia-Virus MAM1 (en. Serratia virus MAM1) mit Bacteriophage ϕMAM1
- Gattung: Nezavisimistyvirus
- Spezies: Erwinia-Virus Ea2809 (en. Erwinia virus Ea2809)
- Gattung: Taipeivirus
- Spezies: Escherichia-Virus KWBSE43-6 (en. Escherichia virus KWBSE43-6)
- Spezies: Klebsiella-Virus 0507KN21 (en. Klebsiella virus 0507KN21, Typus)
- Spezies: Klebsiella-Virus KpS110 (en. Klebsiella virus KpS110)
- Spezies: Klebsiella-Virus Magnus (en. Klebsiella virus Magnus)
- Spezies: Klebsiella-Virus May (en. Klebsiella virus May)
- Spezies: Klebsiella-Virus Menlow (en. Klebsiella virus Menlow)
- Spezies: Serratia-Virus IME250 (en. Serratia virus IME250)
- Gattung: Vapseptimavirus
- Spezies: Vibrio-Virus VAP7 (en. Vibrio virus VAP7)
- ohne zugewiesene Gattung:
- Spezies: Acinetobacter-Phage SH-Ab 15599 (en. Acinetobacter phage SH-Ab 15599)[8][9][10]
- Spezies: Aeromonas-Phage phiA8-29 (en. Aeromonas phage phiA8-29)
- Spezies: Ralstonia-Phage RSP15 (en. Ralstonia phage RSP15)
- Spezies: Rhizobium-Phage AF3 (en. Rhizobium phage AF3)
- Spezies: Serratia-Phage KNP4 (en. Serratia phage KNP4)
- Spezies: Sinorhizobium-Phage phiM9 (en. Sinorhizobium phage phiM9)
- Spezies: Vibrio-Phage VP-1 (en. Vibrio phage VP-1)
- Spezies: Vibrio-Phage YC (en. Vibrio phage YC)
Weblinks
Einzelnachweise
- ICTV: ICTV Master Species List 2019.v1, New MSL including all taxa updates since the 2018b release, March 2020 (MSL #35)
- ICTV: ICTV Master Species List 2020.v1, New MSL including all taxa updates since the 2019 release, March 2021 (MSL #36)
- Virus Taxonomy: 2019 Release. In: talk.ictvonline.org. International Committee on Taxonomy of Viruses. Abgerufen am 3. Mai 2020.
- Jakub Barylski, François Enault, Bas E. Dutilh, Margo B. P. Schuller, Robert A. Edwards, Annika Gillis, Jochen Klumpp, Petar Knezevic, Mart Krupovic, Jens H. Kuhn et al.: Analysis of Spounaviruses as a Case Study for the Overdue Reclassification of Tailed Phages. Systematic Biology, Band 69, Nr. 1, Januar 2020, S. 110–123, doi:10.1093/sysbio/syz036, ePub 25. Mai 2019
- Hans-W. Ackermann: Life in science. Bacteriophage 2(4), 1. Oktober 2012, S. 207, doi:10.4161/bact.23159, PMC 3594207 (freier Volltext), PMID 23533969. – Ein Lebenslauf.
- SIB: ViralZone: Ackermannviridae
- NCBI: Ackermannviridae (family)
- Yunfen Hua, Tingting Luo, Yiqi Yang, Dong Dong, Rui Wang, Yanjun Wang, Mengsha Xu, Xiaokui Guo, Fupin Hu, Ping He: Phage Therapy as a Promising New Treatment for Lung Infection Caused by Carbapenem-Resistant Acinetobacter baumannii in Mice, in: Front. Microbiol., 9. Januar 2018; doi:10.3389/fmicb.2017.02659
- Natalia Bagińska, Anna Pichlak, Andrzej Górski1, Ewa Jończyk-Matysiak: Specific and Selective Bacteriophages in the Fight against Multidrug-resistant Acinetobacter baumannii, in: Virologica Sinica, Band 34, S. 347–357; doi:10.1007/s12250-019-00125-0, Tbl. 1
- NCBI: Acinetobacter phage SH-Ab 15599 (species)