Herelleviridae
Herelleviridae ist die Bezeichnung für eine Familie von Viren mit sog. Kopf-Schwanz-Aufbau (Ordnung Caudovirales), deren natürlichen Wirte Mitglieder des bakteriellen Phylums Firmicutes sind.[3] Die Familie enthält vom International Committee on Taxonomy of Viruses (ICTV) derzeit (Stand Januar 2021) offiziell bestätigt fünf Unterfamilien, 19 Gattungen und 82 Arten (Spezies).[4] Solche Bakterien infizierenden Viren nennt man auch Bakteriophagen.[4][5] Die Familie ging hervor aus der früheren Unterfamilie Spounavirinae der großen Familie Siphoviridae.[6][4][5]
| Herelleviridae | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|

EM-Aufnahme eines Virions aus der | ||||||||||||||
| Systematik | ||||||||||||||
| ||||||||||||||
| Taxonomische Merkmale | ||||||||||||||
| ||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||
| Herelleviridae | ||||||||||||||
| Links | ||||||||||||||
|
Etymologie
Der Name der Familie, Herelle, wurde anlässlich des 100. Jahrestages der Entdeckung der Bakteriophagen zu Ehren ihres Entdeckers Félix d'Hérelle (1873–1994), eines französisch-kanadischen Mikrobiologen vergeben, der Suffix ‚-viridae‘ ist der Standardsuffix für Virenfamilien.[4][5][7]
Beschreibung
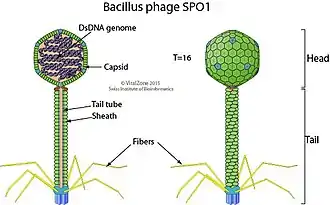
Die Virionen (Virusteilchen) der Herelleviridae haben Kopf-Schwanz-Struktur; der Kopf ist ein ikosaedrisches Kapsid mit einem Durchmesser von 85–100 nm. Die Kapside sind nicht umhüllt und zeigen deutliche die Kapsomerr, d. h. die Kapsiduntereinheiten, diese sind in Fünf- und Sechsecken angeordnet, die sich zum ikosaedrischen Kapsid zusammenfügen. Die Kapside können bis zu 35 Proteine beherbergen. Die unkontrahierten Schwänze sind 130–185 nm lang. Der Hals hat eine Grundplatte von ca. 60 nm und einen kleinen Kragen.[4][8][5]
Das Genome der Herelleviren ist eine lineare doppelsträngige DNA (dsDNA) mit langen terminalen Wiederholungen (englisch repeats) von 3–16 kbp (Kilo-Basenpaaren) Länge. Die Genome sind 125–170 kbp groß und kodieren etwa 165–301 Gene. Mehrere Introns wurden auch in Herellevirus-Genomen nachgewiesen. Die Replikation findet mit Hilfe DNA-Polymerase der Wirte statt.[4][8][5]
Nach den Untersuchungen sind die Phagen dieser Familie obligat lytisch, aber einige können eine anhaltende bzw. pseudolysogene Infektion verursachen.[4][5]
Systematik
Die Herelleviridae bilden nach allgemeiner Auffassung eine monophyletische Klade, was u. a. durch genombasierten Phylogenien gut unterstützt wird. Die Familienmitglieder haben mindestens 60 % Übereinstimmung in der Nukleotidsequenz.[4][5]
Gemäß ICTV mit Stand 11. Juni 2021 gliedert sich die Familie Herelleviridae wie folgt in Unterfamilien und Gattungen (mit einer Auswahl an Spezies)[2]:[4][8][7]
Familie Herelleviridae
- Unterfamilie Bastillevirinae
- Gattung Agatevirus
- Spezies Bacillus-Virus Agate (englisch Bacillus virus Agate, Typus)
- Gattung Bastillevirus
- Spezies Bacillus-Virus Bastille (en. Bacillus virus Bastille, Typus)
- Gattung Bequatrovirus (veraltet: B4virus)
- Spezies Bacillus-Virus B4 (en. Bacillus virus B4, Typus)
- Gattung Caeruleovirus (veraltet: Bc431virus)
- Spezies Bacillus-Virus Bc431 (en. Bacillus virus Bc431, Typus)
- Gattung Eldridgevirus
- Gattung Goettingenvirus
- Gattung Grisebachstrassevirus
- Gattung Jeonjuvirus
- Gattung Matervirus
- Gattung Moonbeamvirus
- Gattung Nitunavirus (veraltet: Nit1virus)
- Spezies Bacillus-Virus NIT1 (en. Bacillus virus NIT1, Typus)
- Gattung Shalavirus
- Gattung Siophivirus
- Gattung Tsarbombavirus
- Spezies Bacillus-Virus TsarBomba (en. Bacillus virus TsarBomba, Typus)
- Gattung Wphvirus
- Spezies Bacillus-Virus WPh (en. Bacillus virus WPh, Typus)
- ohne zugewiesene Gattung:
- Spezies Bacillus-Virus Mater (en. Bacillus virus Mater)
- Spezies Bacillus-Virus Moonbeam (en. Bacillus virus Moonbeamh)
- Spezies Bacillus-Virus SIOphi (en. Bacillus virus SIOphi)
- Unterfamilie Brockvirinae
- Gattung Kochikohdavirus
- Spezies Enterococcus-Virus EF24C (en. Enterococcus virus EF24C, Typus, mit Enterococcus-Phage φEF24C)
- Gattung Schiekvirus
- Spezies Enterococcus-Virus EFDG1 (en. Enterococcus virus EFDG1, Typus)
- UnterfamilieJasinkavirinae
- Gattung Pecentumvirus (veraltet: P100virus)
- Unterfamilie Spounavirinae (ehemals zu Myoviridae)[12]
- Gattung Okubovirus (veraltet: Spo1virus, Spo1likevirus, SPO1-ähnliche Viren)
- Spezies Bacillus-Virus Camphawk (en. Bacillus virus Camphawk)
- Spezies Bacillus-Virus SPO1 (en. Bacillus virus SPO1, Typus, mit Bacillus-Phage SPO1)
- Gattung Siminovitchvirus (veraltet: Cp51virus)
- Spezies Bacillus-Virus CP51 (en. Bacillus virus CP51, Typus)
- Spezies Bacillus-Virus JL (en. Bacillus virus JL)
- Spezies Bacillus-Virus Shanette (en. Bacillus virus Shanette)
- Unterfamilie Twortvirinae
- Gattung Baoshanvirus
- Spezies Staphylococcus-Virus BS1 (en. Staphylococcus virus BS1)
- Spezies Staphylococcus-Virus BS2 (en. Staphylococcus virus BS2, Typus)
- Gattung Kayvirus
- Spezies Staphylococcus-Virus K (en. Staphylococcus virus K, Typus, mit Staphylococcus-Phage K)
- Gattung Sciuriunavirus
- Spezies Staphylococcus-Virus SscM1 (en. Staphylococcus virus SscM1, Typus)
- Gattung Sepunavirus (veraltet: Sep1virus)
- Spezies Staphylococcus-Virus IPLAC1C (en. Staphylococcus virus IPLAC1C, Typus)
- Spezies Staphylococcus-Virus SEP1 (en. Staphylococcus virus SEP1, Typus)
- Gattung Silviavirus
- Spezies Staphylococcus-Virus Remus (en. Staphylococcus virus Remus, Typus)
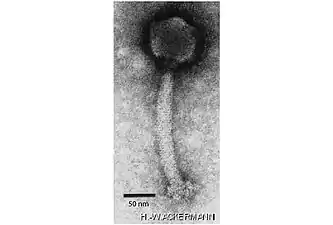
der Gattung Twortvirus
- Gattung Twortvirus (veraltet: Twortlikevirus)
- Spezies Staphylococcus-Virus Twort (en. Staphylococcus virus Twort, Typus, mit Staphylococcus-Phage Twort)[13]
- ohne zugewiesene Unterfamilie
- Gattung Elpedvirus
- Gattung Harbinvirus
- Spezies Lactobacillus-Virus Lpa804 (en. Lactobacillus virus Lpa804, Typus)
- Gattung Hopescreekvirus
- Gattung Mooreparkvirus
- Gattung Salchichonvirus
- Gattung Tybeckvirus
- Gattung Watanabevirus
- ohne Zugewiesene Gattung[12]
- Spezies Brochothrix-Virus A9 (en. Brochothrix virus A9, Zuordnung zu Spounavirinae infrage gestellt)[14][15], infiziert Bakterien der Gattung Brochothrix, Familie Listeriaceae. Nicht zu verwechseln mit dem Riesenphagen Huge Phage A9.[16]
- Spezies Lactobacillus-Virus Lb338-1 (en. Lactobacillus virus Lb338-1)
- Spezies Lactobacillus-Virus LP65 (en. Lactobacillus virus LP65)
Weitere Vorschläge findet man bei NCBI[17]
Einzelnachweise
- ICTV: ICTV Master Species List 2019.v1, New MSL including all taxa updates since the 2018b release, March 2020 (MSL #35)
- ICTV: ICTV Master Species List 2020.v1, New MSL including all taxa updates since the 2019 release, March 2021 (MSL #36)
- Jakub Barylski, Andrew M. Kropinski, Nabil-Fareed Alikhan, Evelien M. Adriaenssens, ICTV Report Consortium: ICTV Virus Taxonomy Profile: Herelleviridae. In: The Journal of General Virology. 101, Nr. 4, April 2020, S. 362–363. doi:10.1099/jgv.0.001392. PMID 32022658.
- Jakub Barylski, Andrew M. Kropinski, Nabil-Fareed Alikhan, Evelien M. Adriaenssens: ICTV Report Herelleviridae. 2020. Via ictvonline.
- Jakub Barylski, François Enault, Bas E. Dutilh, Margo B. P. Schuller, Robert A. Edwards, Annika Gillis, Jochen Klumpp, Petar Knezevic, Mart Krupovic, Jens H. Kuhn, Rob Lavigne, Hanna Maarit Oksanen, Matthew B. Sullivan, Ho Bin Jang, Peter Simmonds, Pakorn Aiewsakun, Johannes Wittmann, Igor Tolstoy, J. Rodney Brister, Andrew M. Kropinski (2020). Taxonomy proposal: To create one (1) new family, Herelleviridae, in the order Caudovirales. University of Helsinki.
- Dann Turner, Andrew M. Kropinski, Evelien M. Adriaenssens: A Roadmap for Genome-Based Phage Taxonomy, in: MDPI Viruses Band 13, Nr. 3, Section Bacterial Viruses, 18. März 2021, 506, doi:10.3390/v13030506
- Jakub Barylski, François Enault, Bas E. Dutilh, Margo B. P. Schuller, Robert A. Edwards, Annika Gillis, Jochen Klumpp, Petar Knezevic, Mart Krupovic, Jens H. Kuhn et al.: Analysis of Spounaviruses as a Case Study for the Overdue Reclassification of Tailed Phages. Systematic Biology, Band 69, Nr. 1, Januar 2020, S. 110–123, doi:10.1093/sysbio/syz036, ePub 25. Mai 2019
- ICTV: ICTV Taxonomy history: Listeria virus A511
- NCBI: Listeria virus A511 (species)
- Matthew Dunne, Mario Hupfeld, Jochen Klumpp, Martin J. Loessner: Molecular Basis of Bacterial Host Interactions by Gram-Positive Targeting Bacteriophages, in: MDPI Viruses, Band 10, Nr. 8, Special Issue Phage-Host Interactions, 397, 28. Juli 2018, doi:10.3390/v10080397.
- Jakub Barylski, François Enault, Bas E. Dutilh, Margo B. P. Schuller, Robert A. Edwards, Annika Gillis, Jochen Klumpp, Petar Knezevic, Mart Krupovic, Jens H. Kuhn et al.: Analysis of Spounaviruses as a Case Study for the Overdue Reclassification of Tailed Phages, in: Systematic Biology, Band 69, Nr. 1, Januar 2020, S. 110–123, Epub 25. Mai 2019, doi:10.1093/sysbio/syz036
- Roman Pantůček, J. Doskar, V. Růzicková, P. Kaspárek et al.: Identification of bacteriophage types and their carriage in Staphylococcus aureus, in: Archives of Virology 149(9), S. 1689–1703, Oktober 2004, doi:10.1007/s00705-004-0335-6, PMID 15593413
- NCBI: Brochothrix virus A9 (species) - keine Unterfamilie zugeordnet
- Samuel Kilcher, Martin J. Loessner, Jochen Klumpp: Brochothrix thermosphacta bacteriophages feature heterogeneous and highly mosaic genomes and utilize unique prophage insertion sites, in: J Bacteriol 192(20), Oktober 2010, S. 5441–5453, Epub 13. August 2010, doi:10.1128/JB.00709-10, PMID 20709901, PMC 2950505 (freier Volltext) – veraltet: Myoviridae: Spounavirinae
- Basem Al-Shayeb, Rohan Sachdeva, Lin-Xing Chen, Cindy J. Castelle, Alexander L. Jaffe, Jennifer A. Doudna, Jillian F. Banfield et al.: Clades of huge phage from across Earth’s ecosystems, in: Nature 578, 12. Februar 2020, S. 425–431, doi:10.1038/s41586-020-2007-4. Dazu:
- bioRxiv, PrePrint vom 11. März 2019, doi:10.1101/572362
- Huge bacteria-eating viruses close gap between life and non-life – Large bacteriophages carry bacterial genes, including CRISPR and ribosomal proteins; auf: EurekAlert! vom 12. Februar 2020
- NCBI: Herelleviridae (family)