Cafeteria-roenbergensis-Virus
Das Cafeteria-roenbergensis-Virus (offiziell Cafeteria roenbergensis virus, CroV) ist eine Virusspezies mit den Stämmen bzw. Isolaten (englisch strains, isolates) Bodo virus-pier water 1 (CroV BV-PW1) und Cafeteria roenbergensis virus MGF-2008 (CroV MGF-2008).[3][4]
| Cafeteria-roenbergensis-Virus | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|

Schemazeichnung eines Cafeteriavirus-Virions (Querschnitt und Seitenansicht)[Anm. 1] | ||||||||||||||||||||
| Systematik | ||||||||||||||||||||
| ||||||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||||||
| ||||||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||||||
| Cafeteria roenbergensis virus | ||||||||||||||||||||
| Kurzbezeichnung | ||||||||||||||||||||
| CroV | ||||||||||||||||||||
| Links | ||||||||||||||||||||
|
Morphologie
Die Cafeteriavirien sind im Meer vorkommende Viren, deren Virusteilchen (Virionen) einen Durchmesser von 280 nm haben. Diese Viren infizieren den im Zooplankton lebenden einzelligen Flagellaten der Art Cafeteria roenbergensis aus der Gattung Cafeteria.[4][5]
CroV gehört zu den größten insgesamt bisher identifizierten Viren. Es wurde bereits in den frühen 1990er Jahren vor der Küste von Texas (USA) entdeckt,[6] ist möglicherweise entfernt mit dem Mimivirus verwandt und wird dem Phylum Nucleocytoviricota (früher Nucleocytoplasmic large DNA viruses (NCLDV)) zugerechnet.
Viele Bakteriophagen haben spezifische Portalstrukturen für die DNA-Verpackung oder die Genomübertragung während der Infektion der Wirtszelle, siehe Caudovirales. Ein sogenanntes „Star-Gate“-Portal (auch Stargate geschrieben) wurde auf dem Kapsid des Mimivirus APMV nachgewiesen. Mit der gleichen Technik wurde festgestellt, dass das Kapsid des Chlorovirus PBCV-1 einen modifizierten Scheitelpunkt (Vertex) mit einer Tasche unterhalb einer nadelartigen Spike-Struktur aufweist, durch den das Genom in die Wirtszelle eingebracht wird. Bei CroV gibt es möglicherweise eine ausgezeichnete Achse mit zwei gegenüberliegenden Polen (englisch two opposing vertices) im ansonsten ikosaedrisch aufgebauten Kapsid, die ein solches (größenmäßig kleines) Portal anzeigen könnten. Ein eindeutiger Nachweis war Xiao et al. (21017) jedoch nicht gelungen, genauere Untersuchungen sind daher noch nötig.[7]
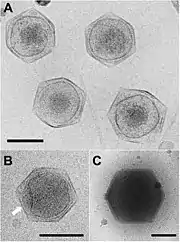
.jpg.webp)
_virions.jpg.webp) Querschnitt der Kryo-EM-Darstellung von CroV, überlagert mit der des Chlorovirus PBCV-1. CroV hat nicht nur einen größeren Durchmesser der Virionen (300 nm vs. 180 nm), seine Kapsidschicht ist auch dicker (10,5 nm vs. 7,5 nm). Umrechnung 10 Å = 1 nm[7][Anm. 2]
Querschnitt der Kryo-EM-Darstellung von CroV, überlagert mit der des Chlorovirus PBCV-1. CroV hat nicht nur einen größeren Durchmesser der Virionen (300 nm vs. 180 nm), seine Kapsidschicht ist auch dicker (10,5 nm vs. 7,5 nm). Umrechnung 10 Å = 1 nm[7][Anm. 2]
Genomgröße
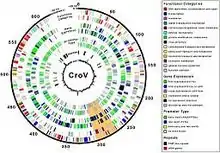
Mit einer Genomlänge von bis zu etwa 730 kbp (DNA-Nukleinbasenpaaren) und über 500 erkannten Genen ist das Erbgut dieses großen Meeresvirus im Gegensatz zu vielen einfach aufgebauten Viren sehr umfangreich und übertrifft sogar das Erbgut einiger komplexer Einzeller. Cafeteria roenbergensis virus BV PW1 hat beispielsweise eine Genomlänge von 617.453 bp bei vorausgesagt 544 kodierten Proteinen.[8] Der GC-Gehalt liegt bei 23 %, vergleichbar mit Hokovirus (21,4 %) und Choanovirus 1 (22 %), zwei anderen (aus Metagenomanalysen) vorgeschlagenen Riesenviren.[9][10]
Wie andere Viren auch, muss das Cafeteria-roenbergensis-Virus für seine Replikation in eine Wirtszelle eindringen, jedoch kann es auf Grund seines umfangreichen Erbguts wichtige Zellbestandteile selbständig herstellen. So exprimieren bestimmte Gene unter anderem DNA-Reparatur-Enzyme, die bei anderen Viren bisher nicht gefunden wurden. Ein anderer etwa 38.000 Basenpaare großer Erbgut-Abschnitt ist wahrscheinlich bakterieller Herkunft und codiert Enzyme, die zur Synthese von Kohlenhydraten notwendig sind. Genau derartige Kohlenhydrate bilden auch die äußere Zellmembran mancher Bakterienstämme.[5] CroV kodiert für acht Untereinheiten der DNA-abhängigen RNA-Polymerase und außerdem für mindestens sechs Transkriptionsfaktoren, wodurch das DNA-Genom ohne die Verwendung von Proteinen der Zelle in mRNA umgeschrieben werden kann. CroV kann dann die mRNAs mit Hilfe der Translationsmaschine der Zelle in Proteine übersetzen, indem es seine eigene tRNA-Synthetase, tRNA und Translationsinitiationsfaktoren verwendet, um die Translation zu seinem eigenen Vorteil zu optimieren.[5]
Das CroV-Genom wird nicht in das Genom der Wirtszelle integriert.[5]
Da man – nach Angabe der Forscher – ein Großteil der in diesem Virus gefundenen genetischen Ausrüstung nur in einer lebenden Zelle erwarten würde, verwischt das Cafeteria-roenbergensis-Virus die Grenze zwischen Viren/Virionen und lebenden Organismen und stellt damit die unter Wissenschaftlern weit verbreitete Einschätzung der Viren als Nichtlebewesen in Frage.[5]
Replikation und Virophage
CroV dringt über Phagozytose in seine Wirtszellen ein. Sobald es in der Zelle ist, zerlegt sich das CroV-Kapsid, wodurch die viralen Proteine und das Genom freigesetzt werden. CroV verbleibt im Zytoplasma, wo es ähnlich wie andere Mimiviridae eine sog. Virusfabrik bildet. Dies sind großen Konstrukte, in denen die Replikation unabhängig vom Wirtszellkern stattfindet. CroV macht keinen Gebrauch von der Transkriptions- oder Translationsmaschinerie der Wirtszelle.[5]
Die Replikation von CroV findet wie bei anderen Mimiviridae in großen Konstrukten statt, die man Virusfabriken nennt. Es wird angenommen, dass hier die DNA-Replikation, die Transkription und der Partikelzusammenbau (Assemblierung) stattfinden.
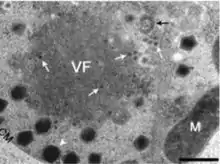 CroV repliziert in einer Virusfabrik (VF). Die weiße Pfeilspitze zeigt neu gebildete CroV-Partikel an. Es liegt eine Koinfektion mit dem Virophagen Mavirus vor, die weißen langstieligen Pfeile zeigen dessen kleine Partikel an.
CroV repliziert in einer Virusfabrik (VF). Die weiße Pfeilspitze zeigt neu gebildete CroV-Partikel an. Es liegt eine Koinfektion mit dem Virophagen Mavirus vor, die weißen langstieligen Pfeile zeigen dessen kleine Partikel an.
CroV ist Wirt des Virophagen Mavirus: Die Virusfabriken sind auch die primären Ziele des Virophagen Mavirus, der die CroV-Maschinerie zur Replikation nutzt.Mavirus ist ein dsDNA-Virus mit unsegmentiertem! zirkulärem Genom mit einer Größe von 19.000 kb. Eine Koinfektion mit Mavirus reduziert die Sterblichkeit der Wirtszellen, indem es die CroV-Infektion und -Replikation stört.[12] Mavirus kann sich in das Genom der Zellen von Cafeteria roenbergensis integrieren und verleiht der Population dadurch Immunität.[13]
Bedeutung für das Ökosystem Ozean
Da das Cafeteria-roenbergensis-Virus eine im Meer weit verbreitete Plankton-Art (Cafeteria roenbergensis) befällt, die sich ihrerseits von Bakterien ernährt und damit die Basis der marinen Nahrungskette darstellt, hat es wahrscheinlich einen großen Einfluss auf das Ökosystem der Ozeane,[5] da ein Virenbefall beispielsweise auch einen Zusammenbruch von Cafeteria-Populationen bewirken kann.[4]
Merkmale der Mimiviridae im Vergleich
| Virus | Aminoacyl-tRNA-Synthetase | Octocorallia-ähnliche MutS | Proteinfilamente (Länge) | Stargate[14] | Bekannter Virophage[15] | Cytoplasmische Virion-Fabrik |
Wirt |
|---|---|---|---|---|---|---|---|
| Megavirus chilensis | 7 (Tyr, Arg, Met, Cys, Trp, Asn, Ile) | ja | ja (75 nm) | ja | nein | ja | Acanthamöben (Unikonta, Amoebozoa) |
| Mamavirus ACMV | 4 (Tyr, Arg, Met, Cys) | ja | ja (120 nm) | ja | ja | ja | Acanthamöben (Unikonta, Amoebozoa) |
| Mimivirus ApMV (Wildtyp M1) | 4 (Tyr, Arg, Met, Cys) | ja | ja (120 nm) | ja | ja | ja | Acanthamöben (Unikonta, Amoebozoa) |
| Mimivirus M4 (bald/fiberless Variante) | 2 (Met, Cys) | nein | nein | ja | resistent | ja | Acanthamöben (Unikonta, Amoebozoa) |
| Cafeteria-roenbergensis-Virus | 1 (Ile) | ja | nein | nein | ja | ja | Phagotrophische Protozoen (Heterokonta, Stramenopiles) |
Systematik
Ein naher Verwandter des CroV scheint die aus einer Metagenomanalyse von Waldbodenproben identifizierte Kandidatenspezies Faunusvirus sp.[16] (nicht zu verwechseln mit der offiziellen Gattung Faunusvirus[17] der Chaseviridae) zu sein.[2] Die meisten Autoren sehen die Cafeteriaviren als basale (mit Stand 2018 noch unbenannte) Unterfamilie innerhalb der Klade der ‚herkömmlichen‘ Mimiviridae (die Mimiviridae ohne die neuerdings als Unterfamilie „Mesomimiviridae“ vorgeschlagene und früher OLPG genannte Gruppe).[2] Eine alternative Sicht (CNRS 2018) rückt die Cafeteriaviren in die Nähe der Klosneuviren und schlägt daher eine gemeinsame Unterfamilie Aquavirinae vor.[18]
Literatur
- Tanya Marie St. John: Characterization of a Large DNA Virus (BV-PW1) infecting the heterotropic marine nanoflagellate Cafeteria sp. wissenschaftliche Arbeit zur Erlangung des Master of Science (M.Sc.) an der University of British Columbia, 2003; Volltext (PDF)
- Philippe Colson et al.: The Giant Cafeteria roenbergensis Virus That Infects a Widespread Marine Phagocytic Protist Is a New Member of the Fourth Domain of Life. In: PLoS One. 2011, Band 6, Nummer 4, Artikel e18935; veröffentlicht online: 29. April 2011, doi:10.1371/journal.pone.0018935.
Weblinks
- Cafeteria roenbergensis virus BV-PW1. National Center for Biotechnology Information (NCBI); abgerufen am 12. Februar 2016.
- Cafeteria roenbergensis virus BV-PW1, complete genome. (vollständiges Genom des Cafeteria-roenbergensis-Virus in graphischer Darstellung) National Center for Biotechnology Information (NCBI); abgerufen am 12. Februar 2016.
- Graziele Oliveira, Bernard La Scola, Jônatas Abrahão: Giant virus vs amoeba: fight for supremacy. In: Virology Journal, Band 16, Nr. 1, Artikel Nummer: 126, veröffentlicht: 4. November 2019, doi:10.1186/s12985-019-1244-3; researchgate.net (PDF).
Anmerkungen
- Die hier eingezeichnete Sternenstruktur gibt es in dieser Form vermutlich nicht. Siehe §Morphologie
- Das Material wurde von dieser Quelle kopiert, die unter einer Creative Commons Attribution 4.0 International License verfügbar ist.
Einzelnachweise
- ICTV: ICTV Master Species List 2019.v1, New MSL including all taxa updates since the 2018b release, March 2020 (MSL #35)
- Frederik Schulz, Lauren Alteio, Danielle Goudeau et al.: Hidden diversity of soil giant viruses. In: Nature Communicationsvolume. Band 9, 2018, Artikelnummer: 4881 vom 19. November 2018, doi:10.1038/s41467-018-07335-2.
- Julien Andreani, Jacques Y. B. Khalil, Emeline Baptiste et al.: Orpheovirus IHUMI-LCC2: A New Virus among the Giant Viruses. In: Frontiers in Microbiology. 22. Januar 2018, doi:10.3389/fmicb.2017.02643.
- Ramon Massana, Javier del Campo, Christian Dinter, Ruben Sommaruga: Crash of a population of the marine heterotrophic flagellate Cafeteria roenbergensis by viral infection. In: Environmental Microbiology. Band 9, 2007, S. 2660–2669, doi:10.1111/j.1462-2920.2007.01378.x,
- Matthias G. Fischer, Michael J. Allen, William H. Wilson, Curtis A. Suttle: Giant virus with a remarkable complement of genes infects marine zooplankton. In: Proceedings of the National Academy of Sciences. 2010. doi:10.1073/pnas.1007615107.
- D. Randy Garza, Curtis A. Suttle: Large double-stranded DNA viruses which cause the lysis of a marine heterotrophic nanoflagellate (Bodo sp.) occur in natural marine viral communities. In: Aquatic Microbial Ecology. Band 9, Nr. 3, 21. Dezember 1995; Volltext (PDF; 1,6 MB).
- Chuan Xiao, Matthias G. Fischer, Duer M. Bolotaulo, Nancy Ulloa-Rondeau, Gustavo A. Avila, Curtis A. Suttle: Cryo-EM reconstruction of the Cafeteria roenbergensis virus capsid suggests novel assembly pathway for giant viruses. In: Nature Scientific Reports. Band 7, Nr. 5484, 14. Juli 2017, doi:10.1038/s41598-017-05824-w.
- Disa Bäckström, Natalya Yutin, Steffen L. Jørgensen et al.; Richard P. Novick (Hrsg.): Virus Genomes from Deep Sea Sediments Expand the Ocean Megavirome and Support Independent Origins of Viral Gigantism. In: mBio. Band 10, Nr. 2, März–April 2019, S. e02497-18, PDF (PDF) doi:10.1128/mBio.02497-18, PMC 6401483 (freier Volltext), PMID 30837339, ResearchGate
- David M. Needham, Susumu Yoshizawa, Toshiaki Hosakaet et al.: A distinct lineage of giant viruses brings a rhodopsin photosystem to unicellular marine predators. In: PNAS. 23. September 2019, doi:10.1073/pnas.1907517116, ISSN 0027-8424, hier: Supplement 1 (xlsx)
- Frederik Schulz, Natalya Yutin, Natalia N. Ivanova, Davi R. Ortega, Tae Kwon Lee, Julia Vierheilig, Holger Daims, Matthias Horn, Michael Wagner: Giant viruses with an expanded complement of translation system components. In: Science. 356, Nr. 6333, 7. April 2017, ISSN 0036-8075, S. 82–85. doi:10.1126/science.aal4657.
- S. Duponchel, M. G. Fischer: Viva lavidaviruses! Five features of virophages that parasitize giant DNA viruses. In: PLoS pathogens. Band 15, Nr. 3, doi:10.1371/journal.ppat.1007592.
- Matthias Fischer, Curtis Suttle: A Virophage at the Origin of Large DNA Transposons. In: Science. 332, Nr. 6026, April 2011, S. 231–234. bibcode:2011Sci...332..231F. doi:10.1126/science.1199412. PMID 21385722.
- M. G. Fischer, Thomas Hackl: Host genome integration and giant virus-induced reactivation of the virophage mavirus. In: Nature. Band 540, Nr. 7632, Dezember 2016, S. 288–291. bibcode:2016Natur.540..288F. doi:10.1038/nature20593. PMID 27929021.
- Nathan Zauberman, Y. Mutsafi, D. B. Halevy, E. Shimoni, E. Klein, C. Xiao, S. Sun, A. Minsky: Distinct DNA Exit and Packaging Portals in the Virus Acanthamoeba polyphaga mimivirus. In: PLoS Biology. Band 6, Nr. 5, 2008, S. e114, doi:10.1371/journal.pbio.0060114, PMID 18479185, PMC 2430901 (freier Volltext).
- M. G. Fischer, C. A. Suttle: A Virophage at the Origin of Large DNA Transposons. In: Science. Band 332, Nr. 6026, 2011, S. 231–234, doi:10.1126/science.1199412, PMID 21385722.
- NCBI: Faunusvirus sp. (species)
- NCBI: Faunusvirus (genus)
- Centre national de la recherche scientifique: List of the main “giant” viruses known as of today. (PDF; 334 kB) Université Aix Marseille, 18. April 2018.